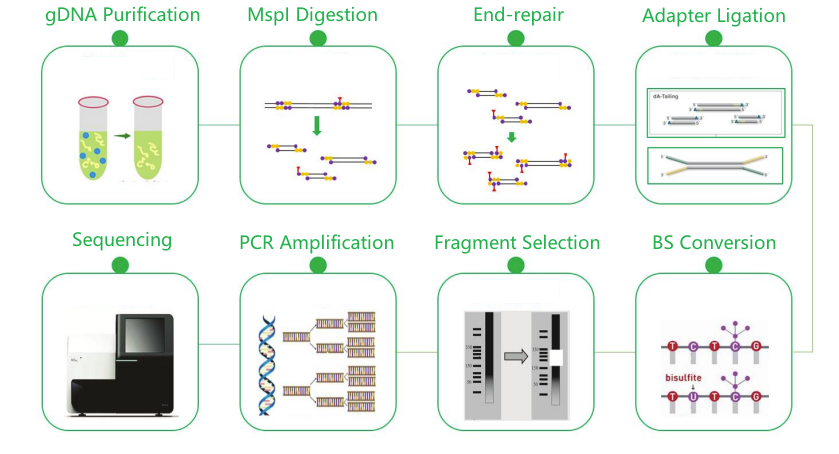
Бісульфітне секвенування зі зниженим представленням (RRBS)
Особливості сервісу
● Потрібен еталонний геном.
● Лямбда ДНК використовується для моніторингу ефективності перетворення бісульфіту.
● Ефективність травлення MspI також контролюється.
● Подвійне ферментне розщеплення для зразків рослин.
● Секвенування на Illumina NovaSeq.
Переваги сервісу
●Економічна та ефективна альтернатива WGBS: можливість проведення аналізу з нижчими витратами та меншими вимогами до зразків.
●Повна платформа:надати комплексні відмінні послуги від обробки зразків, створення бібліотеки та секвенування до біоінформаційного аналізу.
●Великий досвід: завдяки успішно завершеним проектам секвенування RRBS для різноманітних видів, BMKGENE має понад десятирічний досвід, висококваліфіковану аналітичну команду, повний контент і чудову підтримку після продажу.
Специфікації послуги
| Бібліотека | Стратегія секвенування | Рекомендований вихід даних | Контроль якості |
| Бібліотека, розщеплена MspI та оброблена бісульфітом | Illumina PE150 | 8 Гб | Q30 ≥ 85% Перетворення бісульфіту > 99% Ефективність різання MspI > 95% |
Зразок вимог
| Концентрація (нг/мкл) | Загальна кількість (мкг) |
| |
| Геномна ДНК | ≥ 30 | ≥ 1 | Обмежений розпад або забруднення |
Потік роботи служби

Доставка зразків

Будівництво бібліотеки

Секвенування

Аналіз даних
Післяпродажне обслуговування
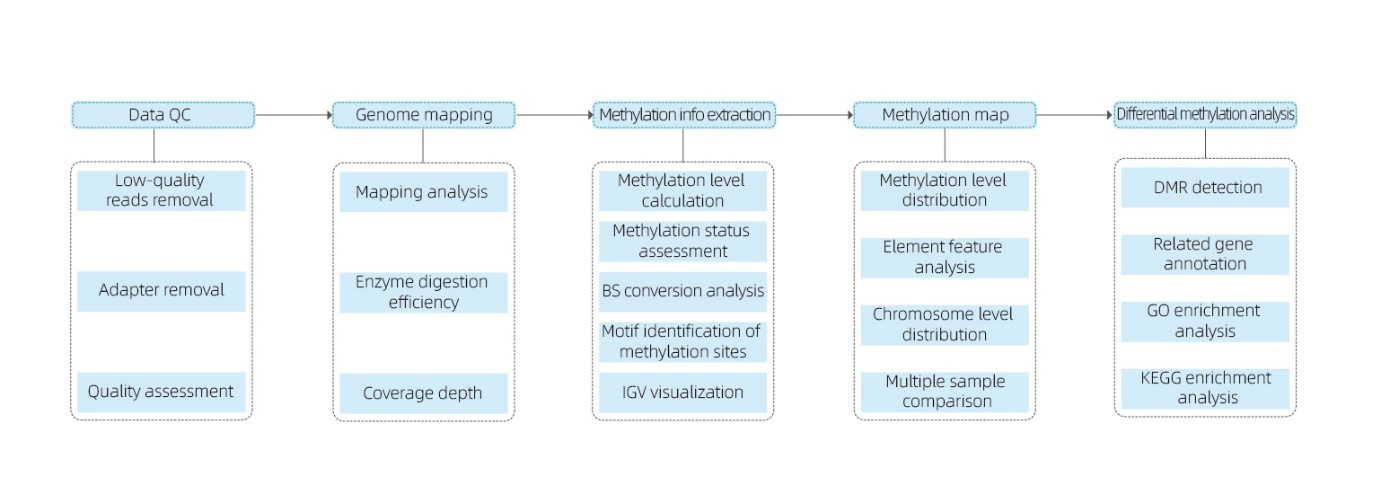
Включає наступний аналіз:
● Контроль якості необробленого секвенування;
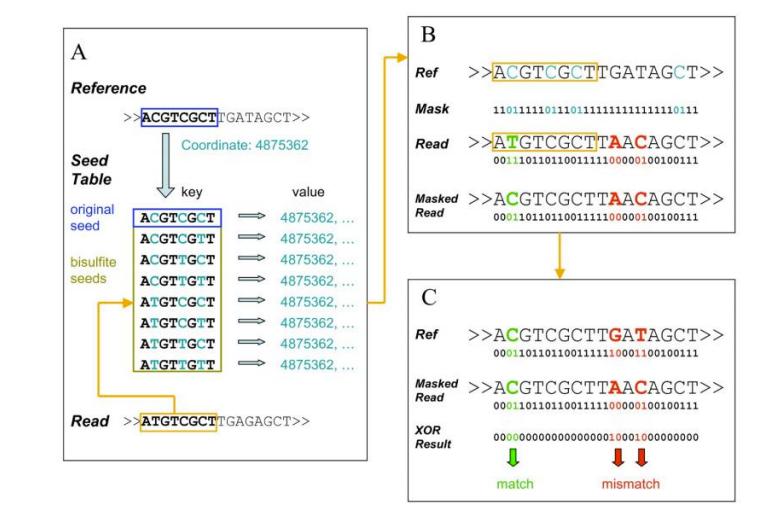
● Картування з еталонним геномом;
● Виявлення 5mC метильованих основ та ідентифікація мотиву;
● Аналіз розподілу метилювання та порівняння зразків;
● Аналіз диференціально метильованих областей (DMR);
● Функціональна анотація генів, асоційованих з DMR.
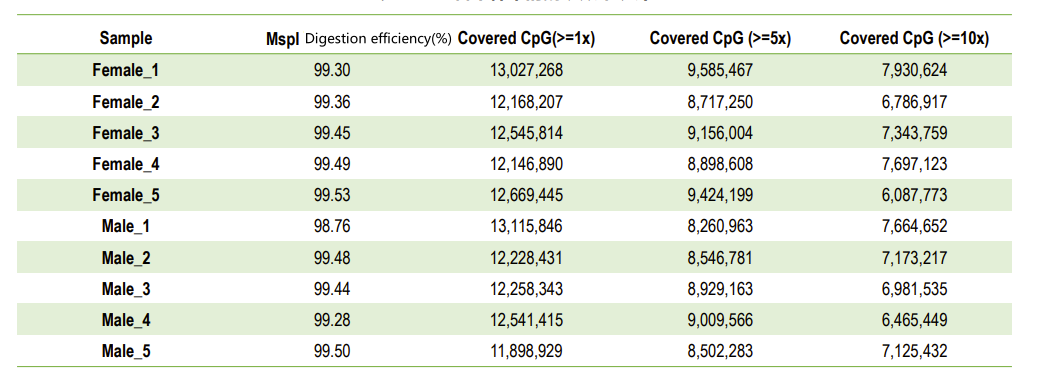
Контроль якості: ефективність травлення (у картуванні геному)
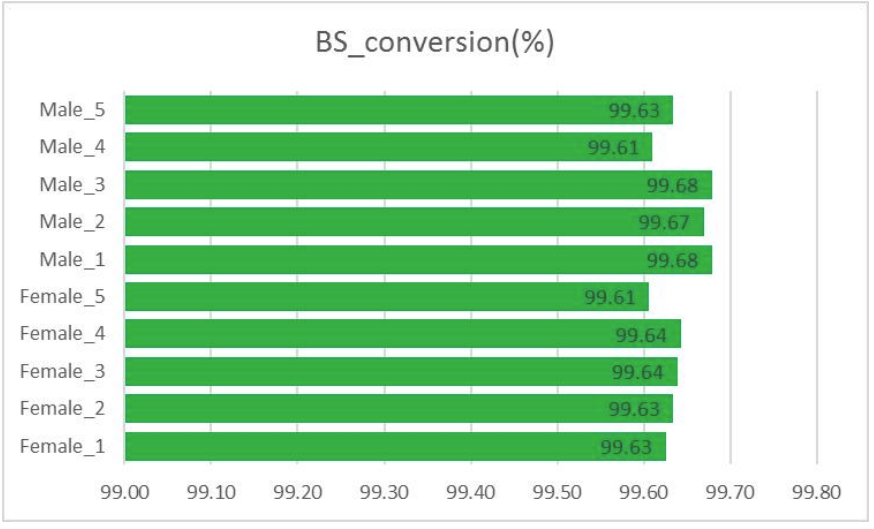
Контроль якості: конверсія бісульфіту (при вилученні інформації про метилювання)
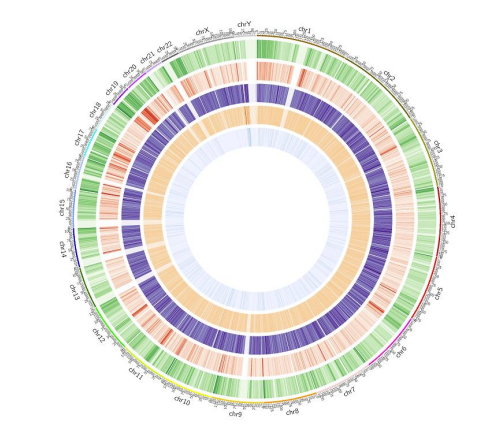
Карта метилювання: поширення метилювання по всьому геному 5 мС
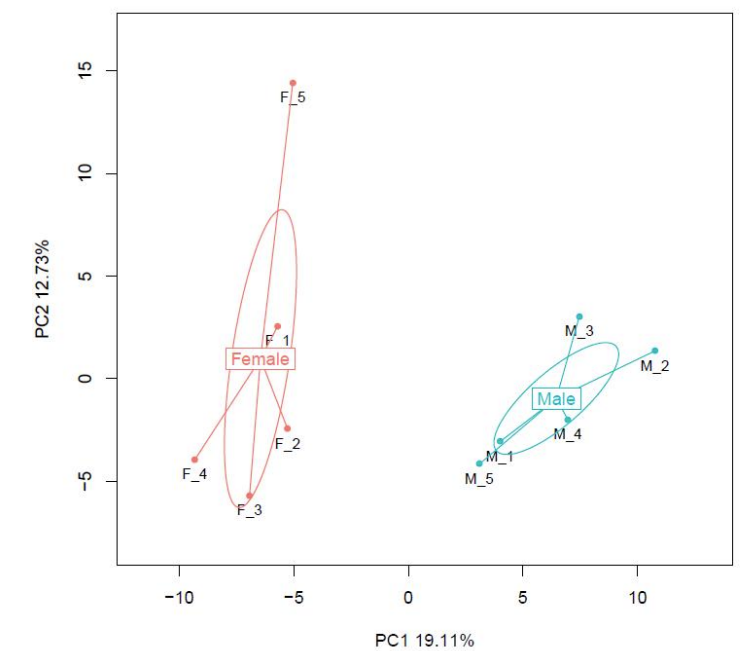
Приклад порівняння: аналіз головних компонентів
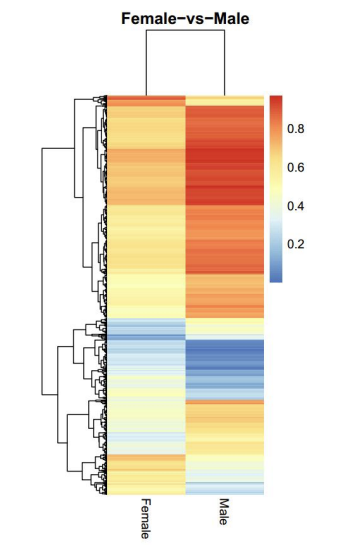
Аналіз диференціально метильованих областей (DMR): теплова карта
Ознайомтеся з науково-дослідницькими досягненнями завдяки послугам BMKGene з бісульфітного секвенування всього генома за допомогою підібраної колекції публікацій.
Li, Z. та ін. (2022) «Високоточне перепрограмування в клітини, подібні до Лейдіга, за допомогою активації CRISPR і паракринних факторів»,PNAS Nexus, 1(4). doi: 10.1093/PNASNEXUS/PGAC179.
Tian, H. та ін. (2023) «Погеномний аналіз метилювання ДНК складу тіла у китайських монозиготних близнюків»,Європейський журнал клінічних досліджень, 53(11), с. e14055. doi: 10.1111/ECI.14055.
Wu, Y. та ін. (2022) «Метилювання ДНК і співвідношення об’єму талії до стегон: загальноепігеномне дослідження асоціацій у китайських монозиготних близнюків»,Журнал ендокринологічних досліджень, 45(12), стор. 2365–2376. doi: 10.1007/S40618-022-01878-4.