-

Метагеномне секвенування -NGS
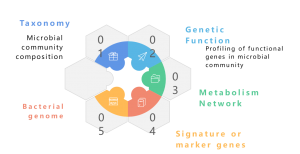
Метагеном — це сукупність загального генетичного матеріалу змішаної спільноти організмів, наприклад, метагеноми середовища та людини. Він містить геноми як культивованих, так і некультивованих мікроорганізмів. Метагеномне секвенування Shotgun за допомогою NGS дозволяє вивчати ці складні геномні ландшафти, вбудовані в зразки навколишнього середовища, забезпечуючи більше, ніж таксономічне профілювання, даючи також детальне уявлення про різноманіття видів, динаміку чисельності та складні популяційні структури. Крім таксономічних досліджень, метагеноміка дробовика також пропонує перспективу функціональної геноміки, дозволяючи досліджувати закодовані гени та їх передбачувану роль в екологічних процесах. Нарешті, встановлення кореляційних мереж між генетичними елементами та факторами середовища сприяє цілісному розумінню складної взаємодії між мікробними спільнотами та їх екологічним фоном. Підсумовуючи, метагеномне секвенування є ключовим інструментом для розкриття геномних тонкощів різноманітних мікробних спільнот, висвітлюючи багатогранні зв’язки між генетикою та екологією в цих складних екосистемах.
Платформи: Illumina NovaSeq і DNBSEQ-T7
-

Метагеномне секвенування-TGS
Метагеном — це сукупність генетичного матеріалу змішаної спільноти організмів, наприклад метагеноми середовища та людини. Він містить геноми як культивованих, так і некультивованих мікроорганізмів. Метагеномне секвенування дозволяє вивчати ці складні геномні ландшафти, вбудовані в екологічні зразки, забезпечуючи більше, ніж таксономічне профілювання. Він також пропонує перспективу функціональної геноміки, досліджуючи закодовані гени та їх передбачувану роль у процесах навколишнього середовища. У той час як традиційні підходи із секвенуванням Illumina широко використовувалися в метагеномних дослідженнях, поява секвенування Nanopore та PacBio змінило сферу. Технології Nanopore та PacBio покращують подальший біоінформаційний аналіз, зокрема збірку метагеномів, забезпечуючи більш безперервну збірку. Звіти показують, що метагеноміка на основі Nanopore та PacBio успішно створила повні та закриті бактеріальні геноми зі складних мікробіомів (Moss, EL та ін., Nature Biotech, 2020). Інтеграція зчитування Nanopore із зчитуванням Illumina забезпечує стратегічний підхід до виправлення помилок, пом’якшуючи притаманну Nanopore низьку точність. Ця синергетична комбінація використовує сильні сторони кожної платформи секвенування, пропонуючи надійне рішення для подолання потенційних обмежень і підвищення точності та надійності метагеномних аналізів.
Платформа: Nanopore PromethION 48, Illumia та PacBio Revio
-

Бісульфітне секвенування повного генома (WGBS)
Бісульфітне секвенування повного геному (WGBS) є золотим стандартом методології для поглибленого дослідження метилювання ДНК, зокрема п’ятої позиції в цитозині (5-mC), ключовому регуляторі експресії генів і клітинної активності. Принцип, що лежить в основі WGBS, передбачає обробку бісульфітом, індукуючи перетворення неметильованих цитозинів в урацил (C в U), залишаючи метильовані цитозини незмінними. Ця методика пропонує роздільну здатність з однією основою, що дозволяє дослідникам всебічно досліджувати метилом і виявляти аномальні моделі метилювання, пов’язані з різними захворюваннями, зокрема раком. Використовуючи WGBS, вчені можуть отримати неперевершене розуміння ландшафтів метилювання в масштабах геному, забезпечуючи детальне розуміння епігенетичних механізмів, які лежать в основі різноманітних біологічних процесів і захворювань.
-

Аналіз транспозазно-доступного хроматину з високопродуктивним секвенуванням (ATAC-seq)
ATAC-seq — це високопродуктивна техніка секвенування, яка використовується для загальногеномного аналізу доступності хроматину. Його використання забезпечує глибше розуміння складних механізмів глобального епігенетичного контролю над експресією генів. У цьому методі використовується гіперактивна транспозаза Tn5 для одночасного фрагментування та позначення відкритих ділянок хроматину шляхом вставки адаптерів для секвенування. Подальша ПЛР-ампліфікація призводить до створення бібліотеки секвенування, яка дозволяє комплексно ідентифікувати відкриті ділянки хроматину в конкретних просторово-часових умовах. ATAC-seq забезпечує цілісне уявлення про доступні ландшафти хроматину, на відміну від методів, які зосереджені виключно на сайтах зв’язування транскрипційних факторів або конкретних ділянках, модифікованих гістоном. Шляхом секвенування цих відкритих ділянок хроматину ATAC-seq виявляє регіони, більш схильні до активних регуляторних послідовностей і потенційних сайтів зв’язування факторів транскрипції, пропонуючи цінну інформацію про динамічну модуляцію експресії генів у геномі.
-

16S/18S/ITS Амплікон секвенування-PacBio
Гени 16S і 18S рРНК разом із ділянкою внутрішнього транскрибованого спейсера (ITS) служать основними молекулярними маркерами відбитків пальців завдяки поєднанню висококонсервативних і гіперваріабельних ділянок, що робить їх безцінними інструментами для характеристики прокаріотичних і еукаріотичних організмів. Ампліфікація та секвенування цих регіонів пропонують підхід без ізоляції для дослідження мікробного складу та різноманітності в різних екосистемах. Хоча секвенування Illumina зазвичай націлено на короткі гіперваріабельні області, такі як V3-V4 16S та ITS1, було продемонстровано, що кращої таксономічної анотації можна досягти шляхом секвенування повної довжини 16S, 18S та ITS. Цей комплексний підхід забезпечує більший відсоток точно класифікованих послідовностей, досягаючи рівня роздільної здатності, що поширюється на ідентифікацію видів. Платформа секвенування однієї молекули в реальному часі (SMRT) від PacBio виділяється тим, що забезпечує високоточні довгі зчитування (HiFi), які охоплюють амплікони повної довжини, конкуруючи з точністю секвенування Illumina. Ця можливість дозволяє дослідникам отримати неперевершену перевагу — панорамний огляд генетичного ландшафту. Розширене охоплення значно підвищує роздільну здатність анотації видів, особливо в бактеріальних або грибкових спільнотах, дозволяючи глибше зрозуміти тонкощі мікробних популяцій.
-

16S/18S/ITS ампліконне секвенування-NGS
Секвенування ампліконів за допомогою технології Illumina, спеціально націлене на генетичні маркери 16S, 18S і ITS, є потужним методом для розкриття філогенії, таксономії та кількості видів у мікробних спільнотах. Цей підхід передбачає секвенування гіперваріабельних ділянок генетичних маркерів домашнього господарства. Спочатку був представлений як молекулярний відбиток пальцяWoeses та іну 1977 році ця методика зробила революцію в профілюванні мікробіома, дозволивши проводити аналізи без ізоляції. Завдяки секвенуванню 16S (бактерії), 18S (гриби) і внутрішнього транскрибованого спейсера (ITS, гриби) дослідники можуть ідентифікувати не лише численні види, але й рідкісні та невизначені. Широко прийняте як основний інструмент, секвенування ампліконів стало інструментом у розпізнаванні диференціальних мікробних композицій у різноманітних середовищах, включаючи людський рот, кишечник, стілець тощо.
-

Повторне секвенування повного генома бактерій і грибків
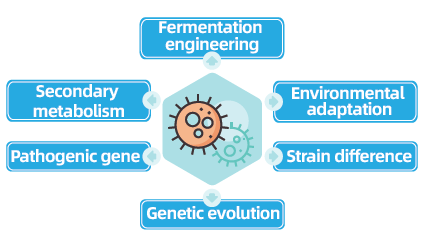
Проекти повторного секвенування повного генома бактерій і грибків є ключовими для вдосконалення мікробної геноміки, дозволяючи завершувати та порівнювати мікробні геноми. Це сприяє розробці ферментації, оптимізації промислових процесів і дослідженню вторинних шляхів метаболізму. Крім того, повторне секвенування грибів і бактерій має вирішальне значення для розуміння адаптації до навколишнього середовища, оптимізації штамів і виявлення динаміки генетичної еволюції, що має широке значення для медицини, сільського господарства та науки про навколишнє середовище.
-

PacBio-Повнорозмірне секвенування ампліконів 16S/18S/ITS
Платформа Amplicon (16S/18S/ITS) розроблена на основі багаторічного досвіду в аналізі проектів мікробного різноманіття, який містить стандартизований базовий аналіз і персоналізований аналіз: базовий аналіз охоплює основний вміст аналізу поточних мікробних досліджень, вміст аналізу багатий і вичерпний, а результати аналізу представлені у вигляді проектних звітів; Зміст персоналізованого аналізу різноманітний. Можна відбирати зразки та гнучко встановлювати параметри відповідно до звіту про базовий аналіз і мети дослідження, щоб реалізувати персоналізовані вимоги. Операційна система Windows, проста і швидка.
-

PacBio-повнорозмірний транскриптом (без посилання)
Використовуючи дані секвенування ізоформ Pacific Biosciences (PacBio) як вхідні дані, цей додаток може ідентифікувати повнорозмірні послідовності транскриптів (без збирання). Шляхом відображення повнорозмірних послідовностей із еталонним геномом можна оптимізувати транскрипти за допомогою відомих генів, транскриптів, кодуючих ділянок тощо. У цьому випадку можна досягти точнішої ідентифікації структур мРНК, наприклад альтернативного сплайсингу тощо. Спільний аналіз із даними секвенування транскриптомів NGS забезпечує більш повну анотацію та точнішу кількісну оцінку експресії на рівні транскрипту, що значною мірою сприяє диференціальній експресії та функціональному аналізу.
-

Бісульфітне секвенування зі зниженим представленням (RRBS)
Бісульфітне секвенування зі зниженим представленням (RRBS) стало економічно ефективною альтернативою бісульфітному секвенуванню цілого геному (WGBS) у дослідженнях метилювання ДНК. Незважаючи на те, що WGBS надає всебічну інформацію, досліджуючи весь геном з єдиною базовою роздільною здатністю, його висока вартість може бути обмежуючим фактором. RRBS стратегічно пом’якшує цю проблему шляхом вибіркового аналізу репрезентативної частини геному. Ця методологія ґрунтується на збагаченні ділянок, багатих острівцями CpG, шляхом розщеплення MspI з наступним відбором розміру фрагментів 200-500/600 bps. Отже, секвенуються лише області, проксимальні до острівців CpG, тоді як ті, що мають віддалені острівці CpG, виключаються з аналізу. Цей процес у поєднанні з бісульфітним секвенуванням дозволяє виявити метилювання ДНК з високою роздільною здатністю, а підхід до секвенування, PE150, фокусується саме на кінцях вставок, а не на середині, підвищуючи ефективність профілювання метилювання. RRBS є безцінним інструментом, який дозволяє економічно ефективне дослідження метилювання ДНК і покращує знання про епігенетичні механізми.
-

Секвенування прокаріотичної РНК
Секвенування РНК дає можливість комплексного профілювання всіх транскриптів РНК у клітинах за певних умов. Ця передова технологія служить потужним інструментом, що розкриває складні профілі експресії генів, генні структури та молекулярні механізми, пов’язані з різноманітними біологічними процесами. Секвенування РНК, яке широко застосовується у фундаментальних дослідженнях, клінічній діагностиці та розробці ліків, дає змогу зрозуміти тонкощі клітинної динаміки та генетичної регуляції. Наша обробка зразків прокаріотичної РНК розроблена для прокаріотичних транскриптомів, включаючи деплецію рРНК і спрямовану підготовку бібліотеки.
Платформа: Illumina NovaSeq
-

Секвенування метатранскриптому
Використовуючи технологію секвенування Illumina, сервіс секвенування метатранскриптомів BMKGENE розкриває динамічну експресію генів різноманітних мікробів, від еукаріот до прокаріотів і вірусів, у таких природних середовищах, як ґрунт, вода, море, кал і кишечник. Наша комплексна послуга дає дослідникам змогу досліджувати повні профілі експресії генів складних мікробних спільнот. Окрім таксономічного аналізу, наш сервіс секвенування метатранскриптомів полегшує дослідження функціонального збагачення, проливаючи світло на диференційовано експресовані гени та їх роль. Відкрийте для себе безліч біологічних ідей, орієнтуючись на складні ландшафти експресії генів, таксономічної різноманітності та функціональної динаміки в цих різноманітних екологічних нішах.
-

Збірка грибкового геному De novo
BMKGENE пропонує універсальні рішення для геномів грибів, що задовольняють різноманітні дослідницькі потреби та бажану повноту геному. Використання лише секвенування короткого зчитування Illumina дозволяє створити чернетку геному. Коротке зчитування та секвенування з довгим зчитуванням за допомогою Nanopore або Pacbio поєднуються для отримання більш тонкого генома гриба з довшими контигами. Крім того, інтеграція секвенування Hi-C ще більше розширює можливості, дозволяючи отримати повний геном на рівні хромосоми.