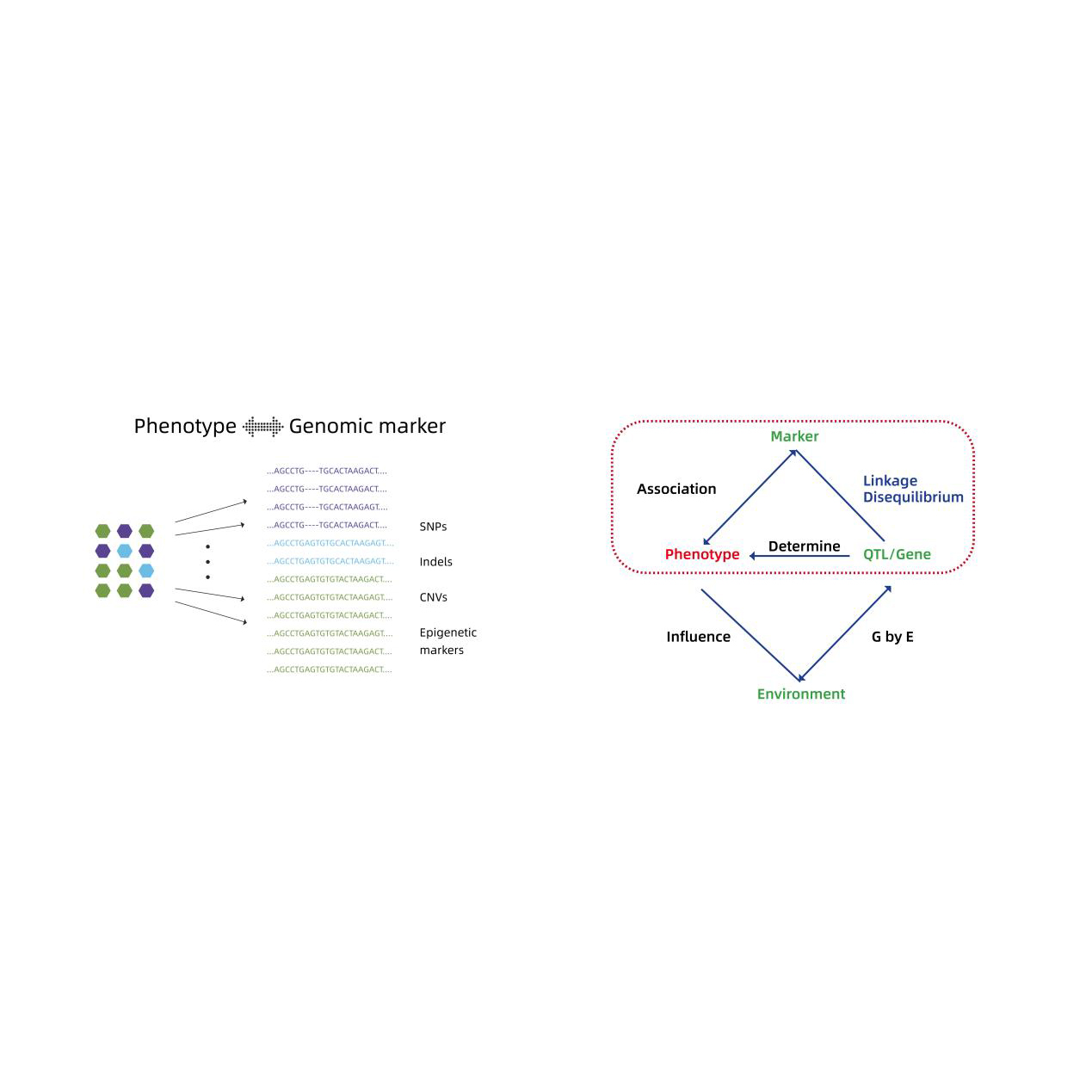
Аналіз асоціації в геномі
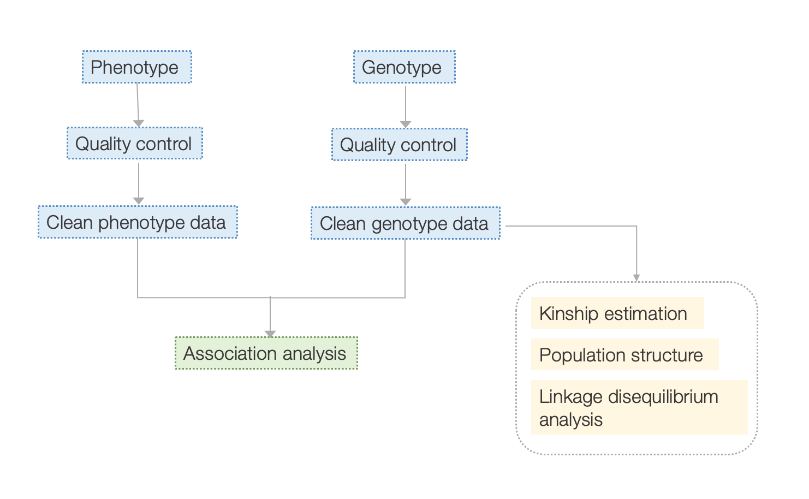
Робочий процес

Переваги послуг
●Широкі записи та публікації: З накопиченим досвідом роботи в GWAS, BMKGENE завершив сотні видовищних проектів у популяційних дослідженнях GWAS, допомагав дослідникам опублікувати понад 100 статей, а кумулятивний фактор впливу досяг 500.
● Комплексний аналіз біоінформатики: Робочий процес включає аналіз асоціації ознак SNP, надання набору генів-кандидатів та їх відповідної функціональної анотації.
●Висококваліфікована команда біоінформатики та цикл короткого аналізу: З великим досвідом вдосконаленого аналізу геноміки, команда BMKGENE надає комплексні аналізи з швидким часом повороту.
●Підтримка після продажу:Наше зобов’язання виходить за рамки завершення проекту з 3-місячним післяпродажним періодом. За цей час ми пропонуємо подальше спостереження за проектами, допомогу у вирішенні несправностей та сесії запитань та запитань для вирішення будь-яких запитів, пов'язаних з результатами.
Технічні характеристики та вимоги
| Тип секвенування | Рекомендована шкала населення | Стратегія послідовності | Нуклеотидні вимоги |
| Ціле секвенування геному | 200 зразків | 10 разів | Концентрація: ≥ 1 нг/ мкл Загальна кількість ≥ 30ng Обмежена або відсутність деградації або забруднення |
| Помилковий фрагмент підсилювача (SLAF) (SLAF) | Глибина тегу: 10x Кількість тегів: <400 Мб: Рекомендується WGS <1 Гб: 100 к. Теги 1 Гб > 2 Гб: 300K теги Максимум теги 500k | Концентрація ≥ 5 нг/мкл Загальна кількість ≥ 80 нг Nanodrop OD260/280 = 1,6-2,5 Агарозний гель: немає або обмеженої деградації або забруднення
|
Вибір матеріалу



Різні сорти, підвид, земля/генбанки/змішані сім'ї/дикі ресурси
Різні сорти, підвид, земля
Сімейна/Сімейна/Диких ресурсів
РОБОТИ РОБОТА

Дизайн експерименту

Доставка зразків

Екстракція РНК

Будівництво бібліотеки

Послідовність

Аналіз даних

Послуги після продажу
Включає такий аналіз:
- Аналіз асоціації в геномі: LM, LMM, EMMAX, FASTLMM Модель
- Функціональна анотація кандидатських генів
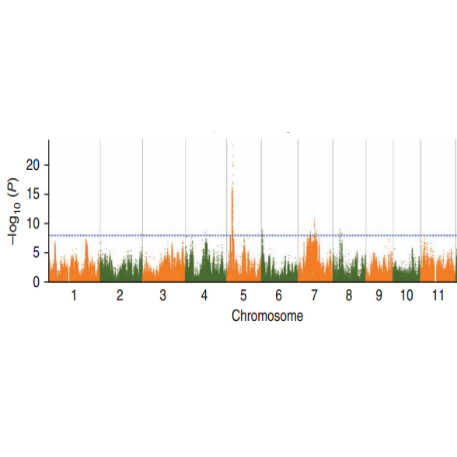
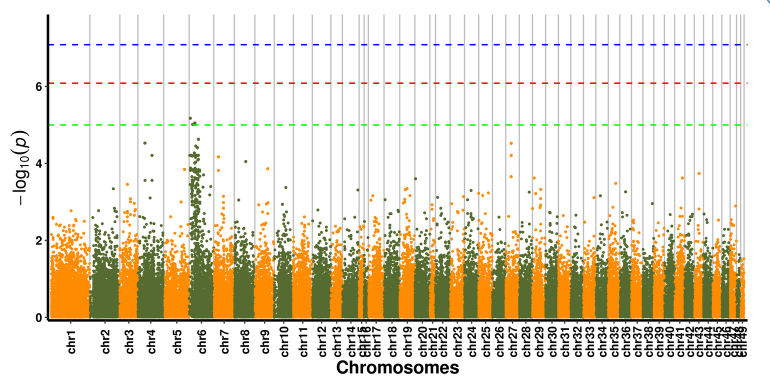
Аналіз асоціації SNP-ознак-Манхеттенський сюжет
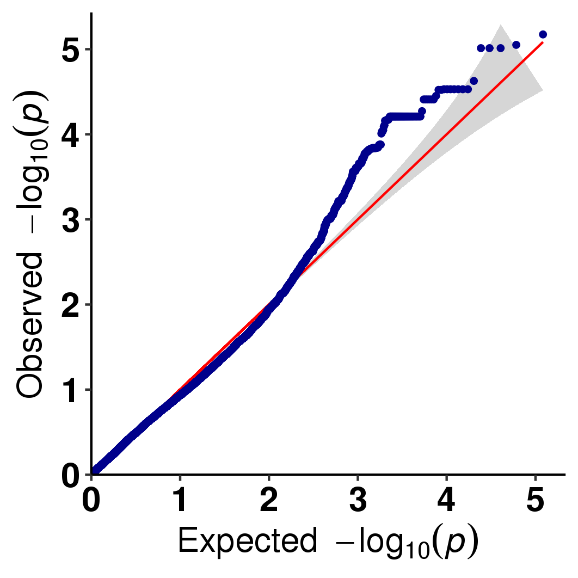
Аналіз асоціації SNP-ознак-графік QQ
Дослідіть просування, спричинені послугами De Gwas Bmkgene через кураторну колекцію публікацій:
LV, L. та ін. (2023) "Ознайомлення з генетичною основою толерантності до аміаку в бритви Clam Sinonovacula Constricta шляхом дослідження асоціації, що стосується геному",Аквакультура, 569, с. 739351. Doi: 10.1016/j.aquaculture.2023.739351.
Лі, X. та ін. (2022) "Багатоомічні аналізи 398 приєднання до проса Foxtail розкривають геномні області, пов'язані з одомашненням, ознаками метаболіту та протизапальними ефектами",Молекулярна рослина, 15 (8), с. 1367–1383. doi: 10.1016/j.molp.2022.07.003.
Li, J. та ін. (2022) «Картографування геномів, що стосуються бездоганних фенотипів у середовищі посухи»,Кордони в науці про рослини, 13, с. 924892. Doi: 10.3389/fpls.2022.924892/bibtex.
Чжао, X. та ін. (2021) 'GMST1, який кодує сульфотрансферазу, надає стійкість до вірусу мозаїки сої G2 та G3',Рослина, клітини та навколишнє середовище, 44 (8), с. 2777–2792. doi: 10.1111/pce.14066.