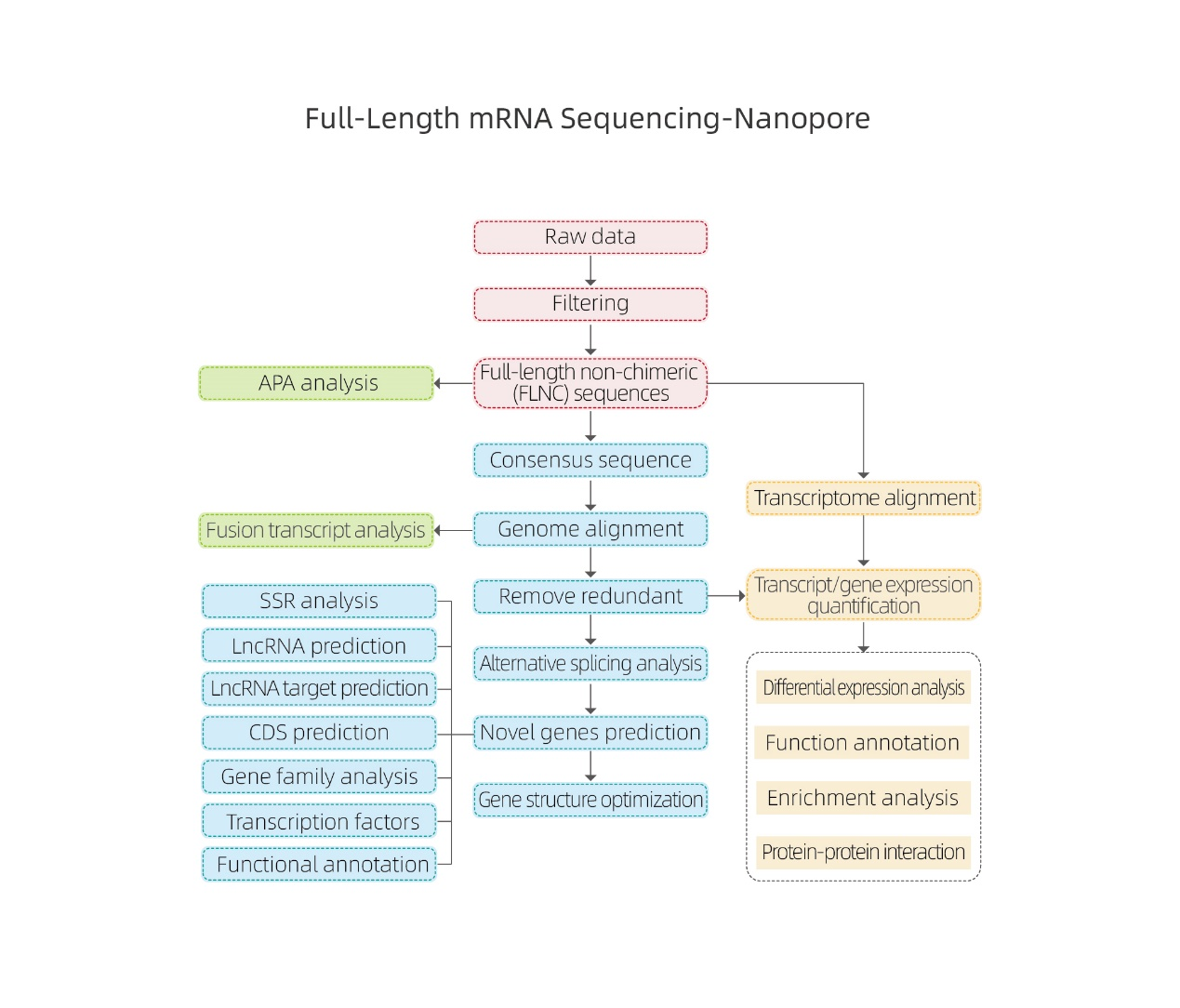
Повнорозмірна мРНК секвенування-нановори
Особливості
● Захоплення мРНК з подальшим синтезом кДНК та підготовкою бібліотеки
● Послідовність стенограм повної довжини
● Біоінформатичний аналіз на основі вирівнювання до референтного геному
● Біоінформатичний аналіз включає не лише експресію на генному та ізоформованому рівні, але й аналіз lncrNA, злиття генів, полі-аденілювання та структури генів
Переваги послуг
●Кількісне визначення експресії на рівні ізоформи: Увімкнення детального та точного аналізу експресії, розкриття зміни, яка може бути замаскована при аналізі всієї експресії генів
●Зменшені вимоги до даних:Порівняно з секвенуванням наступного покоління (NGS), нанопорове секвенування виявляє менші вимоги до даних, що дозволяє рівнозначні рівні кількісного насичення експресії генів з меншими даними.
●Більш висока точність кількісного визначення виразів: І на рівні гена, і ізоформ
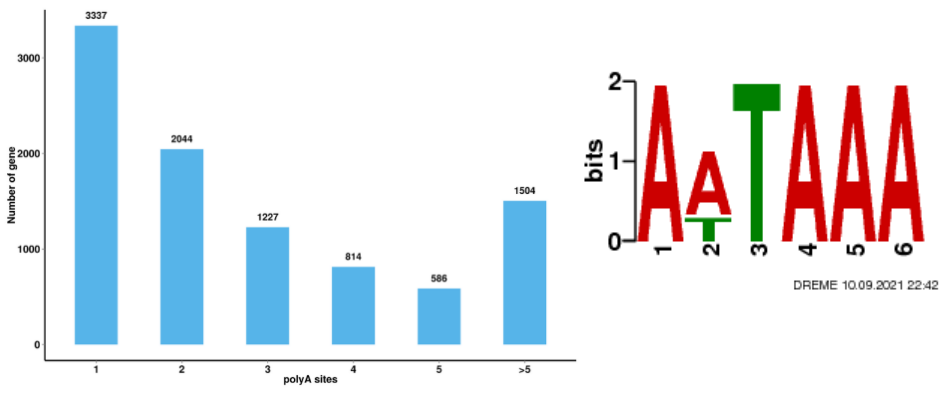
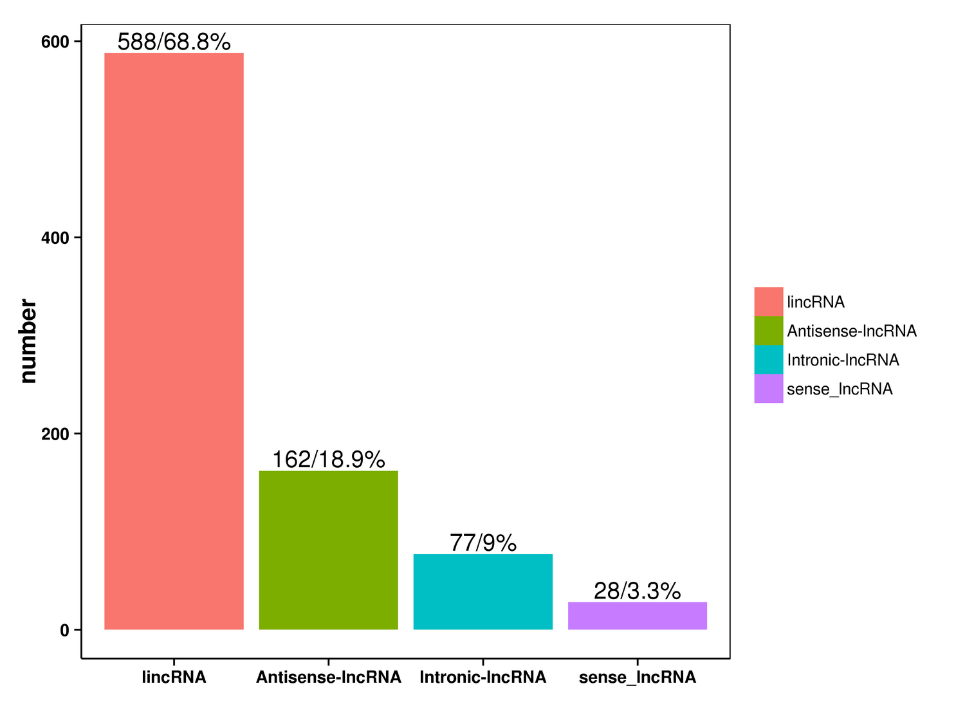
●Ідентифікація додаткової транскриптомічної інформації: Альтернативне поліаденілювання, гени злиття та lcnRNA та їх цільові гени
●Широка експертиза: Наша команда приносить багатий досвід для кожного проекту, виконавши понад 850 нанопорів проектів на повнометражну стенограму та обробляли понад 8000 зразків.
●Підтримка після продажу: Наше зобов’язання виходить за рамки завершення проекту з 3-місячним післяпродажним періодом. За цей час ми пропонуємо подальше спостереження за проектами, допомогу у вирішенні несправностей та сесії запитань та запитань для вирішення будь-яких запитів, пов'язаних з результатами.
Зразки вимог та доставки
| Бібліотека | Стратегія послідовності | Рекомендовані дані | Контроль якості |
| Полі збагачений | Illumina pe150 | 6/12 Гб | Середній показник якості: Q10 |
Вимоги до вибірки:
Нуклеотиди:
| Конц. (Ng/мкл) | Кількість (мкг) | Чистота | Цілісність |
| ≥ 100 | ≥ 1,0 | OD260/280 = 1,7-2,5 OD260/230 = 0,5-2,5 Обмежене або відсутність білка або забруднення ДНК, показаного на гелі. | Для рослин: rin≥7,0; Для тварин: rin≥7,5; 5,0≥28S/18S≥1,0; обмежена або відсутність базової висоти |
● Рослини:
Корінь, стебло або пелюстка: 450 мг
Лист або насіння: 300 мг
Плід: 1,2 г
● Тварина:
Серце чи кишечник: 300 мг
Вісцера або мозок: 240 мг
М'яз: 450 мг
Кістки, волосся або шкіра: 1г
● членистоноги:
Комахи: 6G
Раковина: 300 мг
● Цільна кров: 1 трубка
● Клітини: 106 клітини
Рекомендована доставка зразків
Контейнер: 2 мл центрифугової трубки (олов'яна фольга не рекомендується)
Маркування зразків: група+повторення, наприклад, A1, A2, A3; B1, B2, B3.
Відвантаження:
1. Сухий погляд: зразки потрібно упакувати в мішках і закопувати в сухий льоду.
2. РНАТУРАТИВНІ ТУБИ: Зразки РНК можна сушити в трубці стабілізації РНК (наприклад, RNASTABLE®) та відправляти в кімнатну температуру.
РОБОТИ РОБОТА
Нуклеотиди:

Доставка зразків

Будівництво бібліотеки

Послідовність

Аналіз даних

Послуги після продажу
РОБОТИ РОБОТА
Тканина:

Дизайн експерименту

Доставка зразків

Екстракція РНК

Будівництво бібліотеки

Послідовність

Аналіз даних

Послуги після продажу
● Обробка необроблених даних
● Ідентифікація стенограми
● Альтернативне сплайсинг
● Кількісне визначення експресії на рівні генів та ізоформ
● Диференціальний аналіз експресії
● Анотація функцій та збагачення (DEGS та DETS)
Альтернативний аналіз сплайсингу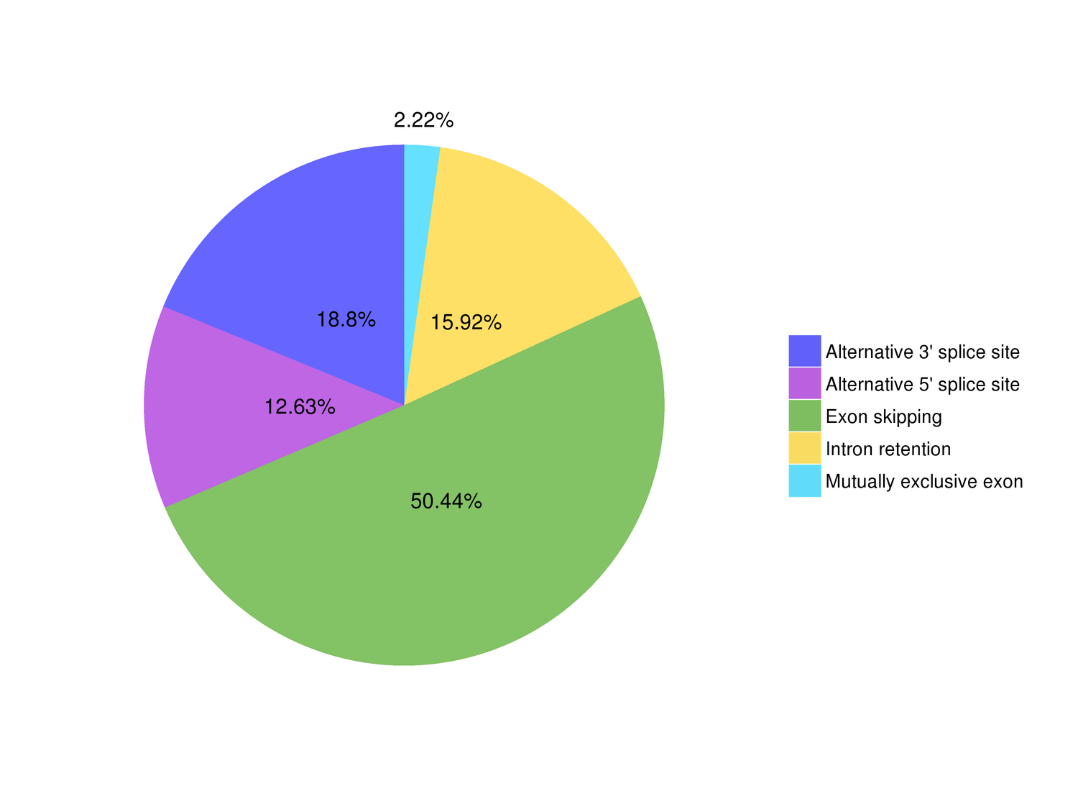 Альтернативний аналіз поліаденілювання (APA)
Альтернативний аналіз поліаденілювання (APA)
прогнозування lncrna
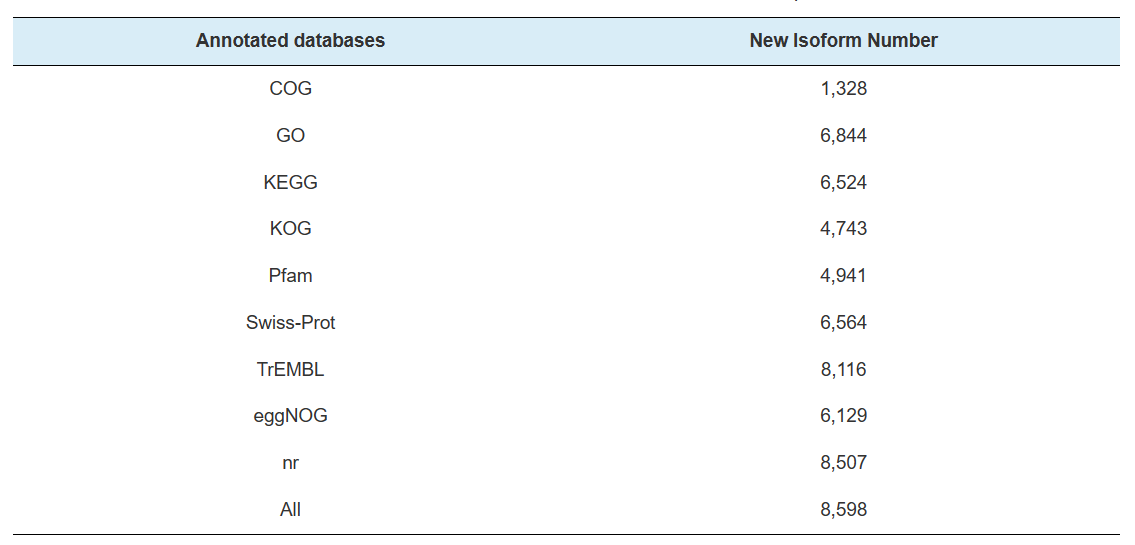
Анотація нових генів
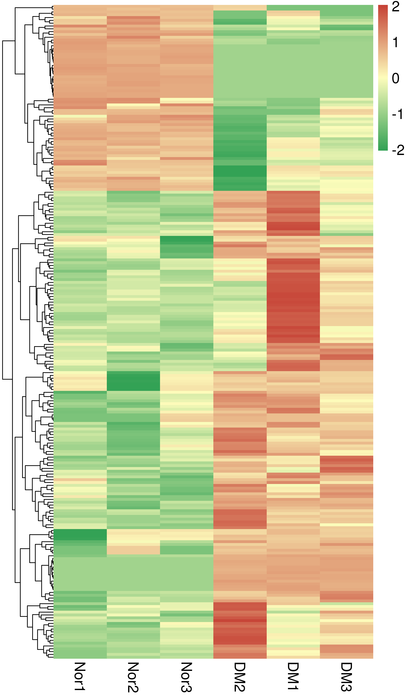
Кластеризація DETS
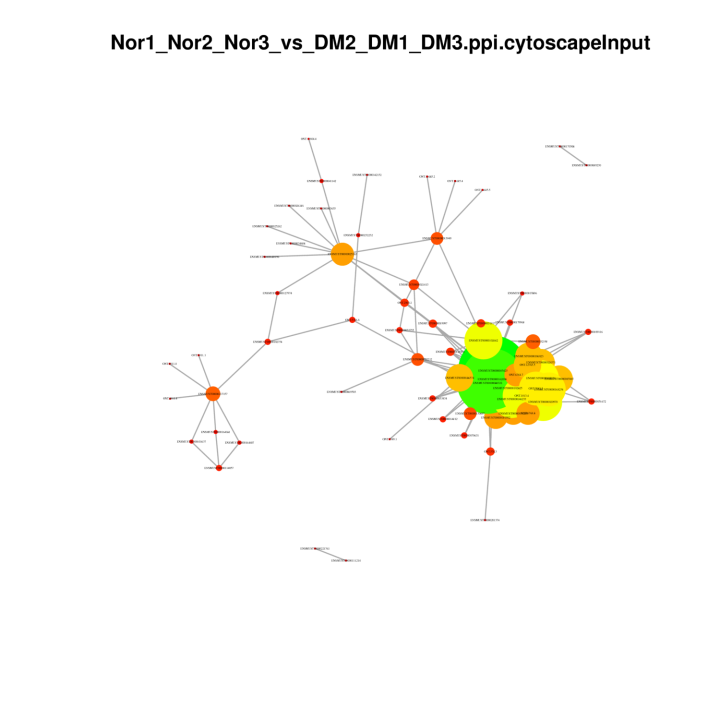
Білкові білкові мережі в DEGS
Дослідіть просування, спричинені наноповими послугами послідовності мРНК BMKGENE через кураторну колекцію публікацій.
Гонг, Б. та ін. (2023) "Епігенетична та транскрипційна активація секреторної кінази FAM20C як онкоген в гліому", Journal of Genetics and Genomics, 50 (6), с. 422–433. doi: 10.1016/j.jgg.2023.01.008.
Він, З. та ін. (2023) "Повнорозмірне транскриптом послідовності лімфоцитів реагує на IFN-γ, виявляє імунну відповідь Th1-Skewed у Flounder (Paralichthys Olivaceus)", імунологія риб та молюсків, 134, с. 108636. Doi: 10.1016/j.fsi.2023.108636.
Ма, Ю. та ін. (2023) "Порівняльний аналіз методів секвенування РНК Pacbio та ONT для ідентифікації отрути Nemopilema nomurai", геноміка, 115 (6), с. 110709. Doi: 10.1016/j.ygeno.2023.110709.
Yu, D. et al. (2023) 'Нано-Seq аналіз виявляє різну функціональну тенденцію між екзосомами та мікровезикулами, отриманими з HUMSC', дослідженням та терапією стовбурових клітин, 14 (1), с. 1–13. doi: 10.1186/S13287-023-03491-5/Таблиці/6.