Повторне секвенування повного генома бактерій і грибків
Переваги сервісу
●Біоінформаційний аналіз включає варіанти виклику:Надання функціональної інформації про повторно секвеновані геноми.
● Великий досвід: Тисячі проектів повторного секвенування мікроорганізмів, які проводяться щорічно, ми пропонуємо понад десятиліття досвіду, висококваліфіковану аналітичну команду, повний контент і чудову підтримку після продажу.
●Підтримка після продажу:Наше зобов’язання поширюється на період після завершення проекту з 3-місячним періодом післяпродажного обслуговування. Протягом цього часу ми пропонуємо подальше спостереження за проектом, допомогу з усунення несправностей і сеанси запитань і відповідей, щоб відповісти на будь-які запитання, пов’язані з результатами.
Специфікації послуги
| Платформа секвенування | Стратегія секвенування | Рекомендовані дані | Контроль якості |
| Illumina NovaSeq | PE150 | Глибина 100х | Q30≥85% |
Зразок вимог
| Концентрація (нг/мкл) | Загальна кількість (нг) | Об'єм (мкл) |
| ≥1 | ≥60 | ≥20 |
Бактерії: ≥1x107 клітини
Одноклітинні гриби: ≥5x106-1x107 клітини
Макро гриби: ≥4 г
Потік роботи служби

Доставка зразків

Будівництво бібліотеки

Секвенування

Аналіз даних

Післяпродажне обслуговування
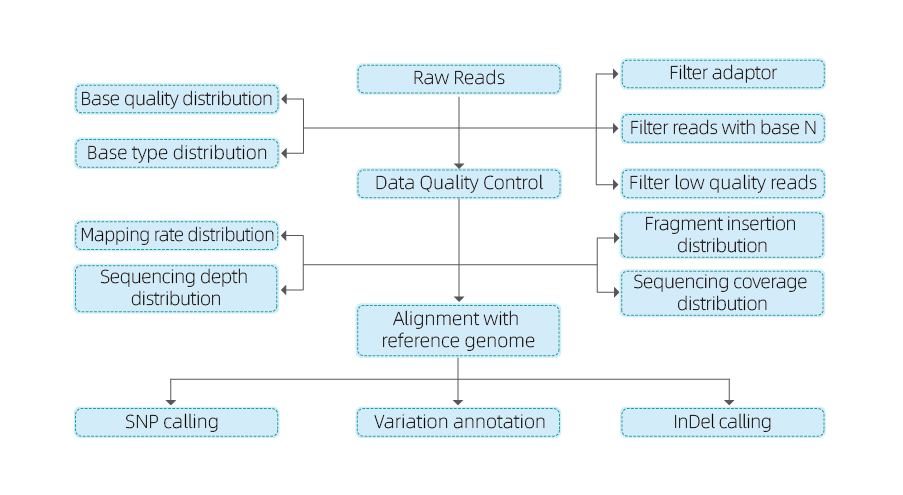
Включає наступний аналіз:
- Контроль якості даних секвенування
- Вирівнювання з еталонним геномом
- Виклик варіантів: SNP та InDel
- Анотація варіанта
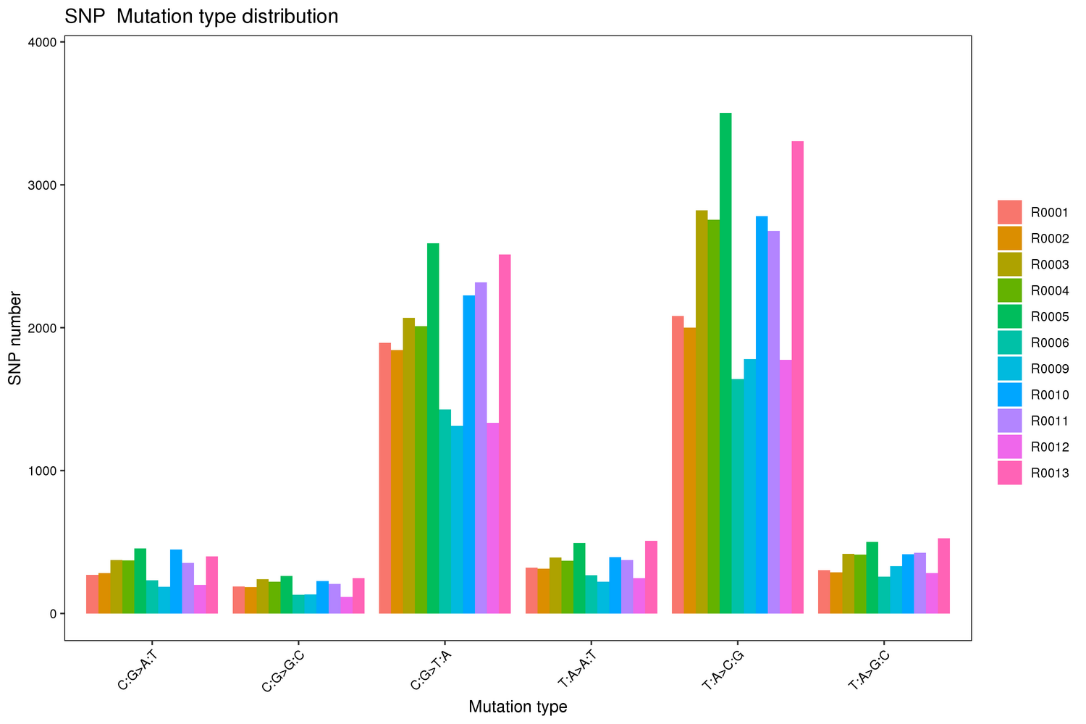
Виклик варіантів: типи SNP
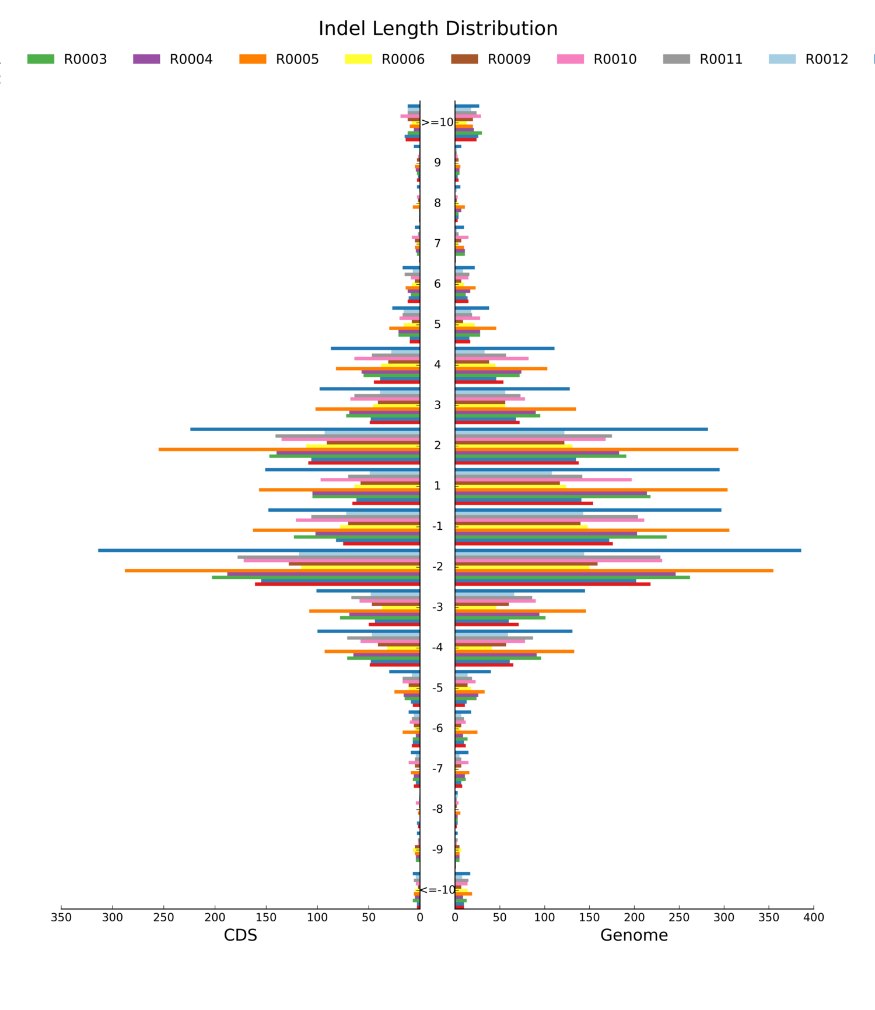
Виклик варіанту: розподіл довжини InDel
Досліджуйте досягнення, які сприяють послуги повторного секвенування мікробного геному BMKGene, за допомогою підібраної колекції публікацій.
Jia, Y. та ін. (2023) «Поєднання повторного секвенування транскриптомів і цілого генома для скринінгу генів стійкості до хвороб пшеничної карликової головки»,Міжнародний журнал молекулярних наук, 24(24). doi: 10.3390/IJMS242417356.
Jiang, M. та ін. (2023) «Контрольований ампіциліном метаболізм глюкози маніпулює переходом від толерантності до резистентності у бактерій»,Досягнення науки, 9(10). doi: 10.1126/SCIADV.ADE8582/SUPPL_FILE/SCIADV.ADE8582_SM.PDF.
Yang, M. та ін. (2022) «Aliidiomarina halalkaliphila sp. nov., галоалкаліфільна бактерія, виділена з содового озера в автономному районі Внутрішня Монголія, Китай', Int. J. Syst. еволюціямікробіол, 72, стор. 5263. doi: 10.1099/ijsem.0.005263.
Чжу, З., Ву, Р. і Ван, Г.-Х. (2024) «Послідовність геному Staphylococcus nepalensis ZZ-2023a, виділеного з Nasonia vitripennis»,Оголошення мікробіологічних ресурсів. doi: 10.1128/MRA.00802-23.