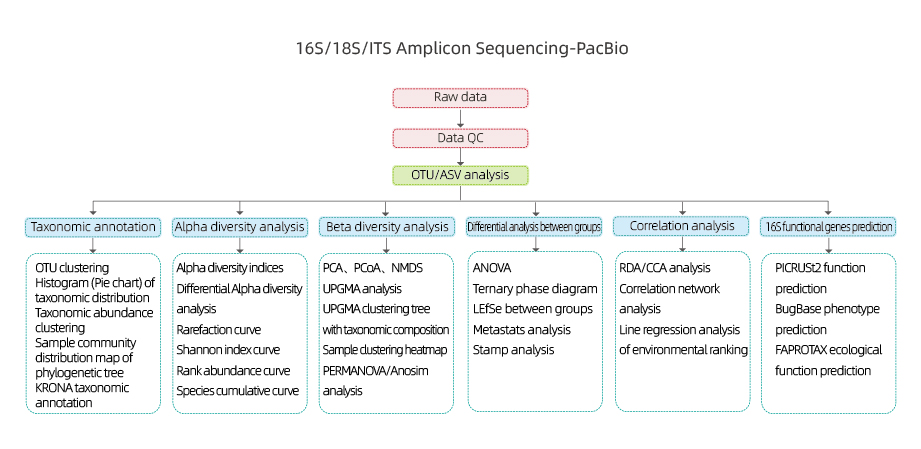
16S/18S/ITS Амплікон секвенування-PacBio
Особливості сервісу
● Платформа секвенування: PacBio Revio
● Режим секвенування: CCS (HiFi читання)
● Ампліфікація цільової області з подальшим тандемним зв'язуванням ампліконів перед підготовкою бібліотеки дзвонів HiFi SMRT
Переваги сервісу
●Вища таксономічна роздільна здатність: Tсеквенування короткого амплікону han,забезпечуючи вищі показники класифікації OTU на рівні видів.
●Високоточний базовий виклик: послідовність режиму PacBio CCS (зчитування HiFi).
●Без ізоляції: Швидка ідентифікація мікробного складу в зразках навколишнього середовища.
●Широке застосування: Дослідження різноманітних мікробних спільнот.
●Комплексний біоінформаційний аналіз: найновіший пакет QIIME2 (кількісне розуміння мікробної екології) з різноманітними аналізами з точки зору бази даних, анотацій, OTU/ASV.
●Великий досвід: Завдяки тисячам проектів із секвенування ампліконів, що проводяться щорічно, BMKGENE пропонує більше десяти років досвіду, висококваліфіковану аналітичну команду, повний контент і чудову післяпродажну підтримку.
Специфікації послуги
| Бібліотека | Стратегія секвенування | Рекомендовані дані |
| Амплікон | PacBio Revio | Теги 10/30/50 K (CCS) |
Зразок вимог
| Концентрація (нг/мкл) | Загальна кількість (мкг) | Об'єм (мкл) |
| ≥5 | ≥0,3 | ≥20 |
Рекомендована доставка зразків
Заморожуйте зразки в рідкому азоті протягом 3-4 годин і зберігайте в рідкому азоті або -80 градусів для тривалого зберігання. Потрібна доставка зразків із сухим льодом.
Потік роботи служби

Доставка зразків

Будівництво бібліотеки

Секвенування

Аналіз даних

Післяпродажне обслуговування
Включає наступний аналіз:
●Контроль якості необроблених даних
●OTU кластеризація/De-Noise (ASV)
●OTU анотація
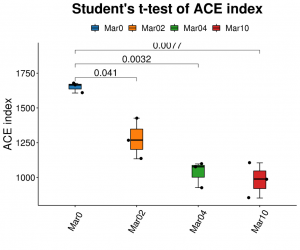
●Аналіз альфа-різноманіття: кілька індексів, включаючи Шеннона, Сімпсона та ACE.
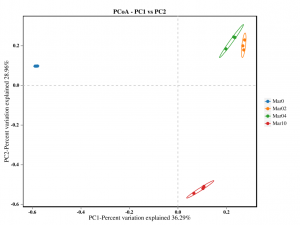
●Бета-аналіз різноманітності
●Міжгруповий аналіз
● Кореляційний аналіз: між факторами навколишнього середовища та складом і різноманітністю OUT
● Функціональне прогнозування гена 16S
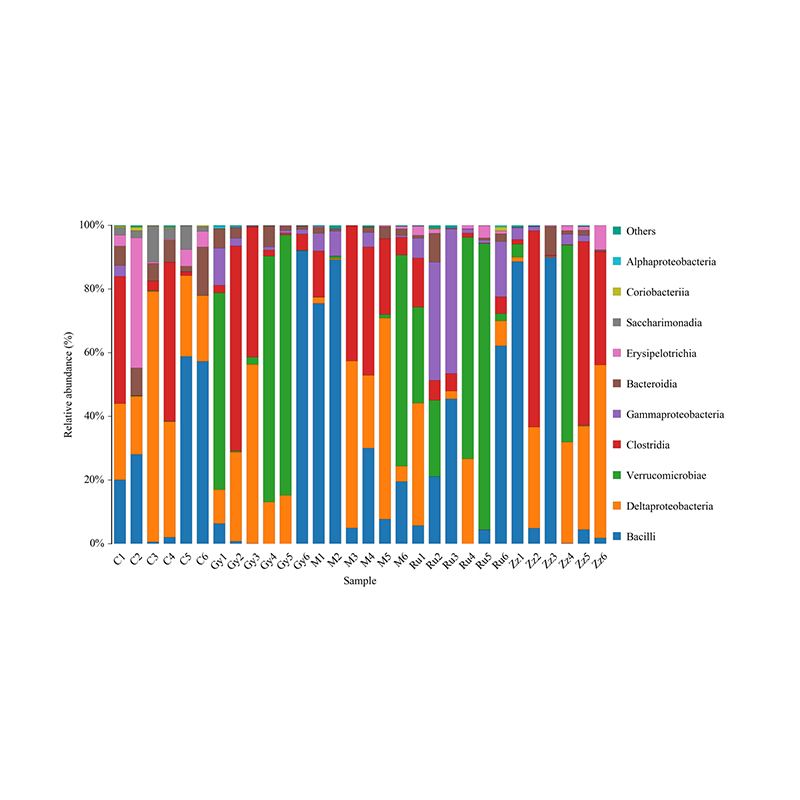
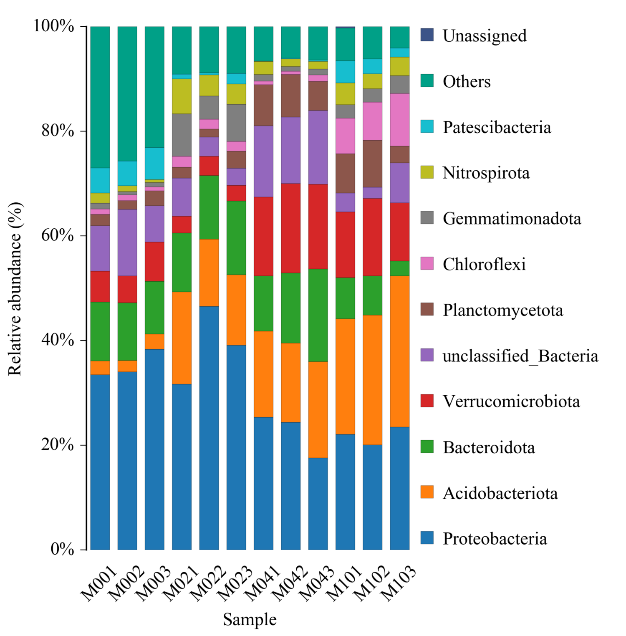
Гістограма таксономічного розподілу
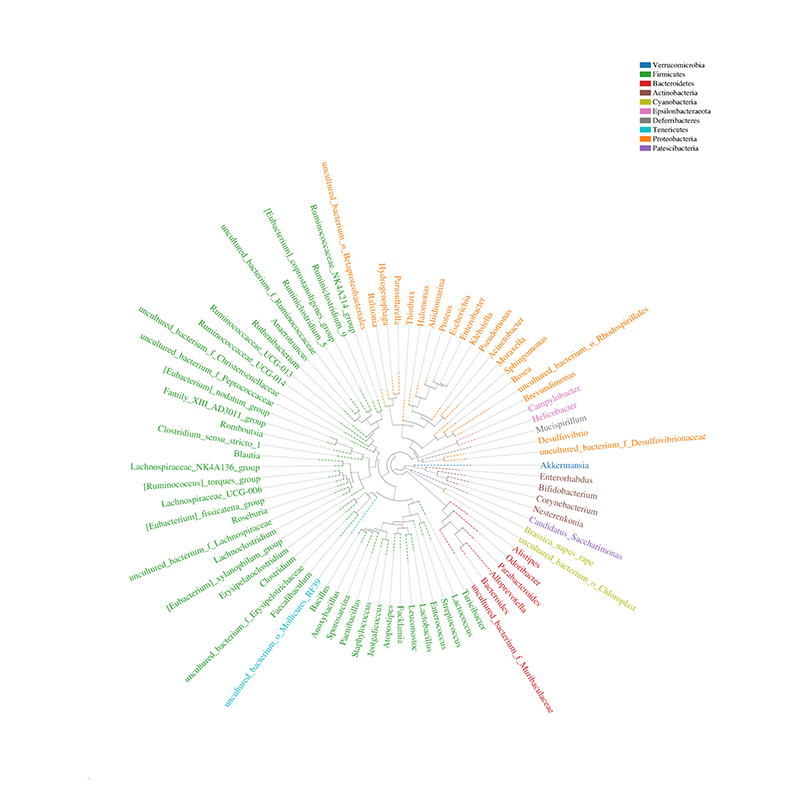
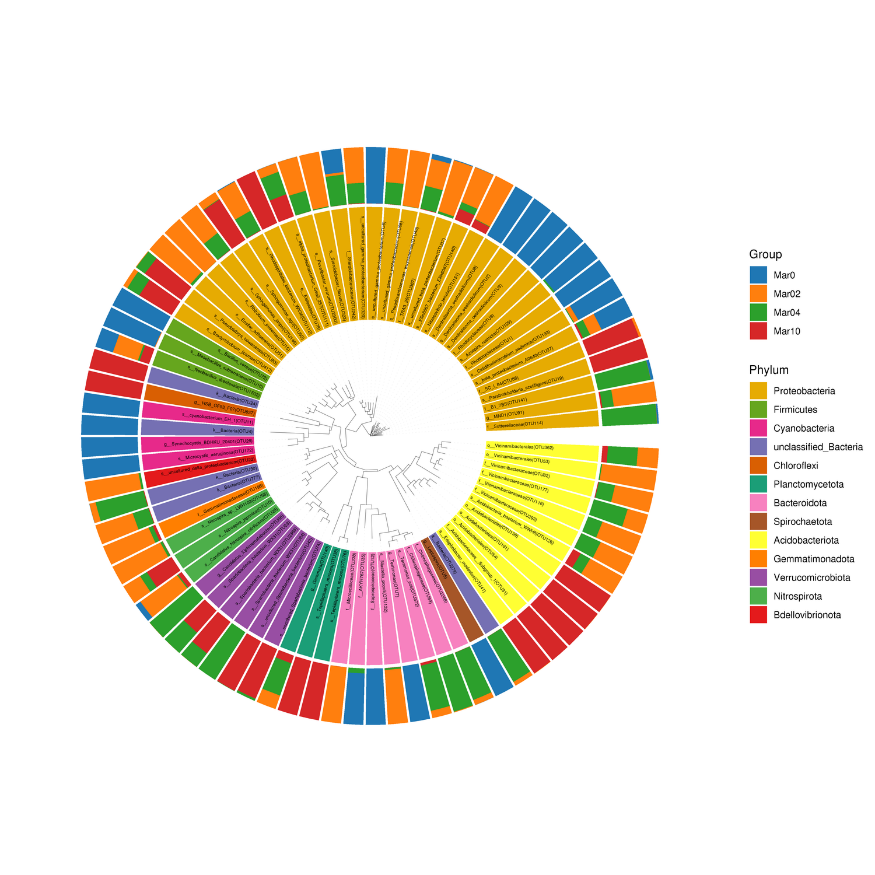
Філогенетичне дерево розподілу спільноти
Альфа-аналіз різноманітності: ACE
Бета-аналіз різноманітності: PCoA
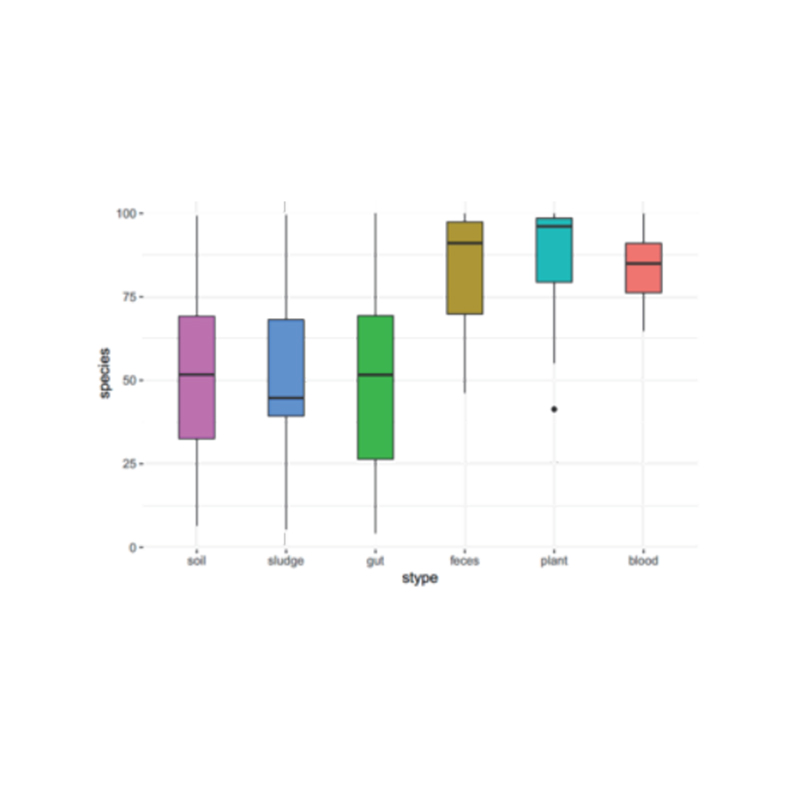
Міжгруповий аналіз: ANOVA
Ознайомтеся з досягненнями завдяки службам секвенування ампліконів BMKGene за допомогою PacBio через підібрану колекцію публікацій.
Gao, X. and Wang, H. (2023) «Порівняльний аналіз профілів і функцій рубця бактерій під час адаптації до різної фенології (зелені та трав’янисті) у альпійських мериносових овець із двома стадіями росту на альпійському лузі», Ферментація, 9( 1), стор. 16. doi: 10.3390/FERMENTATION9010016/S1.
Li, S. та ін. (2023) «Захоплення мікробної темної матерії в ґрунтах пустелі за допомогою метагеноміки на основі культуроміки та аналізу високої роздільної здатності», npj Biofilms and Microbiomes 2023 9:1, 9(1), стор. 1–14. doi: 10.1038/s41522-023-00439-8.
Mu, L. та ін. (2022) «Вплив солей жирних кислот на характеристики бродіння, бактеріальне різноманіття та аеробну стабільність змішаного силосу, приготовленого з люцерни, рисової соломи та пшеничних висівок», Journal of Science of Food and Agriculture, 102(4), стор. 1475– 1487. doi: 10.1002/JSFA.11482.
Yang, J. та ін. (2023) «Взаємодія між біомаркерами окисного стресу та кишковою мікробіотою в антиоксидантній дії екстрактів Sonchus brachyotus DC». в Intestinal Oxidative Stress in Intestinal Oxidative Stres in Adult Zebrafish', Antioxidants 2023, Vol. 12, Сторінка 192, 12(1), стор. 192. doi: 10.3390/ANTIOX12010192.