-

Metagenomic Sequencing -NGS
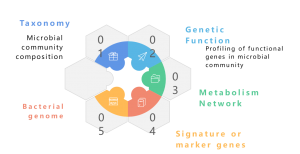
Ang metagenome ay isang koleksyon ng kabuuang genetic na materyal ng isang halo-halong komunidad ng mga organismo, tulad ng mga metagenome sa kapaligiran at pantao. Naglalaman ito ng mga genome ng parehong cultivatable at uncultivatable microorganisms. Ang shotgun metagenomic sequencing na may NGS ay nagbibigay-daan sa pag-aaral ng mga masalimuot na genomic na landscape na ito na naka-embed sa mga sample ng kapaligiran sa pamamagitan ng pagbibigay ng higit pa sa taxonomic profiling, na nagbibigay din ng mga butil na insight sa pagkakaiba-iba ng species, abundance dynamics, at kumplikadong istruktura ng populasyon. Higit pa sa pag-aaral ng taxonomic, ang shotgun metagenomics ay nag-aalok din ng functional genomics perspective, na nagbibigay-daan sa paggalugad ng mga naka-encode na gene at ang kanilang mga putative na tungkulin sa mga prosesong ekolohikal. Sa wakas, ang pagtatatag ng mga network ng ugnayan sa pagitan ng mga genetic na elemento at mga kadahilanan sa kapaligiran ay nag-aambag sa isang holistic na pag-unawa sa masalimuot na interplay sa pagitan ng mga microbial na komunidad at kanilang ekolohikal na background. Sa konklusyon, ang metagenomic sequencing ay nakatayo bilang isang pivotal instrument para sa pag-alis ng genomic intricacies ng magkakaibang microbial na komunidad, na nagbibigay-liwanag sa mga multifaceted na relasyon sa pagitan ng genetics at ecology sa loob ng mga kumplikadong ecosystem na ito.
Mga Platform: Illumina NovaSeq at DNBSEQ-T7
-

Metagenomic Sequencing-TGS
Ang metagenome ay isang koleksyon ng genetic na materyal ng isang halo-halong komunidad ng mga organismo, tulad ng mga metagenome sa kapaligiran at tao. Naglalaman ito ng mga genome ng parehong cultivatable at uncultivatable microorganisms. Ang metagenomic sequencing ay nagbibigay-daan sa pag-aaral ng mga masalimuot na genomic na landscape na ito na naka-embed sa mga ecological sample sa pamamagitan ng pagbibigay ng higit pa sa taxonomic profiling. Nag-aalok din ito ng isang functional na pananaw ng genomics sa pamamagitan ng paggalugad sa mga naka-encode na gene at ang kanilang mga papel na ginagampanan sa mga proseso sa kapaligiran. Bagama't malawakang ginagamit ang tradisyonal na shotgun sa pagkakasunud-sunod ng Illumina sa metagenomic na pag-aaral, ang pagdating ng Nanopore at PacBio na matagal nang nabasa na pagkakasunud-sunod ay nagbago sa larangan. Pinahusay ng teknolohiya ng Nanopore at PacBio ang mga downstream na bioinformatic na pagsusuri, lalo na ang metagenome assembly, na tinitiyak ang higit pang tuluy-tuloy na mga pagtitipon. Ang mga ulat ay nagpapahiwatig na ang Nanopore-based at PacBio-based na metagenomics ay matagumpay na nakabuo ng kumpleto at saradong bacterial genome mula sa mga kumplikadong microbiome (Moss, EL, et al., Nature Biotech, 2020). Ang pagsasama ng Nanopore reads sa Illumina reads ay nagbibigay ng isang madiskarteng diskarte para sa pagwawasto ng error, na nagpapagaan sa likas na mababang katumpakan ng Nanopore. Ang synergistic na kumbinasyong ito ay gumagamit ng mga lakas ng bawat sequencing platform, na nag-aalok ng isang matatag na solusyon upang mapagtagumpayan ang mga potensyal na limitasyon at isulong ang katumpakan at pagiging maaasahan ng metagenomic na pagsusuri.
Platform: Nanopore PromethION 48, Illumia at PacBio Revio
-

Buong genome bisulfite sequencing (WGBS)
Ang Whole Genome Bisulfite Sequencing (WGBS) ay tumatayo bilang gold-standard na pamamaraan para sa malalim na paggalugad ng DNA methylation, partikular ang ikalimang posisyon sa cytosine (5-mC), isang pivotal regulator ng gene expression at cellular activity. Ang prinsipyong pinagbabatayan ng WGBS ay nagsasangkot ng paggamot ng bisulfite, na nag-uudyok sa conversion ng mga unmethylated cytosines sa uracil (C to U), habang iniiwan ang mga methylated cytosine na hindi nagbabago. Ang diskarteng ito ay nag-aalok ng solong-base na resolusyon, na nagpapahintulot sa mga mananaliksik na komprehensibong mag-imbestiga sa methylome at mag-alis ng mga abnormal na pattern ng methylation na nauugnay sa iba't ibang mga kondisyon, lalo na ang cancer. Sa pamamagitan ng paggamit ng WGBS, ang mga siyentipiko ay makakakuha ng walang kapantay na mga insight sa genome-wide methylation landscape, na nagbibigay ng isang nuanced na pag-unawa sa mga mekanismo ng epigenetic na sumasailalim sa magkakaibang biological na proseso at sakit.
-

Assay para sa Transposase-Accessible Chromatin na may High Throughput Sequencing (ATAC-seq)
Ang ATAC-seq ay isang high-throughput na sequencing technique na ginagamit para sa genome-wide chromatin accessibility analysis. Ang paggamit nito ay nagbibigay ng mas malalim na pag-unawa sa mga kumplikadong mekanismo ng pandaigdigang kontrol ng epigenetic sa pagpapahayag ng gene. Gumagamit ang pamamaraan ng hyperactive Tn5 transposase upang sabay na i-fragment at i-tag ang mga bukas na rehiyon ng chromatin sa pamamagitan ng paglalagay ng mga sequencing adapter. Ang kasunod na PCR amplification ay nagreresulta sa paglikha ng isang sequencing library, na nagbibigay-daan para sa komprehensibong pagkakakilanlan ng mga bukas na rehiyon ng chromatin sa ilalim ng mga partikular na kondisyon ng space-time. Nagbibigay ang ATAC-seq ng holistic na view ng mga naa-access na chromatin landscape, hindi katulad ng mga pamamaraan na nakatuon lang sa mga transcription factor binding site o partikular na histone-modified na mga rehiyon. Sa pamamagitan ng pagkakasunud-sunod sa mga bukas na rehiyon ng chromatin na ito, ipinapakita ng ATAC-seq ang mga rehiyon na mas malamang sa mga aktibong pagkakasunud-sunod ng regulasyon at potensyal na mga site na nagbubuklod ng transcription factor, na nag-aalok ng mahalagang mga pananaw sa dinamikong modulasyon ng expression ng gene sa buong genome.
-

16S/18S/ITS Amplicon Sequencing-PacBio
Ang 16S at 18S rRNA genes, kasama ang Internal Transcribed Spacer (ITS) na rehiyon, ay nagsisilbing pivotal molecular fingerprinting marker dahil sa kumbinasyon ng mga ito ng lubos na conserved at hyper-variable na mga rehiyon, na ginagawa itong napakahalagang mga tool para sa pagkilala sa mga prokaryotic at eukaryotic na organismo. Ang amplification at sequencing ng mga rehiyong ito ay nag-aalok ng isang isolation-free na diskarte para sa pagsisiyasat sa microbial composition at pagkakaiba-iba sa iba't ibang ecosystem. Habang ang pagkakasunud-sunod ng Illumina ay karaniwang nagta-target ng mga maikling hypervariable na rehiyon tulad ng V3-V4 ng 16S at ITS1, ipinakita na ang superior taxonomic annotation ay makakamit sa pamamagitan ng sequencing ng buong haba ng 16S, 18S, at ITS. Ang komprehensibong diskarte na ito ay nagreresulta sa mas mataas na porsyento ng mga tumpak na inuri na pagkakasunud-sunod, na nakakamit ng isang antas ng resolusyon na umaabot sa pagkakakilanlan ng mga species. Namumukod-tangi ang Single-Molecule Real-Time (SMRT) sequencing platform ng PacBio sa pamamagitan ng pagbibigay ng napakatumpak na long reads (HiFi) na sumasaklaw sa mga full-length na amplicon, na tumutugon sa katumpakan ng pagkakasunud-sunod ng Illumina. Ang kakayahang ito ay nagbibigay-daan sa mga mananaliksik na makamit ang isang walang kaparis na kalamangan - isang malawak na tanawin ng genetic landscape. Ang pinalawak na saklaw ay makabuluhang pinapataas ang resolusyon sa anotasyon ng mga species, lalo na sa loob ng bacterial o fungal na mga komunidad, na nagbibigay-daan sa isang mas malalim na pag-unawa sa mga intricacies ng mga populasyon ng microbial.
-

16S/18S/ITS Amplicon Sequencing-NGS
Ang pagkakasunud-sunod ng Amplicon gamit ang teknolohiyang Illumina, partikular na nagta-target sa mga genetic na marker ng 16S, 18S, at ITS, ay isang mahusay na paraan para sa pag-alis ng phylogeny, taxonomy, at kasaganaan ng mga species sa loob ng mga microbial na komunidad. Ang diskarte na ito ay nagsasangkot ng pagkakasunud-sunod ng mga hypervariable na rehiyon ng housekeeping genetic marker. Orihinal na ipinakilala bilang molecular fingerprint niWoeses et alnoong 1977, binago ng diskarteng ito ang microbiome profiling sa pamamagitan ng pagpapagana ng mga pagsusuri na walang isolation. Sa pamamagitan ng pagkakasunud-sunod ng 16S (bacteria), 18S (fungi), at Internal Transcribed Spacer (ITS, fungi), matutukoy ng mga mananaliksik hindi lamang ang masaganang species kundi pati na rin ang mga bihira at hindi pa nakikilala. Malawakang pinagtibay bilang isang mahalagang tool, naging instrumento ang pagkakasunud-sunod ng amplicon sa pagtukoy sa pagkakaiba-iba ng mga komposisyon ng microbial sa magkakaibang kapaligiran, kabilang ang bibig ng tao, bituka, dumi, at higit pa.
-

Bacterial at Fungal Whole Genome Re-Sequencing
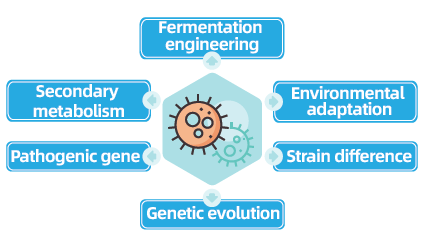
Ang bacterial at fungal whole-genome re-sequencing projects ay mahalaga para sa pagsulong ng microbial genomics sa pamamagitan ng pagpapagana ng pagkumpleto at paghahambing ng microbial genome. Pinapadali nito ang fermentation engineering, ang pag-optimize ng mga prosesong pang-industriya, at ang paggalugad ng pangalawang metabolismo. Higit pa rito, mahalaga ang fungal at bacterial re-sequencing para sa pag-unawa sa adaptasyon sa kapaligiran, pag-optimize ng mga strain, at pagpapakita ng genetic evolution dynamics, na may malawak na implikasyon sa medisina, agrikultura, at environmental science.
-

PacBio-Full-length 16S/18S/ITS Amplicon Sequencing
Ang platform ng Amplicon (16S/18S/ITS) ay binuo na may mga taon ng karanasan sa pagsusuri ng proyekto ng microbial diversity, na naglalaman ng standardized basic analysis at personalized na pagsusuri: sinasaklaw ng basic analysis ang mainstream analysis content ng kasalukuyang microbial research, ang analysis content ay mayaman at komprehensibo, at ang mga resulta ng pagsusuri ay ipinakita sa anyo ng mga ulat ng proyekto; Ang nilalaman ng personalized na pagsusuri ay magkakaiba. Maaaring pumili ng mga sample at ang mga parameter ay maaaring itakda nang may kakayahang umangkop ayon sa pangunahing ulat ng pagsusuri at layunin ng pananaliksik, upang mapagtanto ang mga personalized na pangangailangan. Windows operating system, simple at mabilis.
-

PacBio-Full-length Transcriptome (Non-Reference)
Ang pagkuha ng Pacific Biosciences (PacBio) Isoform sequencing data bilang input, nagagawa ng App na ito na tukuyin ang mga full-length na transcript sequence (nang walang assembly). Sa pamamagitan ng pagmamapa ng mga full-length na sequence laban sa reference na genome, ang mga transcript ay maaaring i-optimize ng mga kilalang gene, transcript, coding region, atbp. Sa kasong ito, mas tumpak na pagkakakilanlan ng mga istruktura ng mRNA, tulad ng alternatibong splicing, atbp, ay maaaring makamit. Ang pinagsamang pagsusuri sa NGS transcriptome sequencing data ay nagbibigay-daan sa mas komprehensibong annotation at mas tumpak na quantification sa expression sa transcript level, na higit na nakikinabang sa downstream differential expression at functional analysis.
-

Pinababang Representasyon ng Bisulfite Sequencing (RRBS)
Ang Reduced Representation Bisulfite Sequencing (RRBS) ay lumitaw bilang isang cost-effective at mahusay na alternatibo sa Whole Genome Bisulfite Sequencing (WGBS) sa DNA methylation research. Habang nagbibigay ang WGBS ng mga komprehensibong insight sa pamamagitan ng pagsusuri sa buong genome sa solong base resolution, ang mataas na gastos nito ay maaaring maging isang limiting factor. Madiskarteng pinapagaan ng RRBS ang hamon na ito sa pamamagitan ng piling pagsusuri sa isang kinatawan na bahagi ng genome. Ang pamamaraang ito ay umaasa sa pagpapayaman ng mga rehiyong mayaman sa isla ng CpG sa pamamagitan ng cleavage ng MspI na sinusundan ng pagpili ng laki ng mga fragment na 200-500/600 bps. Dahil dito, ang mga rehiyon lamang na malapit sa mga isla ng CpG ang pinagsunod-sunod, habang ang mga may malalayong isla ng CpG ay hindi kasama sa pagsusuri. Ang prosesong ito, na sinamahan ng bisulfite sequencing, ay nagbibigay-daan para sa high-resolution na pag-detect ng DNA methylation, at ang sequencing approach, PE150, ay partikular na nakatuon sa mga dulo ng mga insert sa halip na sa gitna, na nagpapataas ng kahusayan ng methylation profiling. Ang RRBS ay isang napakahalagang tool na nagbibigay-daan sa cost-effective na DNA methylation research at nagpapaunlad ng kaalaman sa mga mekanismo ng epigenetic.
-

Prokaryotic RNA Sequencing
Ang RNA sequencing ay nagbibigay kapangyarihan sa komprehensibong pag-profile ng lahat ng RNA transcript sa loob ng mga cell sa ilalim ng mga partikular na kundisyon. Ang makabagong teknolohiyang ito ay nagsisilbing isang makapangyarihang kasangkapan, na naglalahad ng masalimuot na mga profile ng expression ng gene, mga istruktura ng gene, at mga mekanismo ng molekular na nauugnay sa magkakaibang biological na proseso. Malawakang pinagtibay sa pangunahing pananaliksik, klinikal na diagnostic, at pagpapaunlad ng gamot, ang RNA sequencing ay nag-aalok ng mga insight sa mga intricacies ng cellular dynamics at genetic regulation. Ang aming prokaryotic RNA sample processing ay iniakma para sa prokaryotic transcriptomes, na kinasasangkutan ng rRNA depletion at directional library preparation.
Platform: Illumina NovaSeq
-

Metatranscriptome Sequencing
Ang paggamit ng teknolohiya sa pagkakasunud-sunod ng Illumina, ang metatranscriptome sequencing service ng BMKGENE ay nagpapakita ng dynamic na gene expression ng isang magkakaibang hanay ng mga mikrobyo, na sumasaklaw sa mga eukaryote hanggang sa mga prokaryote at mga virus, sa loob ng mga natural na kapaligiran tulad ng lupa, tubig, dagat, dumi, at bituka. Ang aming komprehensibong serbisyo ay nagbibigay ng kapangyarihan sa mga mananaliksik na suriin ang kumpletong mga profile ng gene expression ng mga kumplikadong microbial na komunidad. Higit pa sa pagsusuri ng taxonomic, pinapadali ng aming serbisyo ng metatranscriptome na pagkakasunud-sunod ang paggalugad sa functional enrichment, na nagbibigay-liwanag sa mga gene na naiibang ipinahayag at ang kanilang mga tungkulin. Tumuklas ng napakaraming biological na insight habang nagna-navigate ka sa mga kumplikadong landscape ng gene expression, taxonomic diversity, at functional dynamics sa loob ng magkakaibang environmental niche na ito.
-

De novo Fungal Genome Assembly
Nag-aalok ang BMKGENE ng maraming nalalaman na solusyon para sa mga fungal genome, na tumutugon sa magkakaibang mga pangangailangan sa pananaliksik at ninanais na pagkakumpleto ng genome. Ang paggamit lamang ng maikling-read na pagkakasunud-sunod ng Illumina ay nagbibigay-daan sa pagbuo ng isang draft na genome. Ang mga short-read at long-read na pagkakasunud-sunod gamit ang Nanopore o Pacbio ay pinagsama para sa isang mas pinong fungal genome na may mas mahabang contigs. Bukod dito, ang pagsasama ng Hi-C sequencing ay higit na nagpapahusay sa mga kakayahan, na nagpapagana sa pagkamit ng isang kumpletong chromosome-level genome.