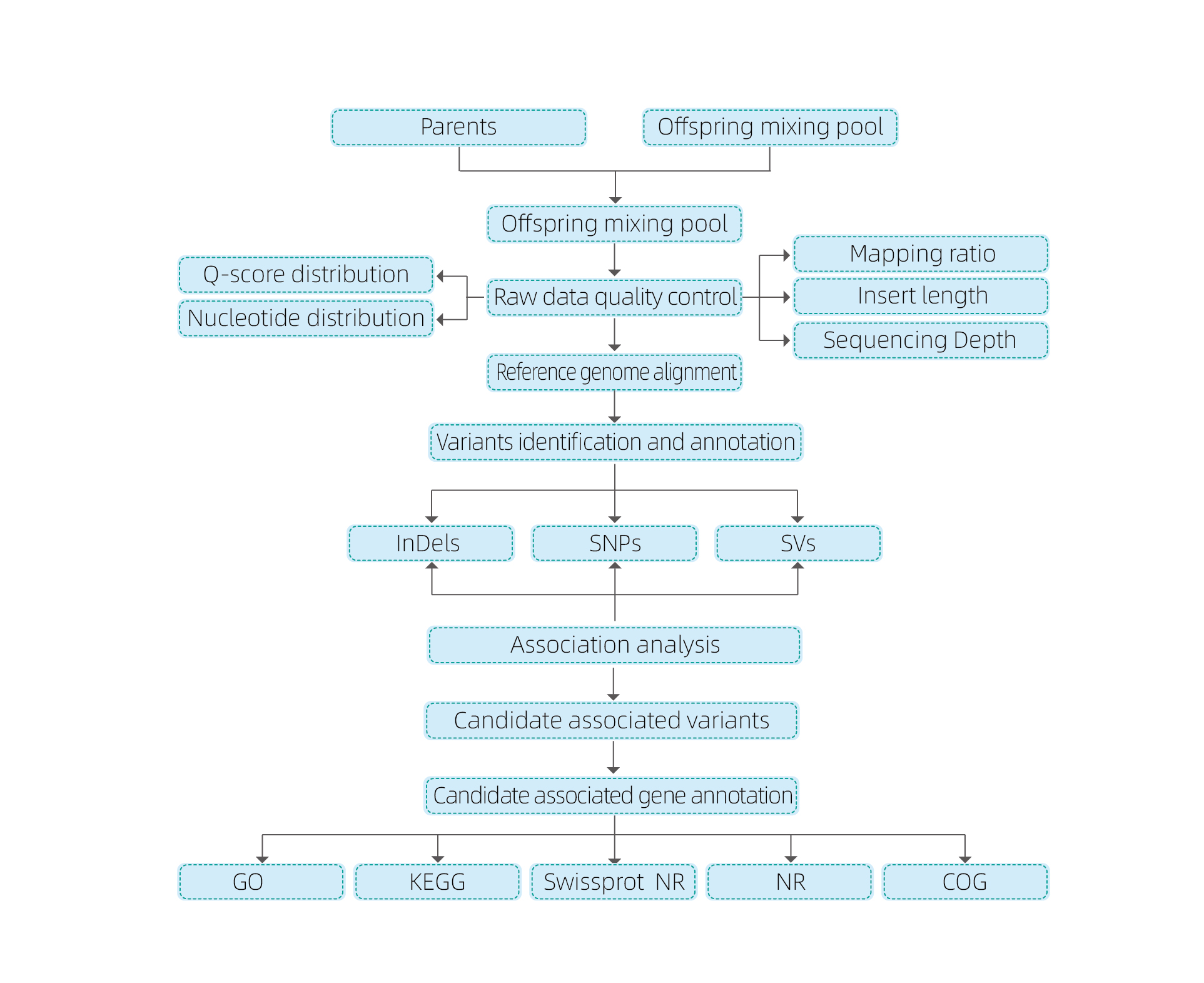
Bulked segregant analysis
Mga kalamangan sa serbisyo

Takagi et al., The Plant Journal, 2013
● Tumpak na lokalisasyon: Paghahalo ng mga bulks na may 30+30 hanggang 200+200 mga indibidwal upang mabawasan ang ingay sa background; Ang hula na hindi naka-synonymous na mutatants na batay sa kandidato.
● Comprehensive analysis: malalim na anotasyon ng function ng kandidato ng kandidato, kabilang ang NR, Swissprot, GO, KEGG, COG, KOG, atbp.
● Mas mabilis na oras ng pag -ikot: mabilis na lokalisasyon ng gene sa loob ng 45 araw ng pagtatrabaho.
● Malawak na karanasan: Ang BMK ay nag -ambag sa libu -libong mga pag -localize ng mga katangian, na sumasakop sa magkakaibang mga species tulad ng mga pananim, mga produktong aquatic, kagubatan, bulaklak, prutas, atbp.
Mga pagtutukoy sa serbisyo
Populasyon:
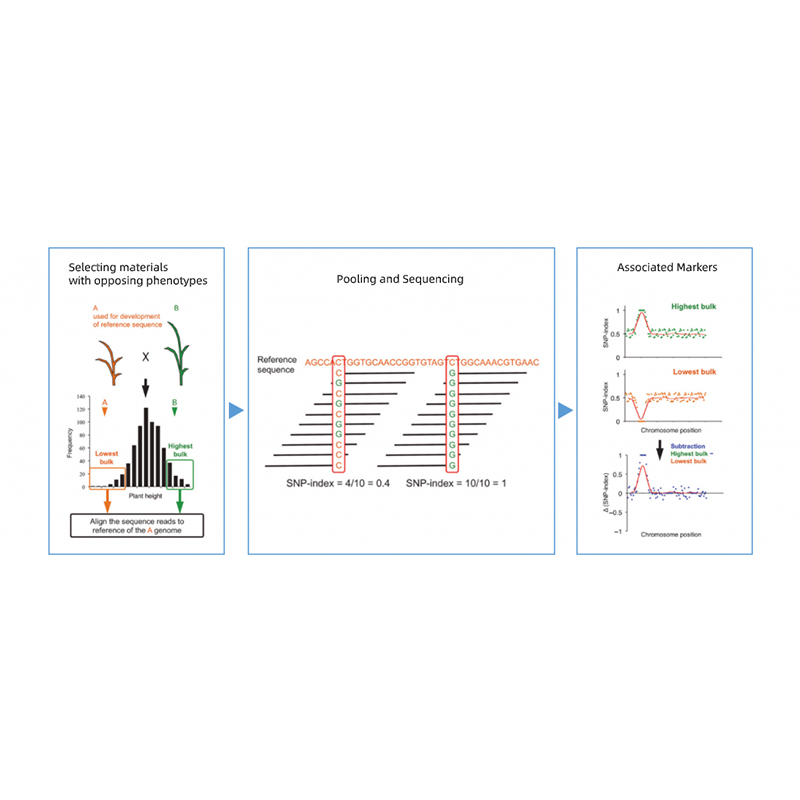
Paghiwalayin ang progeny ng mga magulang na may magkasalungat na mga phenotypes.
EG F2 Progeny, Backcrossing (BC), Recombinant Inbred Line (RIL)
Paghahalo ng pool
Para sa mga kwalipikadong katangian: 30 hanggang 50 indibidwal (minimum 20)/bulk
Para sa dami ng tratis: Nangungunang 5% hanggang 10% na mga indibidwal na may alinman sa matinding phenotypes sa buong populasyon (minimum na 30+30).
Inirerekumenda na lalim ng pagkakasunud -sunod
Hindi bababa sa 20x/magulang at 1x/supling indibidwal (hal. Para sa mga supling na paghahalo ng pool na 30+30 indibidwal, ang lalim ng pagkakasunud -sunod ay magiging 30x bawat bulkan)
Pag -aaral ng Bioinformatics
● Buong genome resequencing
● Pagproseso ng data
● SNP/Indel Calling
● Pag -screening ng Rehiyon ng Kandidato
● Annotation ng Function ng Gene ng Kandidato

Halimbawang mga kinakailangan at paghahatid
Mga kinakailangan sa halimbawang:
Nucleotides:
| GDNA Sample | Sample ng tisyu |
| Konsentrasyon: ≥30 ng/μl | Halaman: 1-2 g |
| Halaga: ≥2 μg (volumn ≥15 μl) | Mga Hayop: 0.5-1 g |
| Purity: OD260/280 = 1.6-2.5 | Buong dugo: 1.5 ml |
Daloy ng trabaho sa serbisyo

Disenyo ng eksperimento

Halimbawang paghahatid

Pagkuha ng RNA

Konstruksyon ng Library

Pagkakasunud -sunod

Pagtatasa ng data

Mga serbisyo pagkatapos ng pagbebenta
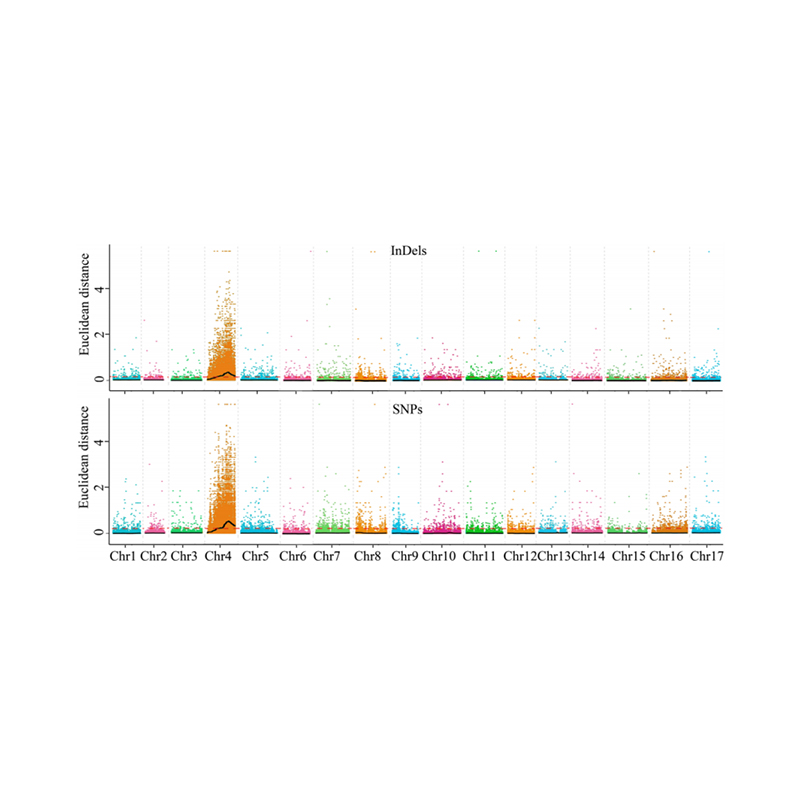
1.Association analysis base sa Euclidean Distance (ED) upang makilala ang rehiyon ng kandidato. Sa sumusunod na figure
X-axis: chromosome number; Ang bawat tuldok ay kumakatawan sa isang halaga ng ED ng isang SNP. Ang itim na linya ay tumutugma sa karapat -dapat na halaga ng ED. Ang isang mas mataas na halaga ng ED ay nagpapahiwatig ng isang mas makabuluhang kaugnayan sa pagitan ng site at ng phenotype. Ang Red Dash Line ay kumakatawan sa threshold ng makabuluhang samahan.

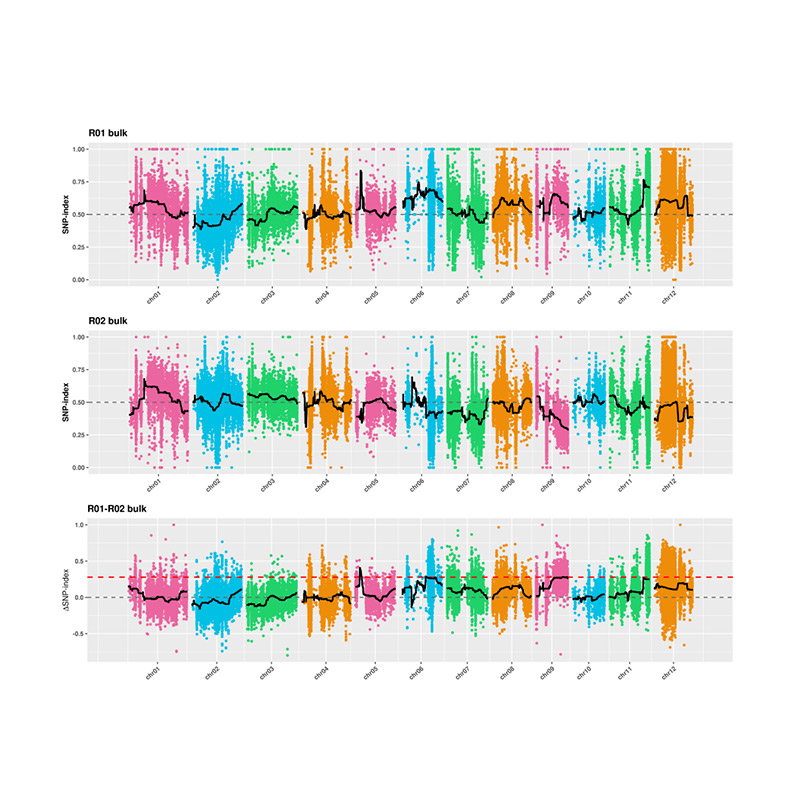
2.Association analysis batay sa walang SNP-index
X-axis: chromosome number; Ang bawat tuldok ay kumakatawan sa halaga ng SNP-index. Ang itim na linya ay nakatayo para sa karapat-dapat na halaga ng SNP-index. Ang mas malaki ang halaga ay, mas makabuluhan ang samahan.

Kaso ng BMK
Ang major-effect na dami ng trait locus fnl7.1 ay nag-encode ng isang huli na embryogenesis na masaganang protina na nauugnay sa haba ng leeg ng prutas sa pipino
Nai -publish: Plant Biotechnology Journal, 2020
Diskarte sa pagkakasunud -sunod:
Mga magulang (Jin5-508, YN): buong genome resequencing para sa 34 × at 20 ×.
DNA Pools (50 Long-Necked at 50 Short-necked): Resequencing para sa 61 × at 52 ×
Mga pangunahing resulta
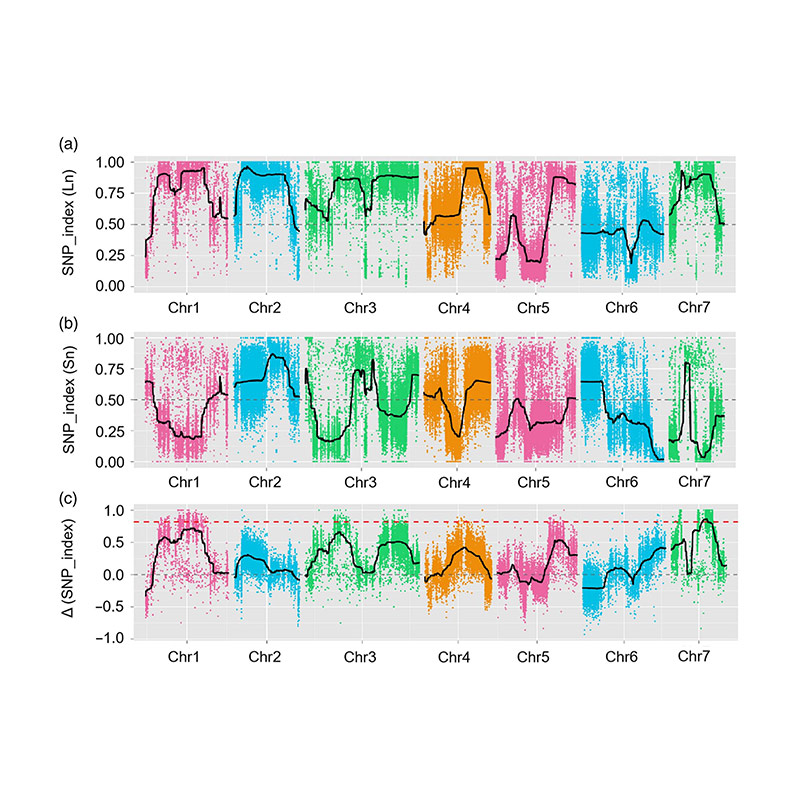
Sa pag-aaral na ito, ang paghihiwalay ng populasyon (F2 at F2: 3) ay nabuo sa pamamagitan ng pagtawid sa long-neck na linya ng pipino JIN5-508 at short-neck yn. Dalawang pool ng DNA ang itinayo ng 50 matinding mga indibidwal na pang-leeg at 50 matinding mga indibidwal na may maikling leeg. Ang Major-effect QTL ay nakilala sa CHR07 ng pagsusuri ng BSA at tradisyonal na pagmamapa ng QTL. Ang rehiyon ng kandidato ay karagdagang paliitin sa pamamagitan ng pinong pag-mapa, dami ng expression expression at transgenic na mga eksperimento, na nagpahayag ng pangunahing gene sa pagkontrol sa leeg-haba, CSFNL7.1. Bilang karagdagan, ang polymorphism sa CSFNL7.1 na tagataguyod ng rehiyon ay natagpuan na nauugnay sa kaukulang expression. Ang karagdagang pagsusuri ng phylogenetic ay iminungkahi na ang FNL7.1 locus ay malamang na nagmula sa India.
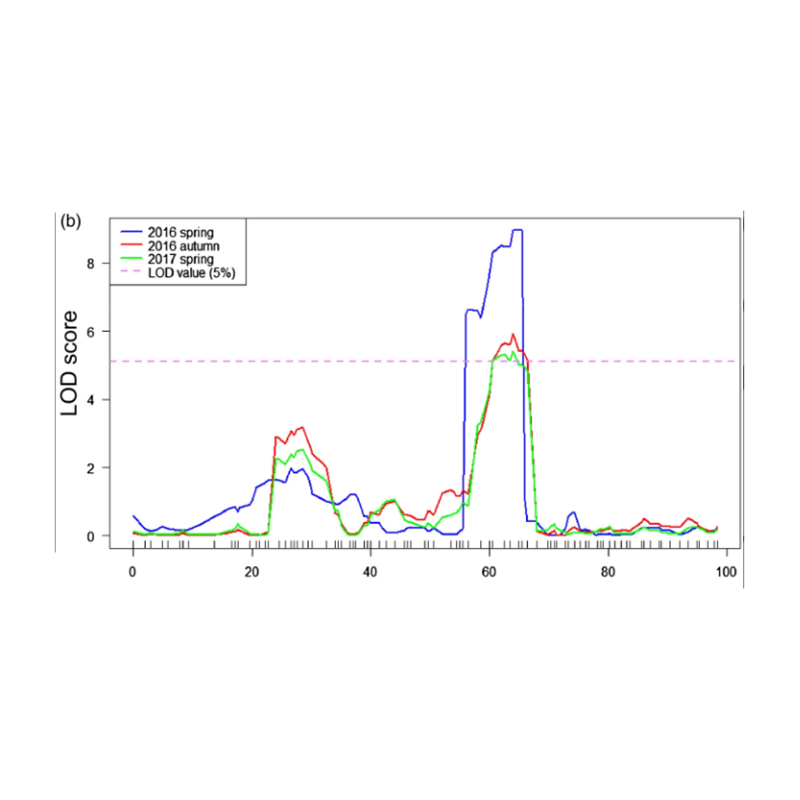
 QTL-Mapping sa BSA Pagsusuri upang makilala ang Rehiyon ng Kandidato na nauugnay sa haba ng Neck ng Pipino |  Ang mga profile ng LOD ng cucumber leeg-haba na QTL na nakilala sa CHR07 |
Xu, X., et al. "Ang pangunahing epekto ng dami ng lokus na FNL7.1 ay nag-encode ng isang huli na embryogenesis na masaganang protina na nauugnay sa haba ng leeg ng prutas sa pipino." Plant Biotechnology Journal 18.7 (2020).