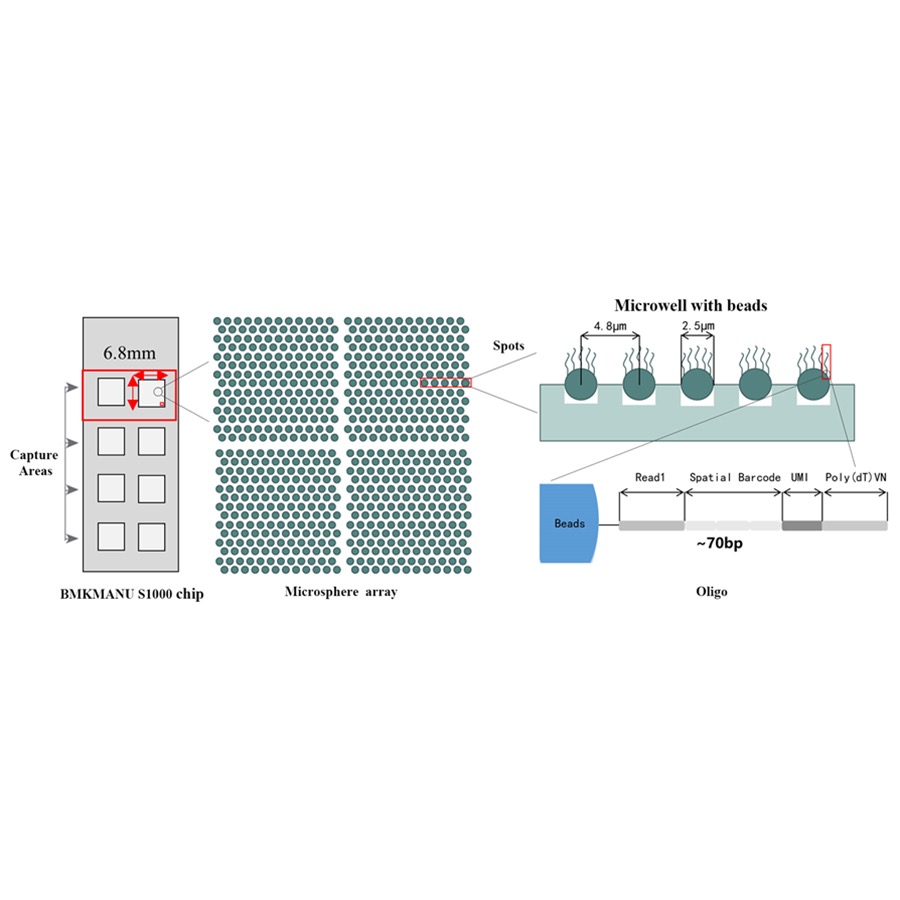
BMKMANU S1000 Spatial Transcriptome
BMKMANU S1000 Spatial Transcriptome Technical Scheme
Mga tampok
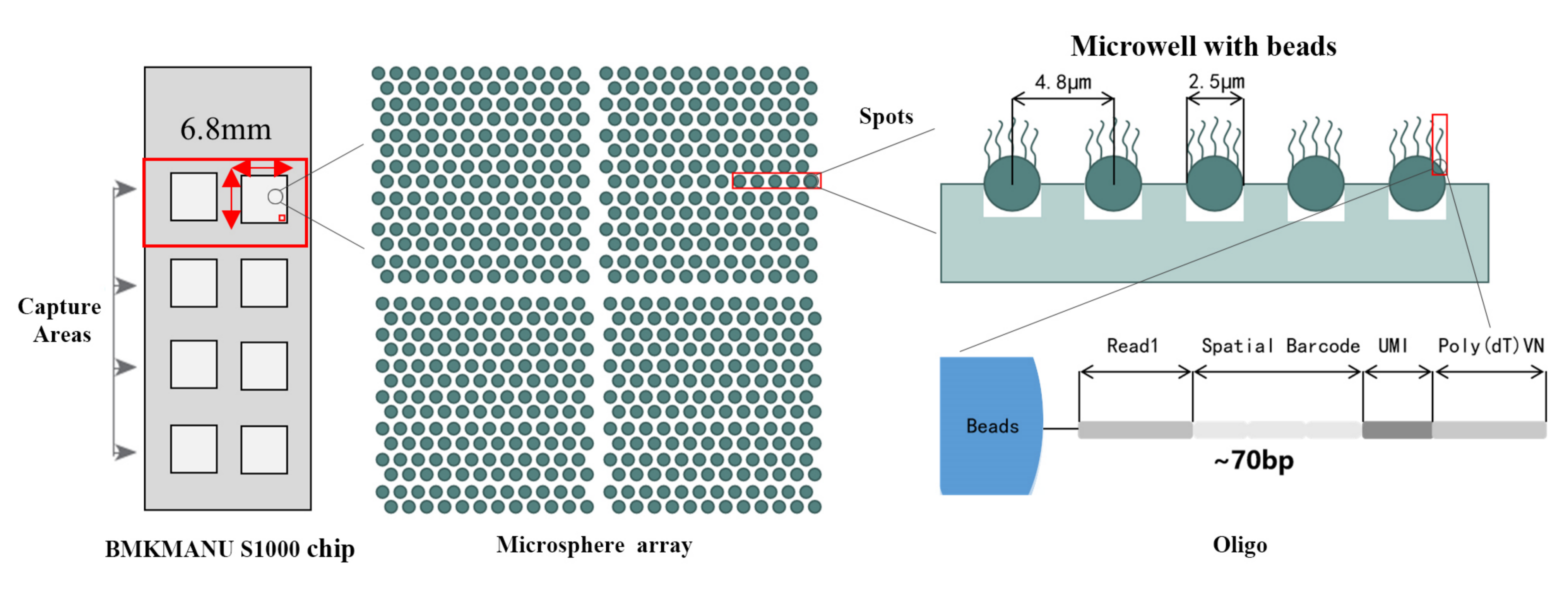
● Resolution: 5 µM
● Spot Diameter: 2.5 µM
● Bilang ng mga spot: humigit-kumulang 2 Milyon
● 3 posibleng mga format ng capture area: 6.8 mm * 6.8 mm, 11 mm * 11 mm o 15 mm * 20 mm
● Ang bawat barcoded bead ay puno ng mga panimulang aklat na binubuo ng 4 na seksyon:
poly(dT) tail para sa mRNA priming at cDNA synthesis
Natatanging Molecular Identifier (UMI) para itama ang bias ng amplification
Spatial na barcode
Binding sequence ng partial read 1 sequencing primer
● H&E at fluorescent staining ng mga seksyon
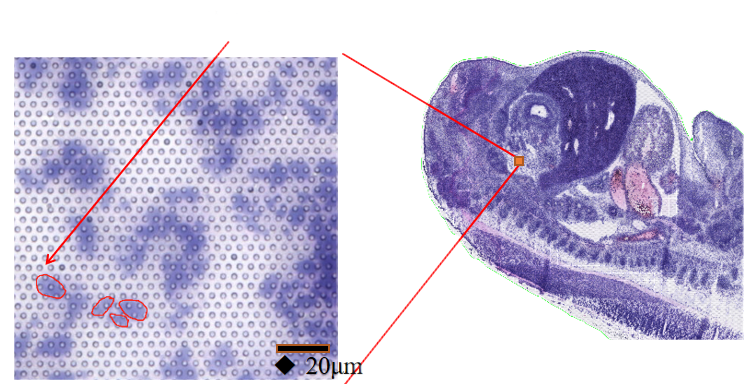
● Posibilidad na gamitinteknolohiya ng cell segmentation: integration ng H&E staining, fluorescent staining, at RNA sequencing para matukoy ang mga hangganan ng bawat cell at wastong magtalaga ng gene expression sa bawat cell.
Mga kalamangan ng BMKMANU S1000
●Sub-cellular na Resolusyon: Ang bawat lugar ng pagkuha ay naglalaman ng >2 milyong spatial na Barcoded Spots na may diameter na 2.5 µm at isang spacing na 5 µm sa pagitan ng mga spot center, na nagpapagana ng spatial transcriptome analysis na may sub-cellular resolution (5 µm).
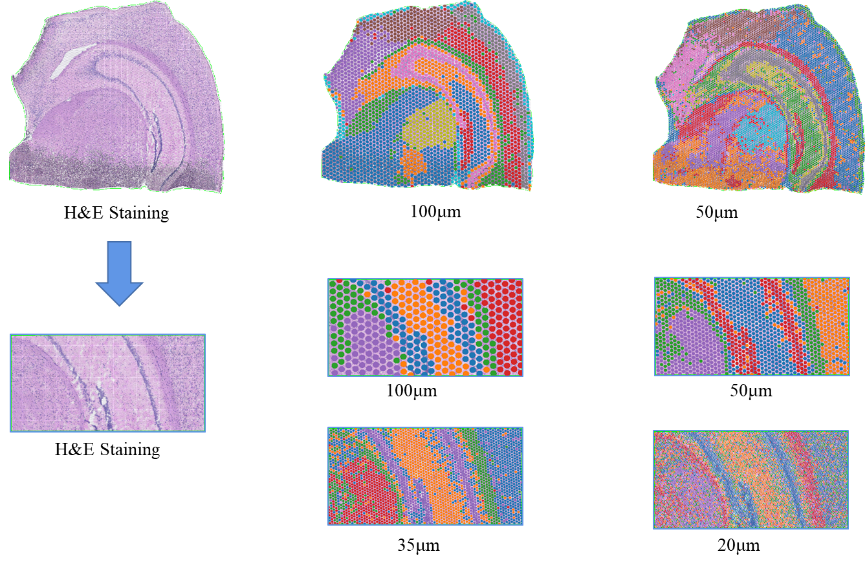
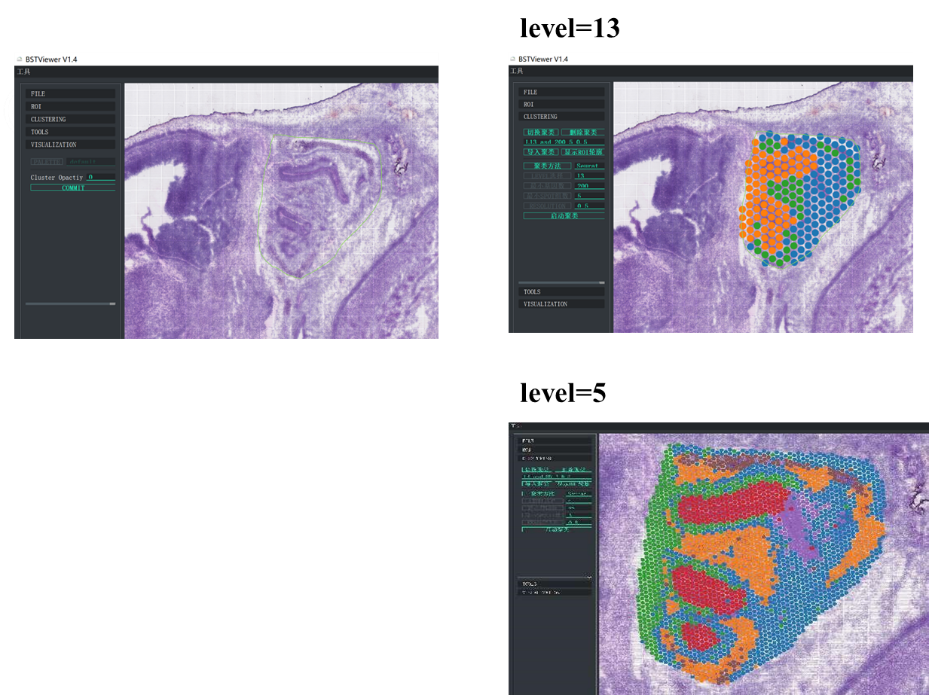
●Multi-level na Resolution Analysis:Flexible na multi-level na pagsusuri mula 100 μm hanggang 5 μm upang malutas ang magkakaibang mga tampok ng tissue sa pinakamainam na resolusyon.
● Posibilidad na Gamitin ang "Tatlo sa isang slide" na Teknolohiya ng Pagse-segment ng Cell:Pinagsasama ang fluorescence staining, H&E staining, at RNA sequencing sa isang slide, binibigyang kapangyarihan ng aming "three-in-one" analysis algorithm ang pagtukoy ng mga hangganan ng cell para sa mga kasunod na transcriptomic na nakabatay sa cell.
●Tugma sa Maramihang Mga Platform ng Pagkakasunud-sunod: Parehong available ang NGS at long-read sequencing.
●Flexible na Disenyo ng 1-8 Active Capture Area: Ang laki ng lugar ng pagkuha ay nababaluktot, na posibleng gumamit ng 3 format (6.8 mm * 6.8 mm., 11 mm * 11 mm at 15 mm * 20 mm)
●One-stop na Serbisyo: Pinagsasama nito ang lahat ng karanasan at mga hakbang na nakabatay sa kasanayan, kabilang ang cryo-sectioning, staining, tissue optimization, spatial barcoding, paghahanda sa library, sequencing, at bioinformatics.
●Comprehensive Bioinformatics at User-friendly Visualization ng mga Resulta:Kasama sa package ang 29 na pagsusuri at 100+ mataas na kalidad na mga numero, na sinamahan ng paggamit ng inhouse na binuong software upang mailarawan at i-customize ang cell splitting at spot clustering.
●Customized Data Analysis at Visualization: magagamit para sa iba't ibang kahilingan sa pananaliksik
●Highly-skilled Technical Team: may karanasan sa mahigit 250 uri ng tissue at 100+ species kabilang ang tao, daga, mammal, isda at halaman.
●Mga Real-time na Update sa Buong Proyekto: na may ganap na kontrol sa eksperimentong pag-unlad.
●Opsyonal na Pinagsamang Pagsusuri na may Single-cell mRNA Sequencing
Mga Detalye ng Serbisyo
|
Sampol Mga kinakailangan
| Aklatan |
Diskarte sa pagkakasunud-sunod
| Inirerekomenda ang data | Kontrol sa Kalidad |
| Mga sample ng cryo na naka-embed sa OCT, 3 bloke bawat sample | S1000 cDNA library | Illumina PE150 (magagamit ang iba pang mga platform) | 100K PE reads bawat 100 uM ( 60-150 Gb ) | RIN>7 |
Para sa higit pang mga detalye sa gabay sa paghahanda ng sample at daloy ng trabaho sa serbisyo, mangyaring huwag mag-atubiling makipag-usap sa aDalubhasa sa BMKGENE
Daloy ng Trabaho ng Serbisyo
Sa yugto ng paghahanda ng sample, isinasagawa ang isang paunang pagsubok sa pagkuha ng bulk RNA upang matiyak na makakakuha ng mataas na kalidad na RNA. Sa yugto ng pag-optimize ng tissue ang mga seksyon ay nabahiran at nakikita at ang mga kondisyon ng permeabilization para sa paglabas ng mRNA mula sa tissue ay na-optimize. Ang na-optimize na protocol ay inilalapat sa panahon ng pagtatayo ng library, na sinusundan ng pagkakasunud-sunod at pagsusuri ng data.
Ang kumpletong daloy ng trabaho ng serbisyo ay nagsasangkot ng mga real-time na pag-update at pagkumpirma ng kliyente upang mapanatili ang isang tumutugon na loop ng feedback, na tinitiyak ang maayos na pagpapatupad ng proyekto.
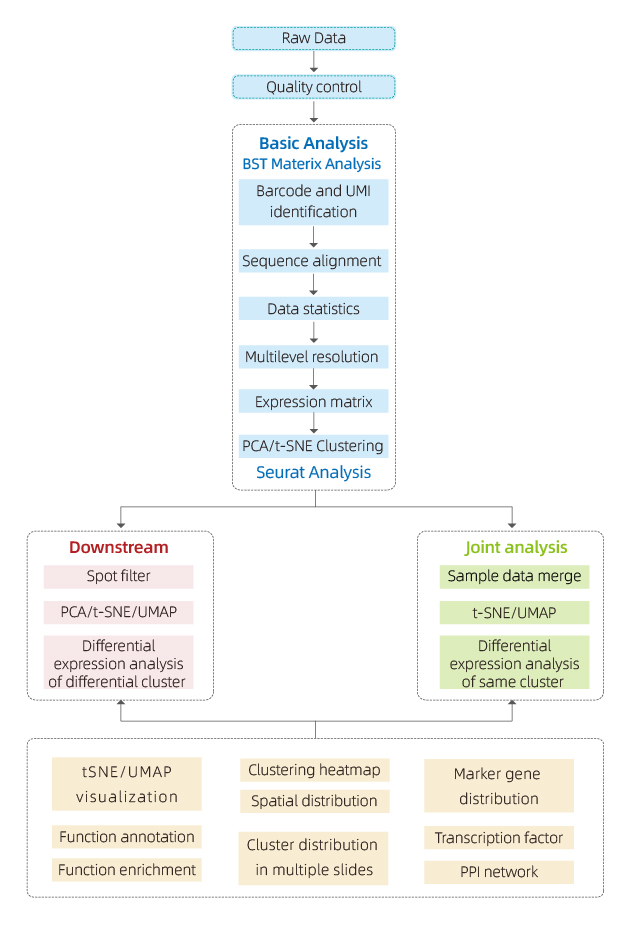
Ang data na nabuo ng BMKMANU S1000 ay sinusuri gamit ang software na "BSTMatrix", na independiyenteng idinisenyo ng BMKGENE, na bumubuo ng Gene Expression Matrix. Mula doon, nabuo ang isang karaniwang ulat na kinabibilangan ng kontrol sa kalidad ng data, pagsusuri sa panloob na sample at pagsusuri sa pagitan ng pangkat.
● Kontrol sa Kalidad ng Data:
Output ng data at pamamahagi ng marka ng kalidad
Pag-detect ng gene bawat lugar
Saklaw ng tissue
● Inner-sample analysis:
Kayamanan ng gene
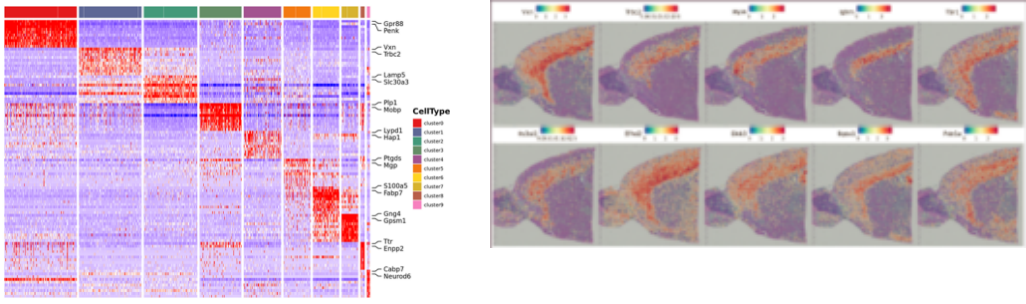
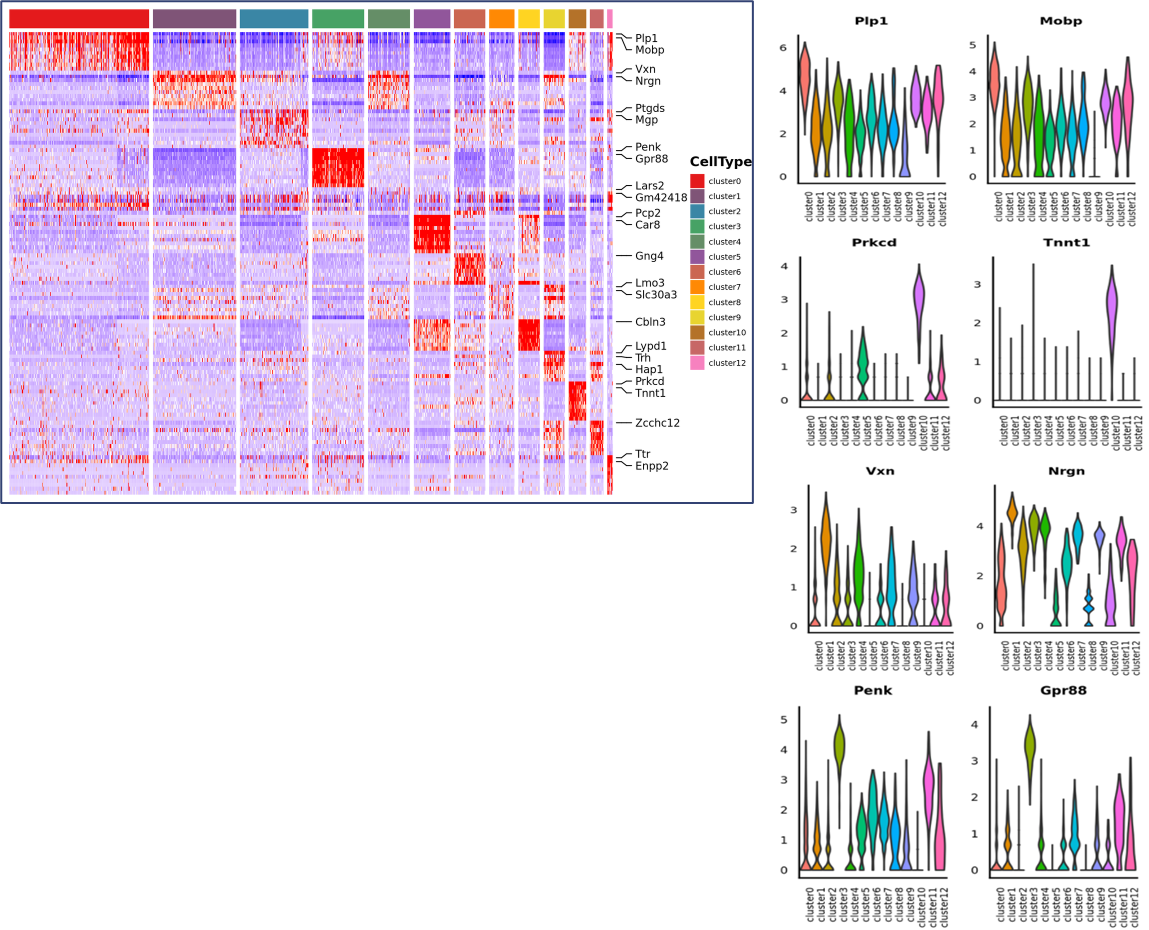
Spot clustering, kabilang ang pinababang pagsusuri ng dimensyon
Pagsusuri ng pagkakaiba-iba ng expression sa pagitan ng mga kumpol: pagkilala sa mga gene ng marker
Functional na anotasyon at pagpapayaman ng mga marker gene
● Pagsusuri sa pagitan ng pangkat:
Muling kumbinasyon ng mga batik mula sa parehong mga sample (hal. may sakit at kontrol) at muling kumpol
Pagkilala ng mga marker gene para sa bawat kumpol
Functional na anotasyon at pagpapayaman ng mga marker gene
Differential Expression ng parehong cluster sa pagitan ng mga grupo
Bukod pa rito, binuo ng BMKGENE ang "BSTViewer" ay isang tool na madaling gamitin na nagbibigay-daan sa user na makita ang gene expression at spot clustering sa iba't ibang mga resolution.
Ang BMKGene ay bumuo ng software para sa user friendly na visualization
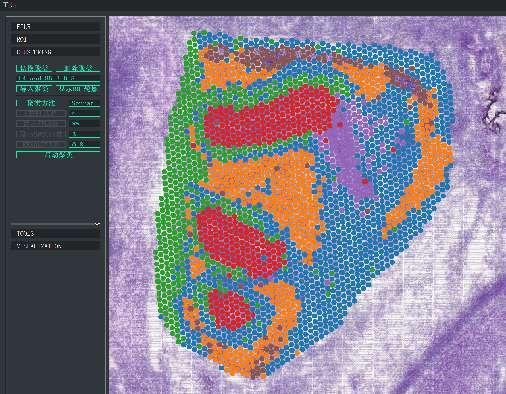
BSTViewer spot clustering sa multi-level na resolution
BSTellViewer: awtomatiko at manu-manong paghahati ng cell
Panloob na sample na Pagsusuri
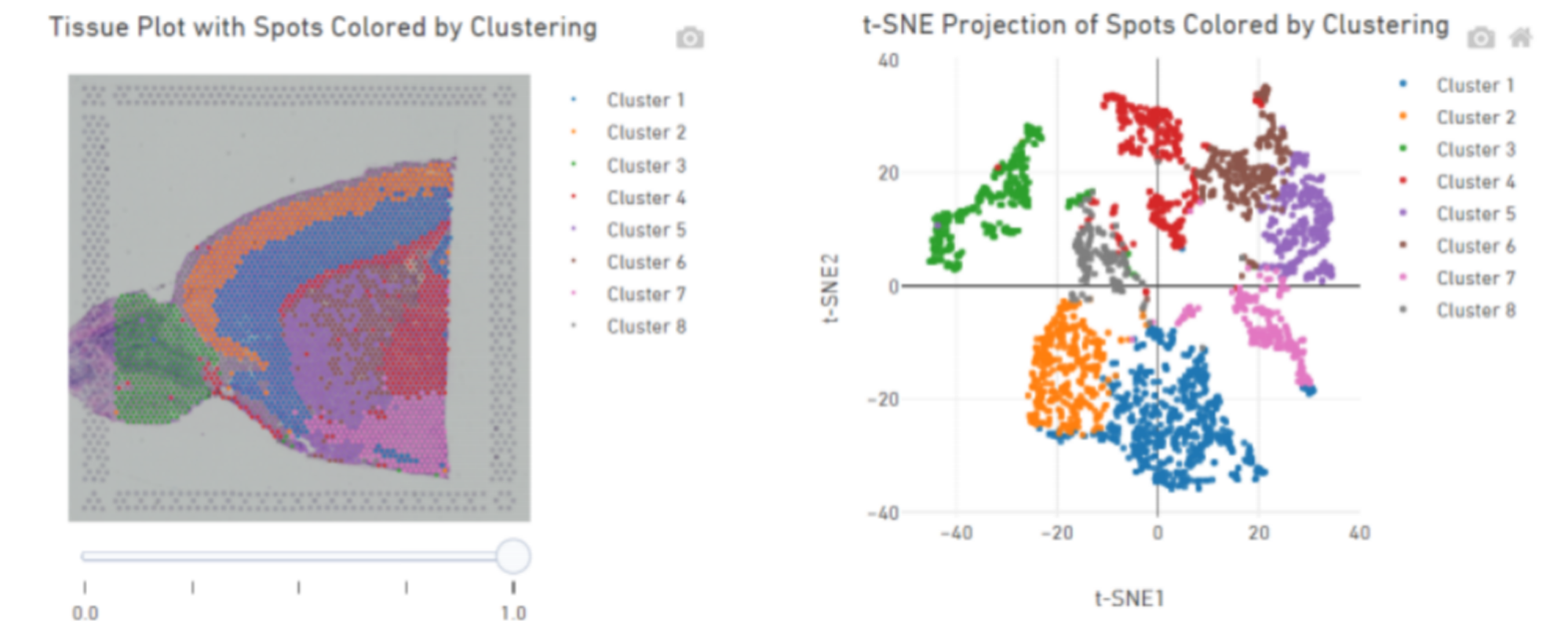
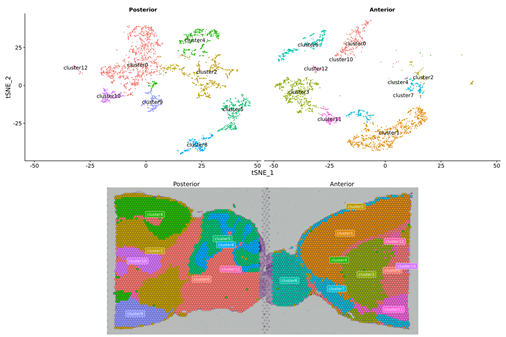
Spot clustering:
Pagkilala sa mga gene ng marker at spatial na pamamahagi:
Pagsusuri sa pagitan ng pangkat
Kumbinasyon ng data mula sa parehong pangkat at re-cluster:
Mga marker na gene ng mga bagong kumpol:
Galugarin ang mga pagsulong na pinadali ng mga serbisyo ng spatial transcriptomics ng BMKGene gamit ang teknolohiyang BMKManu S1000 sa itinatampok na publikasyong ito:
Awit, X. et al. (2023) 'Ipinakikita ng spatial transcriptomics ang light-induced chlorenchyma cells na kasangkot sa pagtataguyod ng shoot regeneration sa tomato callus',Mga Pamamaraan ng National Academy of Sciences ng United States of America, 120(38), p. e2310163120. doi: 10.1073/pnas.2310163120
Ikaw, Y. et al. (2023) 'Systematic na paghahambing ng sequencing-based spatial transcriptomic na pamamaraan',bioRxiv, p. 2023.12.03.569744. doi: 10.1101/2023.12.03.569744.