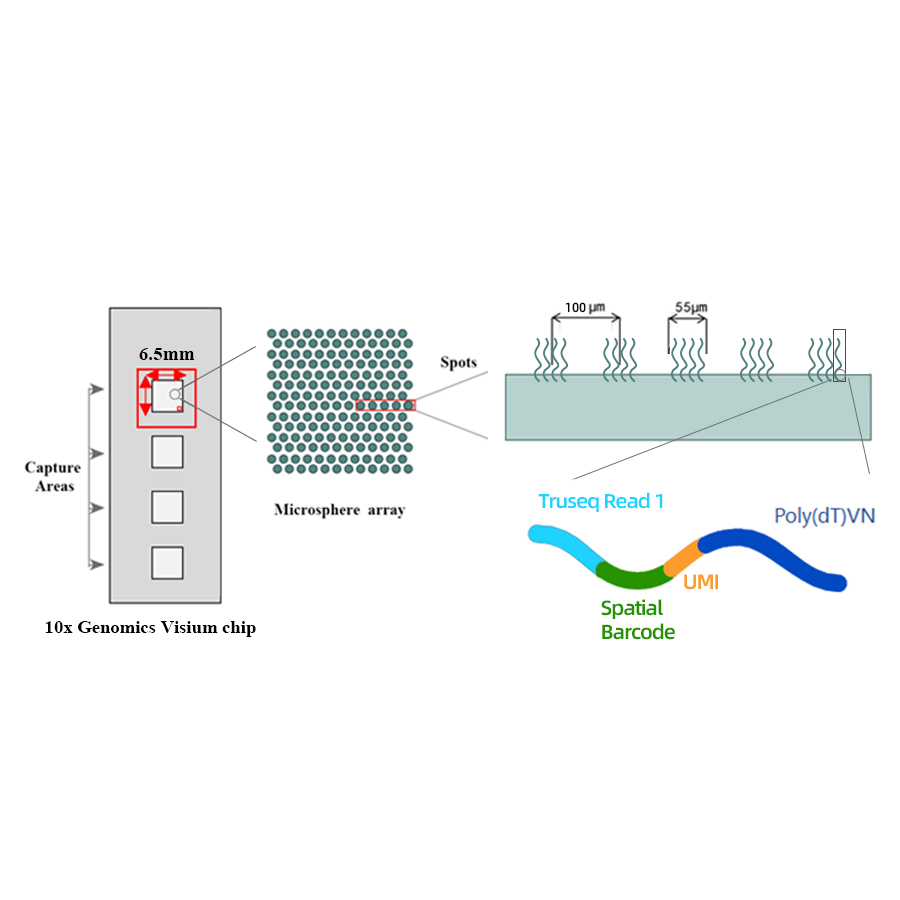
10x Genomics Visium Spatial Transcriptome
Teknikal na Scheme

Mga tampok
● Resolusyon: 100 µM
● Spot Diameter: 55 µM
● Bilang ng mga spot: 4992
● Lugar ng pagkuha: 6.5 x 6.5 mm
● Ang bawat barcoded spot ay puno ng mga panimulang aklat na binubuo ng 4 na seksyon:
- poly(dT) tail para sa mRNA priming at cDNA synthesis
- Natatanging Molecular Identifier (UMI) upang itama ang bias ng amplification
- Spatial na barcode
- Binding sequence ng partial read 1 sequencing primer
● Paglamlam ng H&E ng mga seksyon
Mga kalamangan
●One-stop na Serbisyo: isinasama ang lahat ng karanasan at mga hakbang na nakabatay sa kasanayan, kabilang ang cryo-sectioning, paglamlam, tissue optimization, spatial barcoding, paghahanda sa library, sequencing at bioinformatics .
● Highly-skilled Technical Team: may karanasan sa mahigit 250 uri ng tissue at 100+ species kabilang ang tao, daga, mammal, isda at halaman.
●Real-time na Update sa Buong Proyekto: na may ganap na kontrol sa eksperimentong pag-unlad.
●Comprehensive Standard Bioinformatics:Kasama sa package ang 29 na pagsusuri at 100+ mataas na kalidad na mga numero.
●Customized Data Analysis at Visualization: magagamit para sa iba't ibang kahilingan sa pananaliksik.
●Opsyonal na Pinagsamang Pagsusuri na may Single-cell mRNA Sequencing
Mga pagtutukoy
| Mga Sample na Kinakailangan | Library | Diskarte sa pagkakasunud-sunod | Inirerekomenda ang data | Kontrol sa Kalidad |
| Mga sample ng cryo na naka-embed sa OCT (Optimal na diameter: humigit-kumulang 6x6x6 mm³) 2 bloke bawat sample | 10X Visium cDNA library | Illumina PE150 | 50K PE reads bawat lugar ( 60Gb ) | RIN > 7 |
Para sa higit pang mga detalye sa gabay sa paghahanda ng sample at daloy ng trabaho sa serbisyo, mangyaring huwag mag-atubiling makipag-usap sa aDalubhasa sa BMKGENE
Daloy ng Trabaho ng Serbisyo
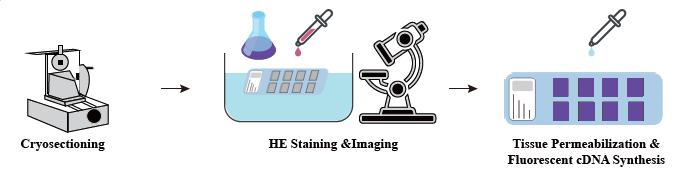
Sa yugto ng paghahanda ng sample, isinasagawa ang isang paunang pagsubok sa pagkuha ng bulk RNA upang matiyak na makakakuha ng mataas na kalidad na RNA. Sa yugto ng pag-optimize ng tissue ang mga seksyon ay nabahiran at nakikita at ang mga kondisyon ng permeabilization para sa paglabas ng mRNA mula sa tissue ay na-optimize. Ang na-optimize na protocol ay inilalapat sa panahon ng pagtatayo ng library, na sinusundan ng pagkakasunud-sunod at pagsusuri ng data.
Ang kumpletong daloy ng trabaho ng serbisyo ay nagsasangkot ng mga real-time na pag-update at pagkumpirma ng kliyente upang mapanatili ang isang tumutugon na loop ng feedback, na tinitiyak ang maayos na pagpapatupad ng proyekto.

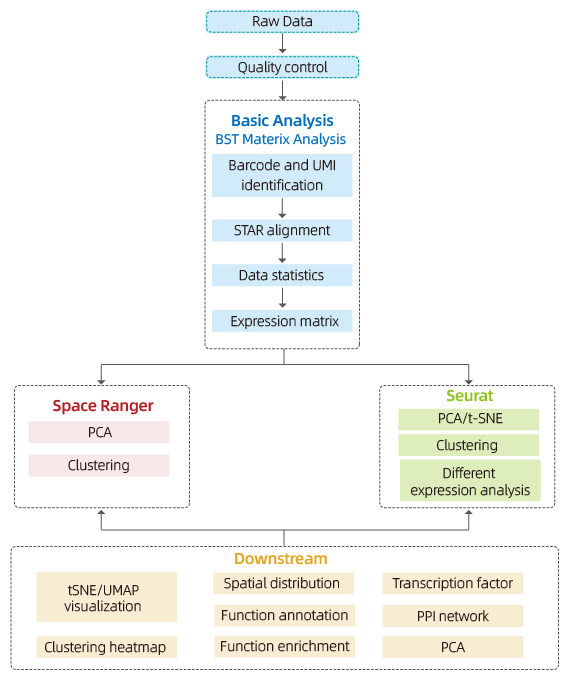
Kasama ang sumusunod na pagsusuri:
Kontrol sa Kalidad ng Data:
o Output ng data at pamamahagi ng marka ng kalidad
o Gene detection bawat lugar
o Saklaw ng tissue
Panloob na sample na Pagsusuri:
o Kayamanan ng gene
o Spot clustering, kabilang ang pinababang pagsusuri ng dimensyon
o Differential expression analysis sa pagitan ng mga cluster: pagkilala sa mga marker genes
o Functional na anotasyon at pagpapayaman ng mga marker gene
Pagsusuri sa pagitan ng pangkat
o Muling kumbinasyon ng mga batik mula sa parehong mga sample (hal. may sakit at kontrol) at muling kumpol
o Pagkilala ng mga marker gene para sa bawat kumpol
o Functional na anotasyon at pagpapayaman ng mga marker gene
o Differential Expression ng parehong cluster sa pagitan ng mga grupo
Panloob na sample na Pagsusuri
Spot clustering

Pagkilala sa mga gene ng marker at spatial na pamamahagi


Pagsusuri sa pagitan ng pangkat
Kumbinasyon ng data mula sa parehong pangkat at re-cluster
Mga marker na gene ng mga bagong kumpol
Tuklasin ang mga pagsulong na pinadali ng serbisyo ng spatial transcriptomics ng BMKGene ng 10X Visium Sa mga itinatampok na publikasyong ito:
Chen, D. et al. (2023) Ang 'mthl1, isang potensyal na Drosophila homologue ng mammalian adhesion GPCRs, ay kasangkot sa mga reaksyon ng antitumor sa mga na-injected na oncogenic na mga cell sa mga langaw',Mga Pamamaraan ng National Academy of Sciences ng United States of America, 120(30), p. e2303462120. doi: /10.1073/pnas.2303462120
Chen, Y. et al. (2023) 'STEEL ay nagbibigay-daan sa high-resolution na delineation ng spatiotemporal transcriptomic data',Mga Briefing sa Bioinformatics, 24(2), pp. 1–10. doi: 10.1093/BIB/BBAD068.
Liu, C. et al. (2022) 'Isang spatiotemporal atlas ng organogenesis sa pagbuo ng mga bulaklak ng orchid',Pananaliksik sa Nucleic Acids, 50(17), pp. 9724–9737. doi: 10.1093/NAR/GKAC773.
Wang, J. et al. (2023) 'Pagsasama-sama ng Spatial Transcriptomics at Single-nucleus RNA Sequencing ay Nagpapakita ng Potensyal na Therapeutic Strategies para sa Uterine Leiomyoma',International Journal of Biological Sciences, 19(8), pp. 2515–2530. doi: 10.7150/IJBS.83510.