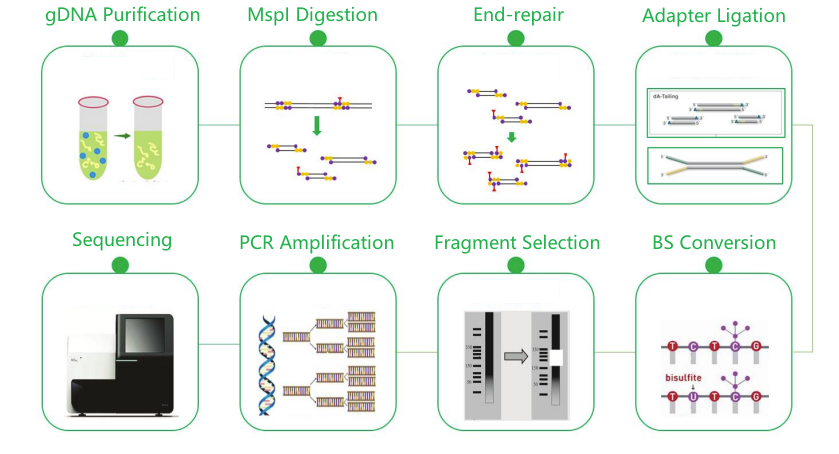
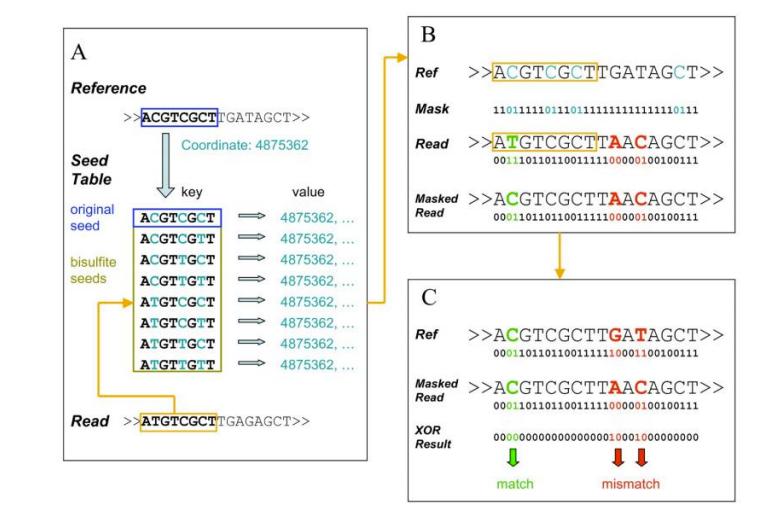
ลำดับไบซัลไฟต์ที่เป็นตัวแทนลดลง (RRBS)
คุณสมบัติการบริการ
● ต้องมีจีโนมอ้างอิง
● Lambda DNA ใช้ในการติดตามประสิทธิภาพการแปลงไบซัลไฟต์
● ประสิทธิภาพการย่อย MspI ก็ได้รับการตรวจสอบเช่นกัน
● การย่อยด้วยเอนไซม์สองเท่าสำหรับตัวอย่างพืช
● การจัดลำดับบน Illumina NovaSeq
ข้อดีของการบริการ
ทางเลือกที่คุ้มค่าและมีประสิทธิภาพสำหรับ WGBS: ทำให้สามารถดำเนินการวิเคราะห์ได้ด้วยต้นทุนที่ต่ำลงและมีความต้องการตัวอย่างที่น้อยลง
แพลตฟอร์มที่สมบูรณ์:ให้บริการที่เป็นเลิศแบบครบวงจรตั้งแต่การประมวลผลตัวอย่าง การสร้างห้องสมุด และการจัดลำดับไปจนถึงการวิเคราะห์ชีวสารสนเทศ
ความเชี่ยวชาญที่กว้างขวาง: ด้วยความสำเร็จของโครงการจัดลำดับ RRBS ในหลากหลายสายพันธุ์ BMKGENE จึงนำประสบการณ์กว่าทศวรรษ ทีมวิเคราะห์ที่มีทักษะสูง เนื้อหาที่ครอบคลุม และการสนับสนุนหลังการขายที่ยอดเยี่ยม
ข้อมูลจำเพาะของบริการ
| ห้องสมุด | กลยุทธ์การจัดลำดับ | เอาต์พุตข้อมูลที่แนะนำ | การควบคุมคุณภาพ |
| MspI ที่ถูกย่อยและไลบรารีที่ได้รับการบำบัดด้วยไบซัลไฟต์ | อิลลูมิน่า พีอี150 | 8 กิกะไบต์ | ไตรมาสที่ 30 ≥ 85% การแปลงไบซัลไฟต์ > 99% ประสิทธิภาพการตัด MspI > 95% |
ข้อกำหนดตัวอย่าง
| ความเข้มข้น (นาโนกรัม/ไมโครลิตร) | ปริมาณทั้งหมด (ไมโครกรัม) |
| |
| จีโนมดีเอ็นเอ | ≥ 30 | ≥ 1 | การย่อยสลายหรือการปนเปื้อนมีจำกัด |
ขั้นตอนการทำงานบริการ

การจัดส่งตัวอย่าง

การก่อสร้างห้องสมุด

การเรียงลำดับ

การวิเคราะห์ข้อมูล
บริการหลังการขาย
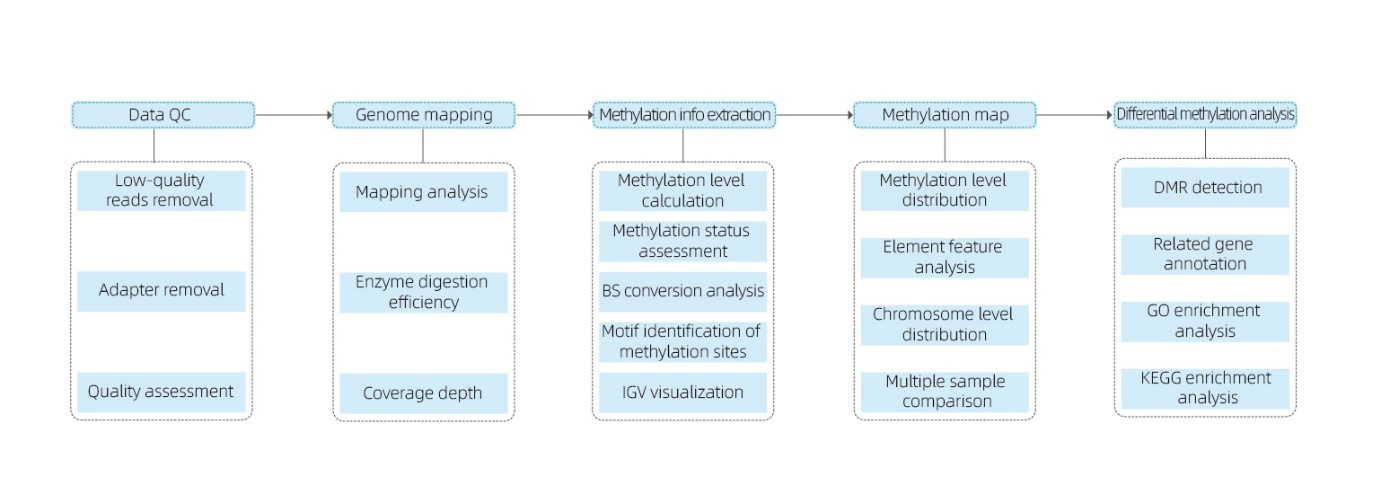
รวมถึงการวิเคราะห์ต่อไปนี้:
● การควบคุมคุณภาพลำดับดิบ
● การทำแผนที่เพื่ออ้างอิงจีโนม
● การตรวจจับฐานเมทิลเลต 5mC และการระบุมาตรฐาน
● การวิเคราะห์การกระจายตัวของเมทิลเลชั่นและการเปรียบเทียบตัวอย่าง
● การวิเคราะห์บริเวณที่มีเมทิลเลตที่แตกต่างกัน (DMR)
● คำอธิบายประกอบการทำงานของยีนที่เกี่ยวข้องกับ DMR
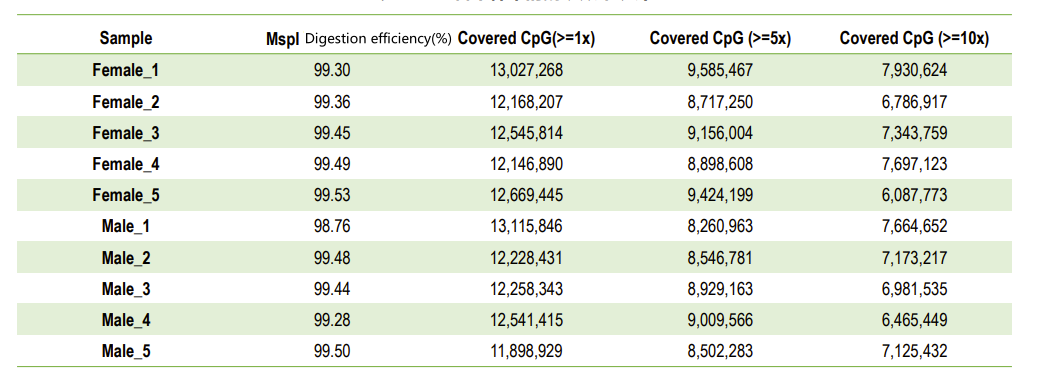
การควบคุมคุณภาพ: ประสิทธิภาพการย่อยอาหาร (ในการทำแผนที่จีโนม)
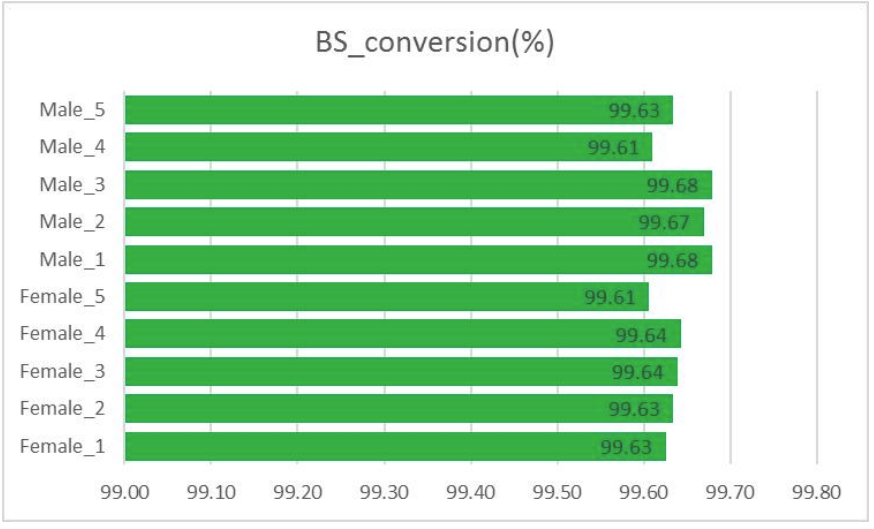
การควบคุมคุณภาพ: การแปลงไบซัลไฟต์ (ในการสกัดข้อมูลเมทิลเลชัน)
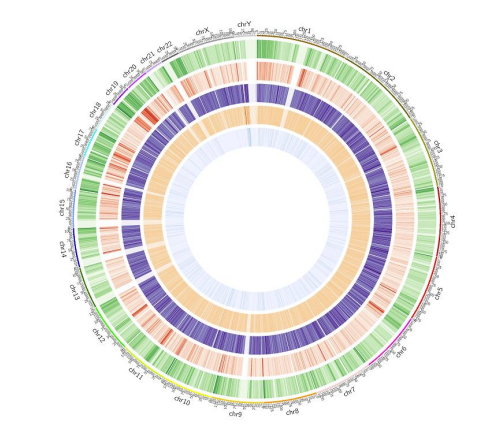
แผนที่เมทิลเลชัน: การกระจายทั่วทั้งจีโนมเมทิลเลชั่น 5mC
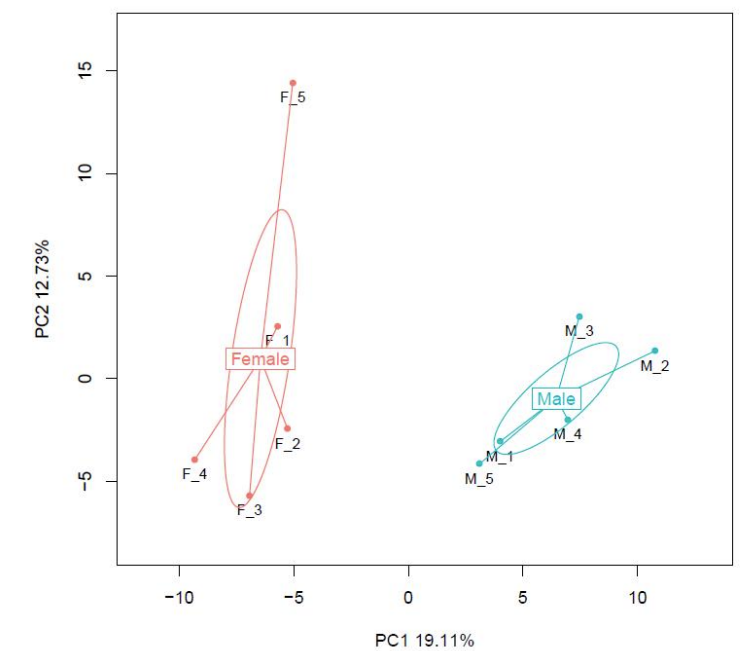
การเปรียบเทียบตัวอย่าง: การวิเคราะห์องค์ประกอบหลัก
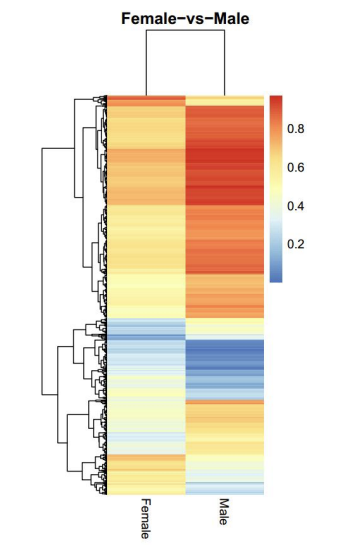
การวิเคราะห์บริเวณดิฟเฟอเรนเชียลเมทิลเลต (DMR): แผนที่ความร้อน
สำรวจความก้าวหน้าด้านการวิจัยที่ได้รับการสนับสนุนจากบริการจัดลำดับจีโนมไบซัลไฟต์ทั้งหมดของ BMKGene ผ่านคอลเลคชันสิ่งพิมพ์ที่คัดสรรมาอย่างดี
หลี่ ซี และคณะ (2022) 'การเขียนโปรแกรมใหม่ที่มีความเที่ยงตรงสูงลงในเซลล์ที่มีลักษณะคล้าย Leydig โดยการเปิดใช้งาน CRISPR และปัจจัยพาราคริน'พีนัส เน็กซัส, 1(4). ดอย: 10.1093/PNASNEXUS/PGAC179.
เทียน เอช และคณะ (2023) 'การวิเคราะห์เมทิลเลชั่นของ DNA ทั่วทั้งจีโนมขององค์ประกอบของร่างกายในฝาแฝดโมโนไซโกติกของจีน'วารสารยุโรปแห่งการสืบสวนทางคลินิก, 53(11), น. e14055. ดอย: 10.1111/ECI.14055.
วู ย. และคณะ (2022) 'DNA methylation และอัตราส่วนเอวต่อสะโพก: การศึกษาความสัมพันธ์ของ epigenome ในฝาแฝด monozygotic ของจีน'วารสารการสืบสวนต่อมไร้ท่อ, 45(12), หน้า 2365–2376. ดอย: 10.1007/S40618-022-01878-4.