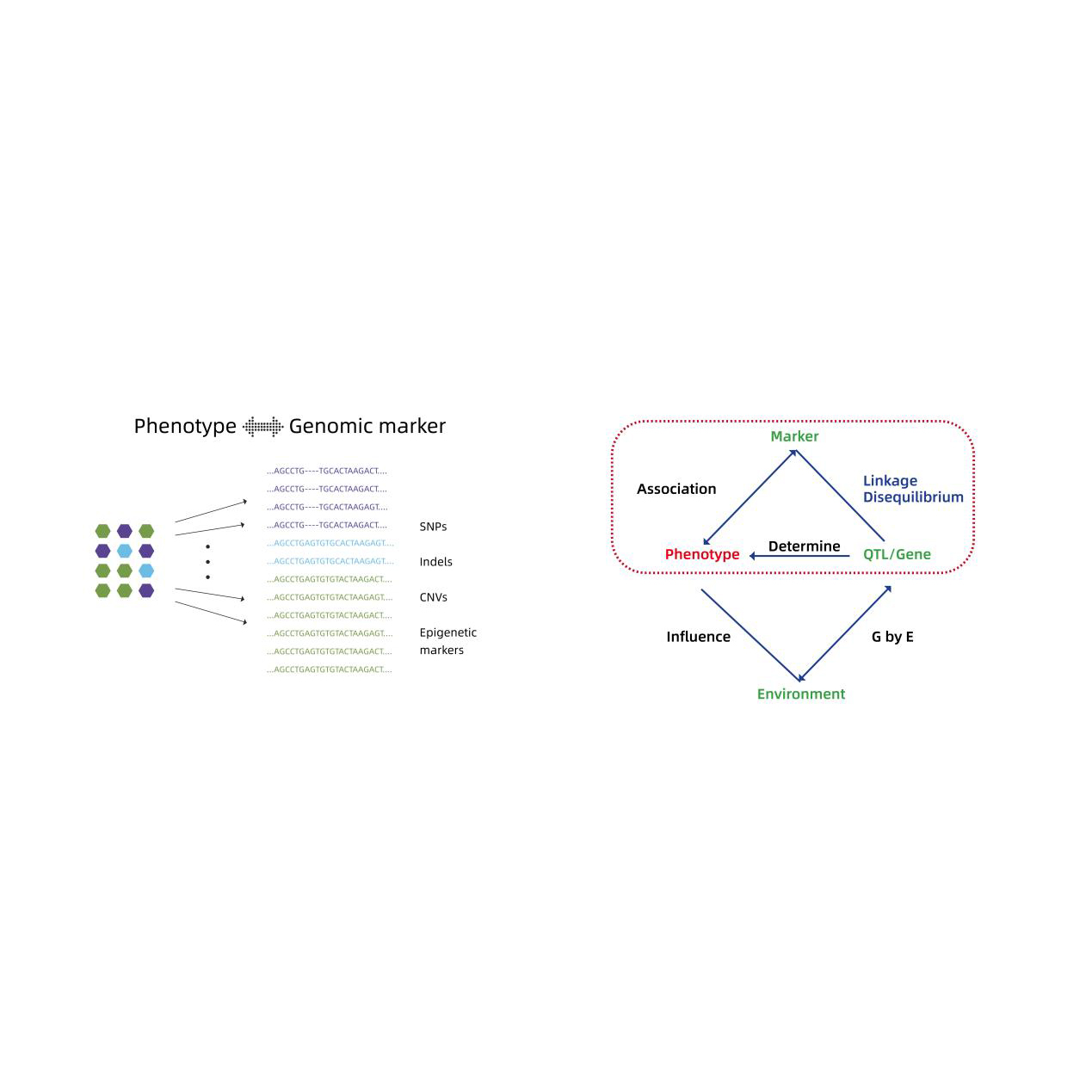
การวิเคราะห์ความสัมพันธ์ทั่วทั้งจีโนม
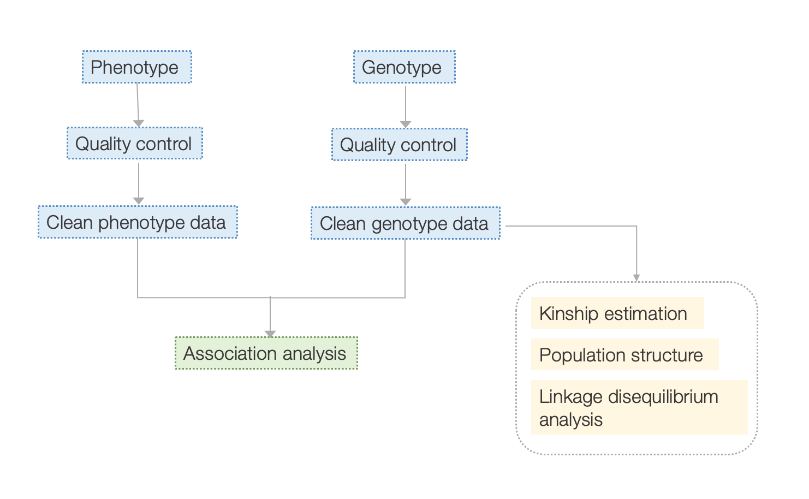
ขั้นตอนการทำงาน

ข้อดีของการบริการ
บันทึกความเชี่ยวชาญและสิ่งพิมพ์ที่กว้างขวาง: ด้วยประสบการณ์ที่สั่งสมมาใน GWAS BMKGene ได้ดำเนินโครงการวิจัย GWAS เกี่ยวกับประชากรหลายร้อยโครงการจนเสร็จสิ้น ช่วยนักวิจัยในการเผยแพร่บทความมากกว่า 100 บทความ และปัจจัยผลกระทบสะสมสูงถึง 500 รายการ
● การวิเคราะห์ชีวสารสนเทศที่ครอบคลุม: เวิร์กโฟลว์ประกอบด้วยการวิเคราะห์การเชื่อมโยงลักษณะ SNP โดยส่งมอบชุดของยีนผู้สมัครและคำอธิบายประกอบการทำงานที่เกี่ยวข้อง
ทีมชีวสารสนเทศศาสตร์ที่มีทักษะสูงและวงจรการวิเคราะห์ที่สั้น: ด้วยประสบการณ์อันยอดเยี่ยมในการวิเคราะห์จีโนมิกส์ขั้นสูง ทีมงานของ BMKGene จึงนำเสนอการวิเคราะห์ที่ครอบคลุมโดยใช้เวลาดำเนินการที่รวดเร็ว
การสนับสนุนหลังการขาย:ความมุ่งมั่นของเราขยายไปไกลกว่าการเสร็จสิ้นโครงการด้วยระยะเวลาบริการหลังการขาย 3 เดือน ในช่วงเวลานี้ เราเสนอการติดตามผลโครงการ ความช่วยเหลือในการแก้ไขปัญหา และช่วงถามตอบเพื่อตอบข้อสงสัยใดๆ ที่เกี่ยวข้องกับผลลัพธ์
ข้อกำหนดและข้อกำหนดการบริการ
| ประเภทของลำดับ | ขนาดประชากรที่แนะนำ | กลยุทธ์การจัดลำดับ | ข้อกำหนดของนิวคลีโอไทด์ |
| การหาลำดับจีโนมทั้งหมด | 200 ตัวอย่าง | 10x | ความเข้มข้น: ≥ 1 ng/ µL จำนวนรวม≥ 30ng จำกัดหรือไม่มีการย่อยสลายหรือการปนเปื้อน |
| ชิ้นส่วนขยายเฉพาะจุด (SLAF) | ความลึกของแท็ก: 10x จำนวนแท็ก: < 400 Mb: แนะนำให้ใช้ WGS < 1Gb: 100,000 แท็ก 1GB > 2Gb: แท็ก 300K แท็กสูงสุด 500,000 แท็ก | ความเข้มข้น ≥ 5 นาโนกรัม/ไมโครลิตร จำนวนรวม ≥ 80 ng นาโนดรอป OD260/280=1.6-2.5 เจลอะกาโรส: ไม่มีหรือจำกัดการย่อยสลายหรือการปนเปื้อน
|
การเลือกใช้วัสดุ



พันธุ์ที่แตกต่างกัน ชนิดย่อย พันธุ์พื้นเมือง/ธนาคารยีน/ตระกูลผสม/ทรัพยากรป่า
พันธุ์ต่าง ๆ ชนิดย่อย พันธุ์พื้นเมือง
ครอบครัวลูกครึ่ง/ครอบครัวลูกครึ่ง/ทรัพยากรธรรมชาติ
ขั้นตอนการทำงานบริการ

การออกแบบการทดลอง

การจัดส่งตัวอย่าง

การสกัดอาร์เอ็นเอ

การก่อสร้างห้องสมุด

การเรียงลำดับ

การวิเคราะห์ข้อมูล

บริการหลังการขาย
รวมถึงการวิเคราะห์ต่อไปนี้:
- การวิเคราะห์ความสัมพันธ์ทั่วทั้งจีโนม: โมเดล LM, LMM, EMMAX, FASTLMM
- คำอธิบายประกอบการทำงานของยีนผู้สมัคร
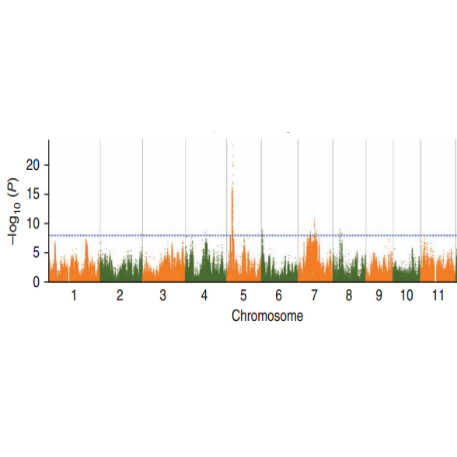
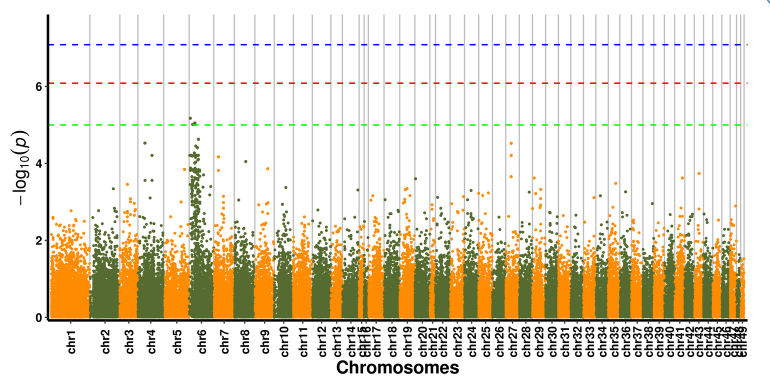
การวิเคราะห์ความสัมพันธ์ของลักษณะ SNP - โครงเรื่องแมนฮัตตัน
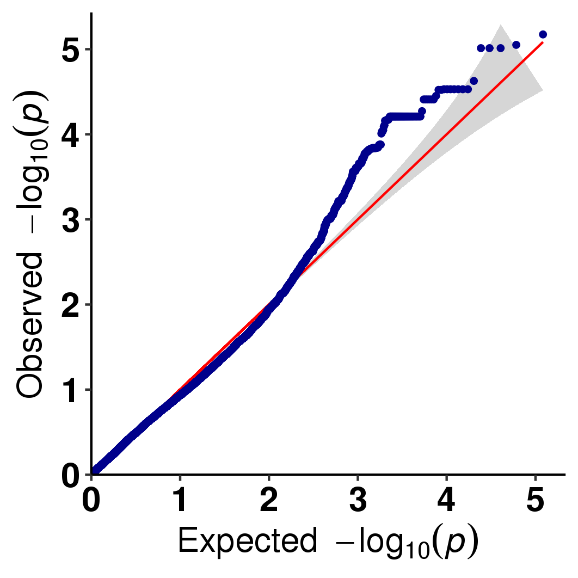
การวิเคราะห์ความสัมพันธ์ของลักษณะ SNP - พล็อต QQ
สำรวจความก้าวหน้าที่อำนวยความสะดวกโดยบริการ de GWAS ของ BMKGene ผ่านคอลเลคชันสิ่งพิมพ์ที่คัดสรรมาอย่างดี:
Lv, L. และคณะ (2023) 'ข้อมูลเชิงลึกเกี่ยวกับพื้นฐานทางพันธุกรรมของความทนทานต่อแอมโมเนียในหอยมีดโกน Sinonovacula constricta โดยการศึกษาความสัมพันธ์ทั่วทั้งจีโนม'การเพาะเลี้ยงสัตว์น้ำ, 569, น. 739351. ดอย: 10.1016/J.AQUACULTURE.2023.739351.
หลี่ เอ็กซ์ และคณะ (2022) 'การวิเคราะห์แบบหลายโอมิกส์ของลูกเดือยหางจิ้งจอก 398 ต้นเผยให้เห็นบริเวณจีโนมที่เกี่ยวข้องกับการเลี้ยงในบ้าน ลักษณะเมตาบอไลต์ และผลต้านการอักเสบ'พืชโมเลกุล, 15(8), หน้า 1367–1383. ดอย: 10.1016/j.molp.2022.07.003.
หลี่เจและคณะ (2022) 'การทำแผนที่ความสัมพันธ์ทั่วทั้งจีโนมของฟีโนไทป์แทบจะไม่มี Hulless ในสภาพแวดล้อมที่แห้งแล้ง'พรมแดนด้านพืชศาสตร์, 13, น. 924892. ดอย: 10.3389/FPLS.2022.924892/BIBTEX.
Zhao, X. และคณะ (2021) 'GmST1 ซึ่งเข้ารหัสซัลโฟทรานสเฟอเรส ให้ความต้านทานต่อไวรัสโมเสกถั่วเหลืองสายพันธุ์ G2 และ G3'พืช เซลล์ และสิ่งแวดล้อม, 44(8), หน้า 2777–2792. ดอย: 10.1111/PCE.14066.