การชุมนุมจีโนมของเชื้อรา Novo
คุณสมบัติบริการ
ด้วยสามตัวเลือกที่เป็นไปได้ให้เลือกขึ้นอยู่กับระดับความสมบูรณ์ของจีโนมที่ต้องการ:
●ตัวเลือกจีโนมร่าง: การเรียงลำดับการอ่านระยะสั้นกับ Illumina Novaseq PE150
●ตัวเลือกจีโนมที่ดีของเชื้อรา:
การสำรวจจีโนม: Illumina Novaseq PE150
ชุดจีโนม: Pacbio revio (HIFI อ่าน) หรือ Nanopore Promethion 48
●จีโนมเชื้อราระดับโครโมโซม:
การสำรวจจีโนม: Illumina Novaseq PE150
ชุดจีโนม: Pacbio revio (HIFI อ่าน) หรือ Nanopore Promethion 48
contig anchoring ด้วยชุดประกอบ Hi-C
ข้อได้เปรียบด้านการบริการ
มีกลยุทธ์การเรียงลำดับหลายแบบ: สำหรับเป้าหมายการวิจัยที่แตกต่างกันและข้อกำหนดของความสมบูรณ์ของจีโนม
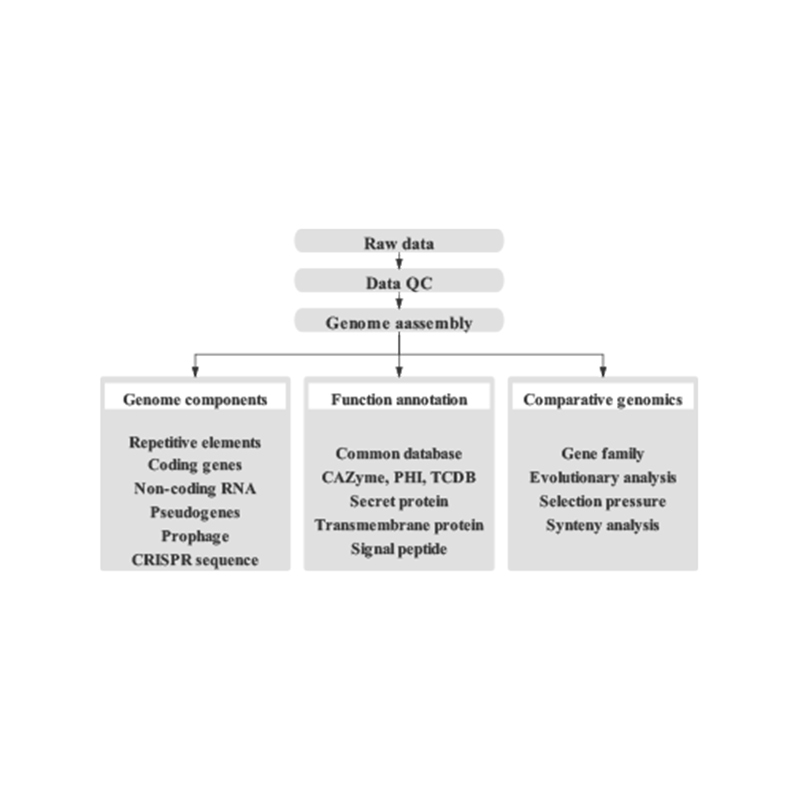
ขั้นตอนการทำงานทางชีวสารสนเทศศาสตร์ที่สมบูรณ์:ซึ่งรวมถึงการประกอบจีโนมและการทำนายขององค์ประกอบจีโนมที่หลากหลายคำอธิบายประกอบของยีนที่ใช้งานได้และการยึด contig
ความเชี่ยวชาญอย่างกว้างขวาง: ด้วยจีโนมจุลินทรีย์มากกว่า 12,000 ตัวที่รวมตัวกันเรานำประสบการณ์มานานกว่าทศวรรษทีมวิเคราะห์ที่มีทักษะสูงเนื้อหาที่ครอบคลุมและการสนับสนุนหลังการขายที่ยอดเยี่ยม
การสนับสนุนหลังการขาย:ความมุ่งมั่นของเราครอบคลุมเกินกว่าที่โครงการจะเสร็จสิ้นด้วยระยะเวลาการบริการหลังการขาย 3 เดือน ในช่วงเวลานี้เราเสนอการติดตามโครงการความช่วยเหลือในการแก้ไขปัญหาและเซสชันคำถามและคำตอบเพื่อตอบคำถามใด ๆ ที่เกี่ยวข้องกับผลลัพธ์
ข้อกำหนดบริการ
| บริการ | กลยุทธ์การเรียงลำดับ | การควบคุมคุณภาพ |
| ร่างจีโนม | Illumina PE150 100X | Q30≥85% |
| จีโนมที่ดี | การสำรวจจีโนม: Illumina PE150 50 X แอสเซมบลี: Pacbio hifi 30x หรือ nanopore 100x | Contig N50 ≥1MB (PACBIO Unicellular) Contig N50 ≥2MB (ONT Unecellular) Contig N50 ≥500KB (อื่น ๆ ) |
| จีโนมระดับโครโมโซม | การสำรวจจีโนม: Illumina PE150 50 X แอสเซมบลี: Pacbio hifi 30x หรือ nanopore 100x แอสเซมบลี Hi-C 100x | อัตราส่วนการยึด Contig> 90%
|
ข้อกำหนดบริการ
| ความเข้มข้น (ng/µl) | จำนวนเงินทั้งหมด (µg) | ระดับเสียง (µl) | OD260/280 | OD260/230 | |
| Pacbio | ≥20 | ≥2 | ≥20 | 1.7-2.2 | ≥1.6 |
| นาโน | ≥40 | ≥2 | ≥20 | 1.7-2.2 | 1.0-3.0 |
| อิลลูมินา | ≥1 | ≥0.06 | ≥20 | - | - |
เชื้อราเดียว: ≥3.5x1010 เซลล์
เชื้อราแมโคร: ≥10กรัม
งานบริการ

การส่งตัวอย่าง

การก่อสร้างห้องสมุด

การเรียงลำดับ

การวิเคราะห์ข้อมูล

บริการหลังการขาย
รวมถึงการวิเคราะห์ต่อไปนี้:
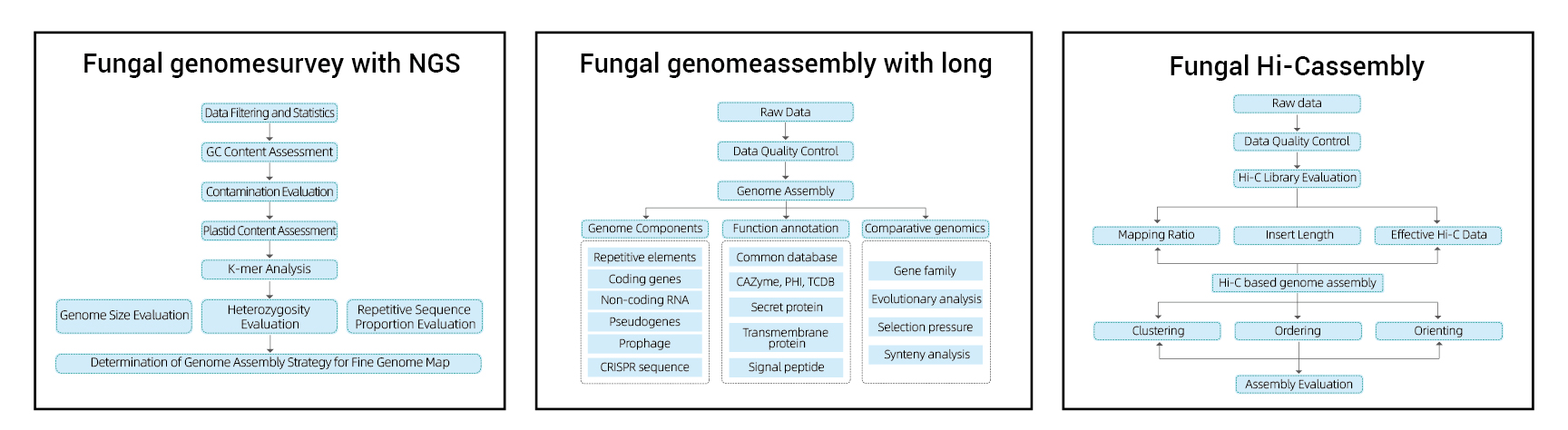
การสำรวจจีโนม:
- การเรียงลำดับการควบคุมคุณภาพข้อมูล
- การประมาณจีโนม: ขนาด, heterozygosity, องค์ประกอบซ้ำ ๆ
การประกอบจีโนมที่ดี:
- การเรียงลำดับการควบคุมคุณภาพข้อมูล
- เดอโนโวการประกอบ
- การวิเคราะห์องค์ประกอบจีโนม: การทำนายซีดีและองค์ประกอบจีโนมหลายชนิด
- คำอธิบายประกอบการทำงานที่มีฐานข้อมูลทั่วไปหลายแห่ง (GO, KEGG, ฯลฯ ) และฐานข้อมูลขั้นสูง (การ์ด, VFDB ฯลฯ )
แอสเซมบลี HI-C:
- การประเมินห้องสมุด HI-C
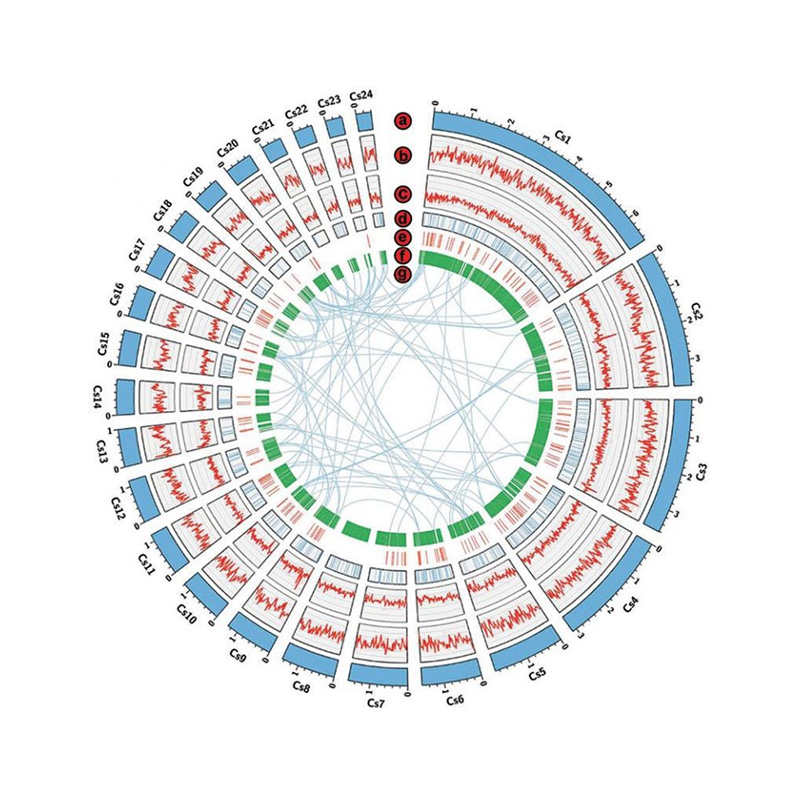
- contigs anchoring clustering การสั่งซื้อและการปรับทิศทาง
- การประเมินแอสเซมบลี Hi-C: ขึ้นอยู่กับจีโนมอ้างอิงและแผนที่ความร้อน
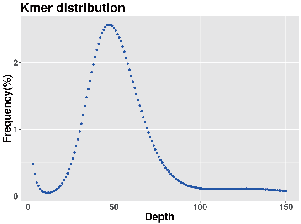
การสำรวจจีโนม: การกระจาย k-mer
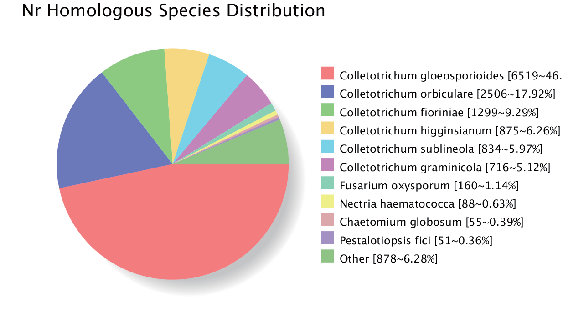
การประกอบจีโนม: คำอธิบายประกอบที่คล้ายคลึงกันของยีน (ฐานข้อมูล NR)
การประกอบจีโนม: คำอธิบายประกอบของยีนที่ใช้งานได้ (GO)
สำรวจความก้าวหน้าที่อำนวยความสะดวกโดยบริการประกอบจีโนมเชื้อราของ BMKGENE ผ่านการรวบรวมสิ่งพิมพ์ที่รวบรวมไว้
Hao, J. et al. (2023) 'การทำโปรไฟล์ OMIC แบบบูรณาการของเห็ดยา Inonotus Obliquus ภายใต้เงื่อนไขที่จมอยู่ใต้น้ำ'BMC Genomics, 24 (1), pp. 1–12 ดอย: 10.1186/s12864-023-09656-z/ตัวเลข/3
Lu, L. et al. (2023) 'การหาลำดับจีโนมเผยให้เห็นวิวัฒนาการและกลไกการเกิดโรคของข้าวสาลีที่คมชัด eyespot rhizoctonia cerealis'วารสารพืชผล, 11 (2), pp. 405–416 ดอย: 10.1016/j.cj.2022.07.024
จาง, H. et al. (2023) 'ทรัพยากรจีโนมสำหรับสี่สายพันธุ์ Clarireedia ที่ก่อให้เกิดจุดเงินดอลลาร์บนสนามหญ้าที่หลากหลาย'โรคพืช, 107 (3), pp. 929–934 ดอย: 10.1094/PDIS-08-22-1921-A
จางเอสเอสและคณะ (2023) 'หลักฐานทางพันธุกรรมและโมเลกุลของระบบผสมพันธุ์ tetrapolar ในเห็ดที่กินได้ Grifola Frondosa'วารสาร, 9 (10), p. 959. ดอย: 10.3390/jof9100959/s1