ลำดับ mRNA แบบเต็มความยาว
คุณสมบัติ
●การจับภาพโพลี -a mRNA ตามด้วยการสังเคราะห์ cDNA และการเตรียมห้องสมุด
●ลำดับการถอดเสียงเต็มความยาว
●การวิเคราะห์ทางชีวภาพตามการจัดแนวกับจีโนมอ้างอิง
●การวิเคราะห์ทางชีวสารสนเทศไม่เพียง แต่การแสดงออกที่ยีนและระดับไอโซฟอร์ม แต่ยังวิเคราะห์ lncRNA, การหลอมรวมของยีน, โพลี-อะเดนิเลชั่นและโครงสร้างยีน
ข้อได้เปรียบด้านการบริการ
ปริมาณการแสดงออกในระดับไอโซฟอร์ม: การเปิดใช้งานการวิเคราะห์การแสดงออกอย่างละเอียดและแม่นยำการเปิดเผยการเปลี่ยนแปลงที่อาจถูกปกปิดเมื่อวิเคราะห์การแสดงออกของยีนทั้งหมด
ความต้องการข้อมูลที่ลดลง:เมื่อเปรียบเทียบกับการจัดลำดับรุ่นต่อไป (NGS) การจัดลำดับ nanopore แสดงความต้องการข้อมูลที่ต่ำกว่าช่วยให้ระดับความอิ่มตัวของปริมาณการแสดงออกของยีนที่เทียบเท่ากับข้อมูลที่เล็กลง
ความแม่นยำสูงขึ้นของปริมาณการแสดงออก: ทั้งในระดับยีนและไอโซฟอร์ม
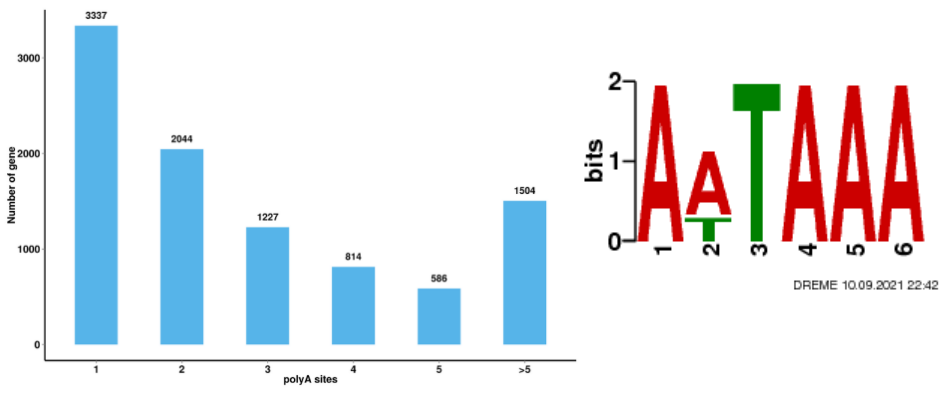
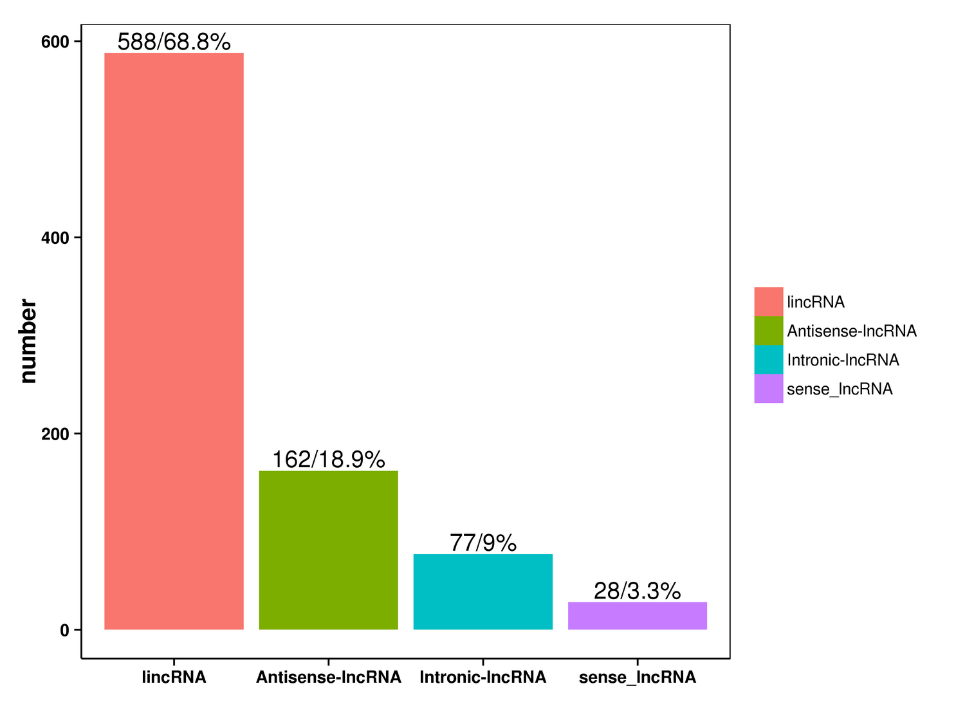
การระบุข้อมูล transcriptomic เพิ่มเติม: polyadenylation ทางเลือกยีนฟิวชั่นและ lcnRNA และยีนเป้าหมายของพวกเขา
ความเชี่ยวชาญอย่างกว้างขวาง: ทีมงานของเรานำประสบการณ์มากมายมาสู่ทุกโครงการโดยเสร็จสิ้นโครงการ transcriptome เต็มรูปแบบเต็มรูปแบบกว่า 850 nanopore และประมวลผลตัวอย่างมากกว่า 8,000 ตัวอย่าง
การสนับสนุนหลังการขาย: ความมุ่งมั่นของเราครอบคลุมเกินกว่าที่โครงการจะเสร็จสิ้นด้วยระยะเวลาการบริการหลังการขาย 3 เดือน ในช่วงเวลานี้เราเสนอการติดตามโครงการความช่วยเหลือในการแก้ไขปัญหาและเซสชันคำถามและคำตอบเพื่อตอบคำถามใด ๆ ที่เกี่ยวข้องกับผลลัพธ์
ข้อกำหนดตัวอย่างและการจัดส่ง
| ห้องสมุด | กลยุทธ์การเรียงลำดับ | ข้อมูลแนะนำ | การควบคุมคุณภาพ |
| โพลี A เสริม | Illumina PE150 | 6/12 GB | คะแนนคุณภาพเฉลี่ย: Q10 |
ข้อกำหนดตัวอย่าง:
นิวคลีโอไทด์:
| conc. (ng/μl) | จำนวน (μg) | ความบริสุทธิ์ | ความซื่อสัตย์ |
| ≥ 100 | ≥ 1.0 | OD260/280 = 1.7-2.5 OD260/230 = 0.5-2.5 การปนเปื้อนโปรตีนหรือดีเอ็นเอที่ จำกัด หรือไม่มีเลยที่แสดงบนเจล | สำหรับพืช: rin≥7.0; สำหรับสัตว์: rin≥7.5; 5.0≥28S/18S≥1.0; ระดับความสูงที่ จำกัด หรือไม่มีเลย |
●พืช:
รูตลำต้นหรือกลีบดอก: 450 มก.
ใบหรือเมล็ด: 300 มก.
ผลไม้: 1.2 กรัม
●สัตว์:
หัวใจหรือลำไส้: 300 มก.
อวัยวะภายในหรือสมอง: 240 มก.
กล้ามเนื้อ: 450 มก.
กระดูกผมหรือผิวหนัง: 1G
● Arthropods:
แมลง: 6g
Crustacea: 300 มก.
●เลือดทั้งหมด: 1 หลอด
●เซลล์: 106 เซลล์
การจัดส่งตัวอย่างที่แนะนำ
ภาชนะบรรจุ: ไม่แนะนำให้ใช้หลอดเซนติเมตร 2 มล. (ไม่แนะนำให้ใช้ฟอยล์ดีบุก)
การติดฉลากตัวอย่าง: กลุ่ม+ทำซ้ำเช่น A1, A2, A3; B1, B2, B3
การจัดส่ง:
1. น้ำแข็งแห้ง: ตัวอย่างต้องบรรจุในถุงและฝังในน้ำแข็งแห้ง
2. หลอด rnastable: ตัวอย่าง RNA สามารถทำให้แห้งในหลอดเสถียร RNA (เช่นRnastable®) และจัดส่งในอุณหภูมิห้อง
งานบริการ
นิวคลีโอไทด์:

การส่งตัวอย่าง

การก่อสร้างห้องสมุด

การเรียงลำดับ

การวิเคราะห์ข้อมูล

บริการหลังการขาย
งานบริการ
เนื้อเยื่อ:

การออกแบบการทดลอง

การส่งตัวอย่าง

การสกัด RNA

การก่อสร้างห้องสมุด

การเรียงลำดับ

การวิเคราะห์ข้อมูล

บริการหลังการขาย
●การประมวลผลข้อมูลดิบ
●การระบุการถอดเสียง
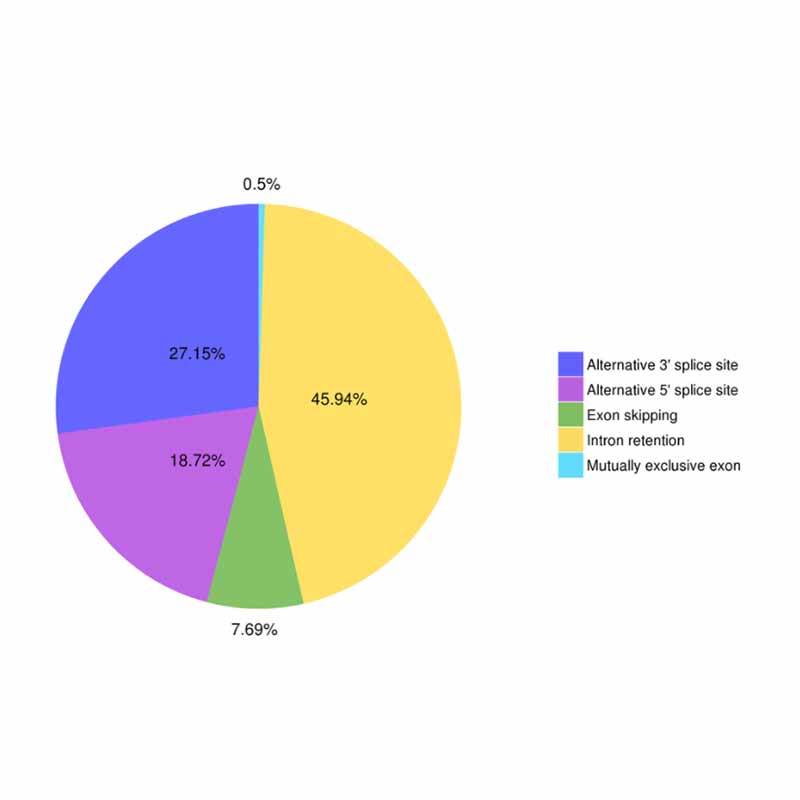
●การประกบทางเลือก
●ปริมาณการแสดงออกในระดับยีนและระดับไอโซฟอร์ม
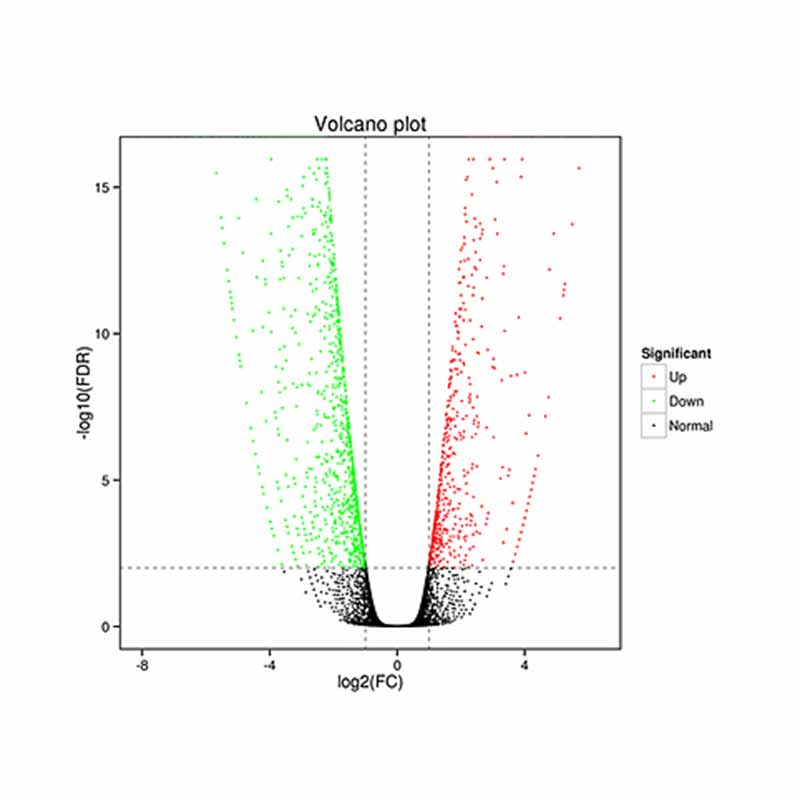
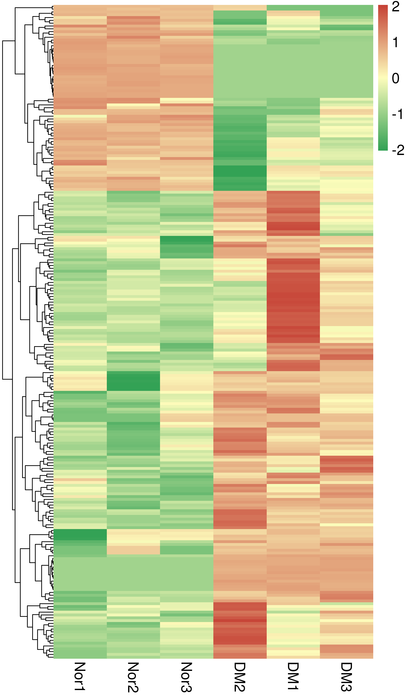
●การวิเคราะห์การแสดงออกที่แตกต่างกัน
●ฟังก์ชั่นคำอธิบายประกอบและการเพิ่มคุณค่า (DEGs and dets)
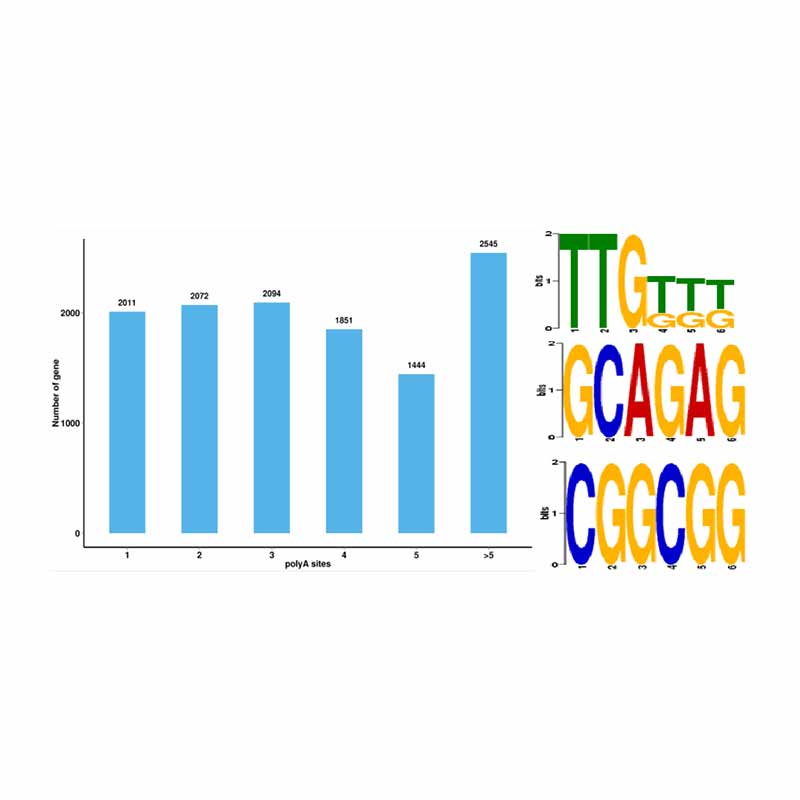
การวิเคราะห์การประกบทางเลือก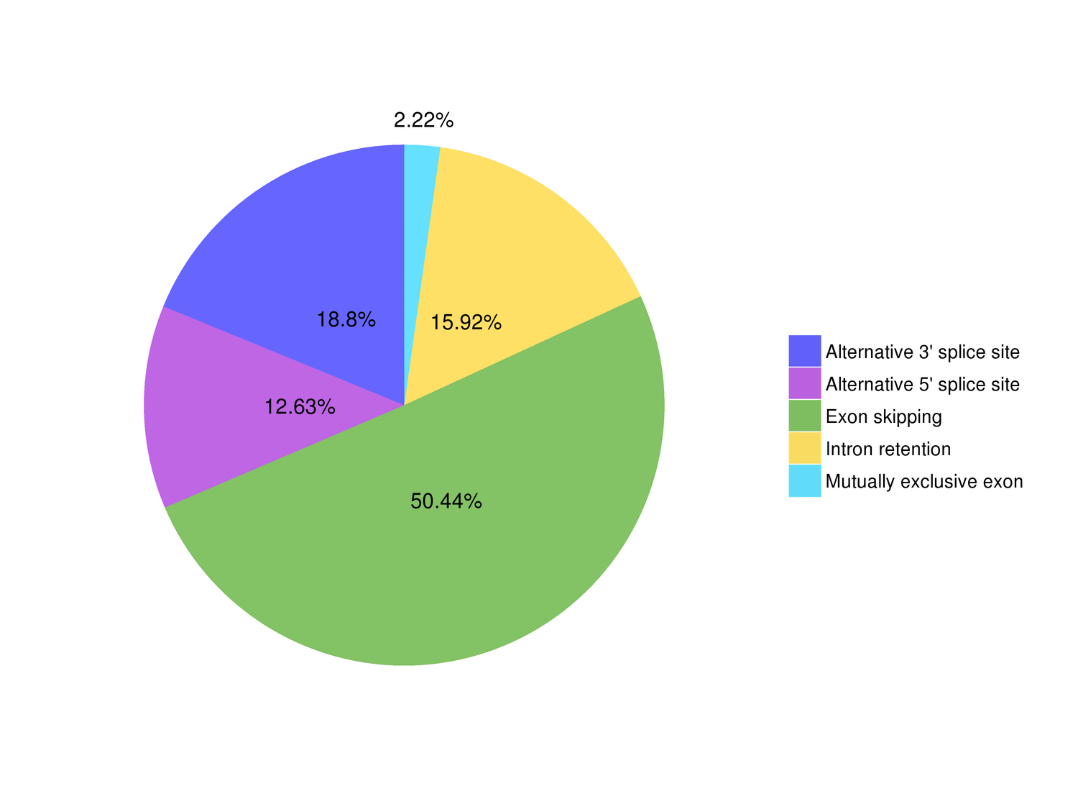 การวิเคราะห์ polyadenylation ทางเลือก (APA)
การวิเคราะห์ polyadenylation ทางเลือก (APA)
การทำนาย lncRNA
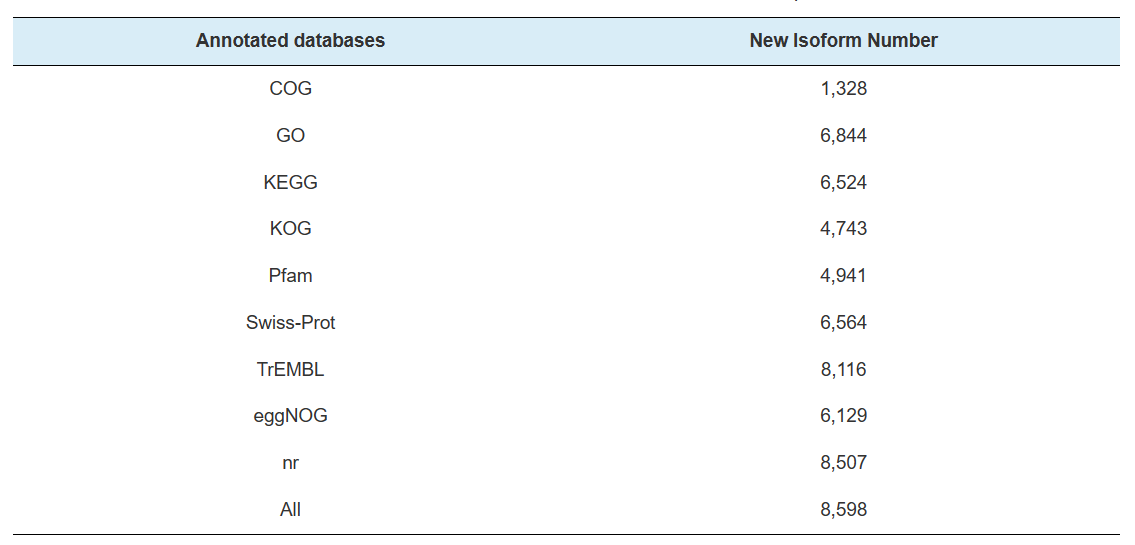
คำอธิบายประกอบของยีนนวนิยาย
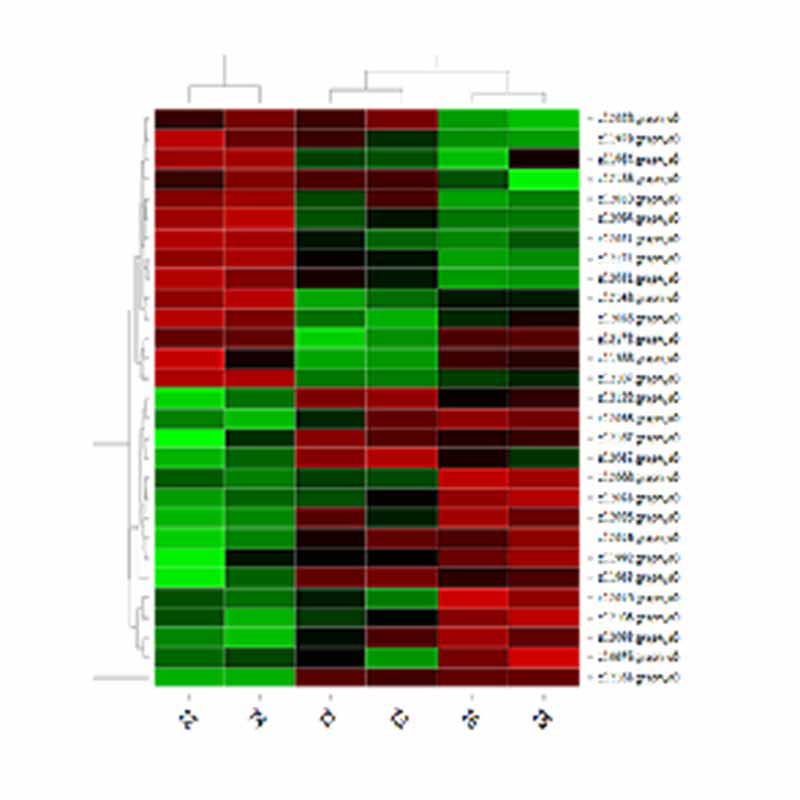
การจัดกลุ่มของ dets
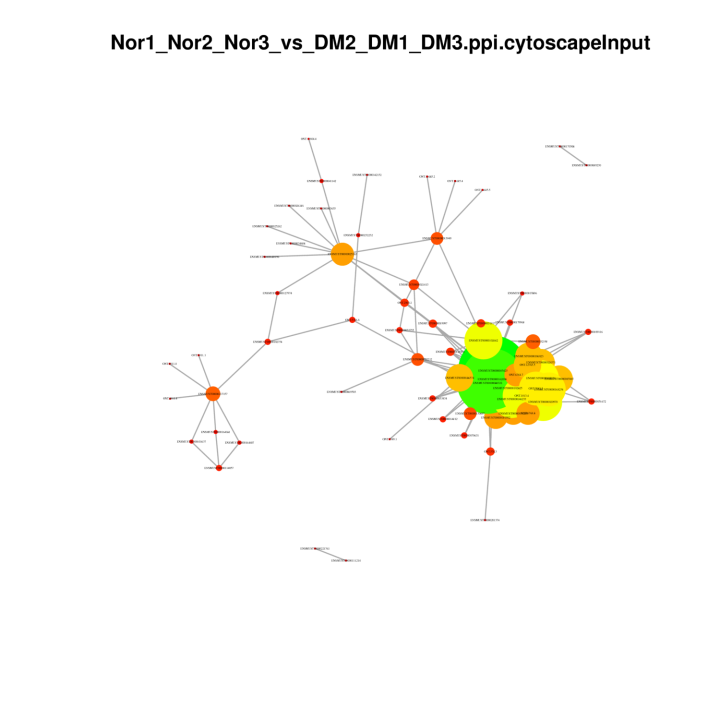
เครือข่ายโปรตีนโปรตีนใน DEGS
สำรวจความก้าวหน้าที่อำนวยความสะดวกโดยบริการลำดับ mRNA แบบเต็มความยาวของ BMKGENE ของ BMKGENE ผ่านการรวบรวมสิ่งพิมพ์ที่รวบรวมไว้
กง, B. และคณะ (2023) 'การเปิดใช้งาน epigenetic และ transcriptional ของ secretory kinase fam20c เป็น oncogene ใน glioma', วารสารพันธุศาสตร์และจีโนม, 50 (6), pp. 422–433 ดอย: 10.1016/j.jgg.2023.01.008
เขา Z. et al. (2023) 'การเรียงลำดับ transcriptome ความยาวเต็มรูปแบบของเซลล์เม็ดเลือดขาวตอบสนองต่อ IFN-γเผยให้เห็นการตอบสนองทางภูมิคุ้มกันแบบ TH1-th1 ใน flounder (paralichthys olivaceus)', ภูมิคุ้มกันวิทยาปลาและหอย, 134, p. 108636. ดอย: 10.1016/j.fsi.2023.108636
Ma, Y. et al. (2023) 'การวิเคราะห์เปรียบเทียบวิธีการเรียงลำดับ PACBIO และ ONT RNA สำหรับการระบุ Nemopilema Nomurai Venom', Genomics, 115 (6), p. 110709. ดอย: 10.1016/j.ygeno.2023.110709
Yu, D. et al. (2023) 'การวิเคราะห์ Nano-Seq เผยให้เห็นแนวโน้มการทำงานที่แตกต่างกันระหว่าง exosomes และ microvesicles ที่ได้มาจาก HUMSC', การวิจัยเซลล์ต้นกำเนิดและการบำบัด, 14 (1), pp. 1–13 ดอย: 10.1186/s13287-023-03491-5/ตาราง/6