จีโนมเปรียบเทียบ
ข้อได้เปรียบด้านการบริการ
บันทึกความเชี่ยวชาญและสิ่งพิมพ์ที่กว้างขวาง: ด้วยการสะสม BMKGENE ได้เสร็จสิ้นโครงการจีโนมเปรียบเทียบมากกว่า 90 โครงการโดยมีปัจจัยผลกระทบสะสมถึง 900
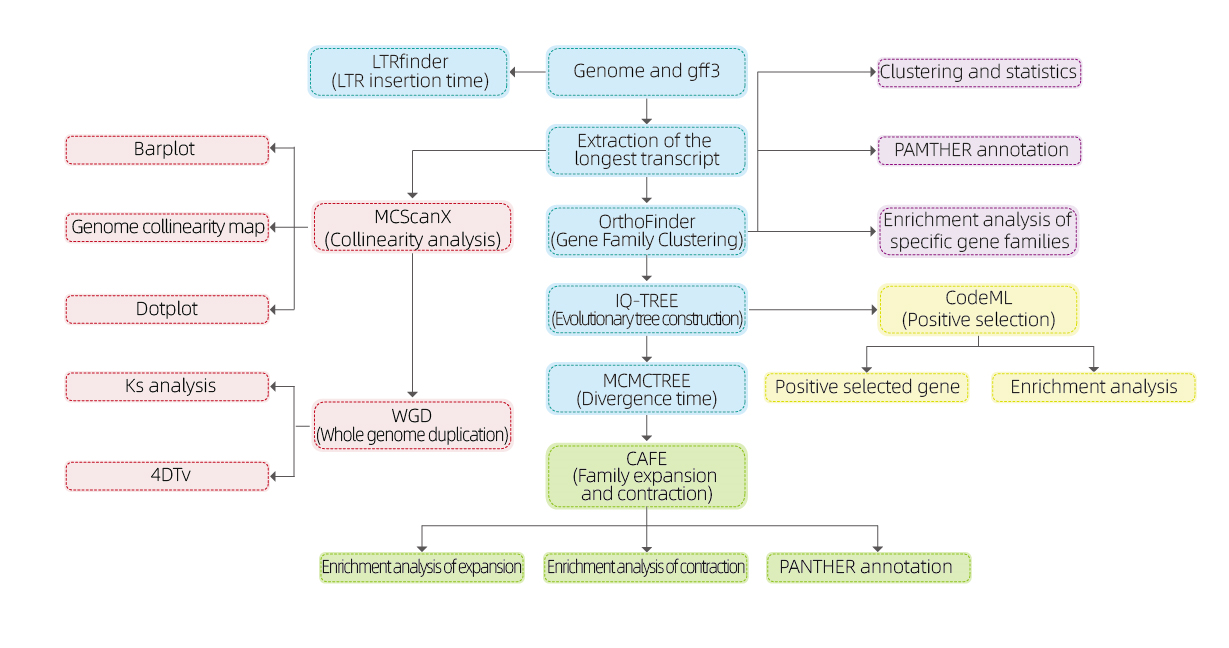
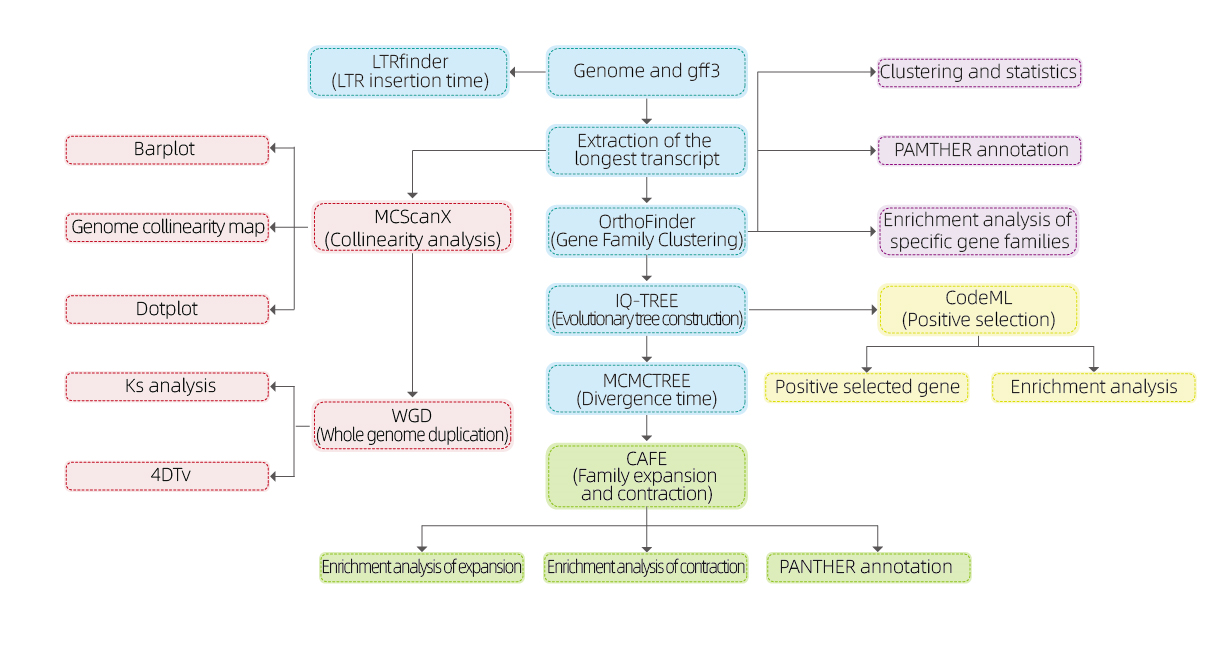
การวิเคราะห์ทางชีวสารสนเทศที่ครอบคลุม: แพ็คเกจการวิเคราะห์มีการวิเคราะห์ที่จำเป็นมากที่สุดแปดประการโดยให้ตัวเลขพร้อมที่เผยแพร่อย่างดีและช่วยให้สามารถตีความผลลัพธ์ได้อย่างง่ายดาย
ทีมชีวสารสนเทศศาสตร์ที่มีทักษะสูงและรอบการวิเคราะห์สั้น ๆ: ด้วยประสบการณ์ที่ยอดเยี่ยมในการวิเคราะห์จีโนมเปรียบเทียบทีมของ BMKGENE ตอบสนองความต้องการการวิเคราะห์ส่วนบุคคลที่หลากหลายในระยะเวลาสั้น ๆ
การสนับสนุนหลังการขาย:ความมุ่งมั่นของเราครอบคลุมเกินกว่าที่โครงการจะเสร็จสิ้นด้วยระยะเวลาการบริการหลังการขาย 3 เดือน ในช่วงเวลานี้เราเสนอการติดตามโครงการความช่วยเหลือในการแก้ไขปัญหาและเซสชันคำถามและคำตอบเพื่อตอบคำถามใด ๆ ที่เกี่ยวข้องกับผลลัพธ์
ข้อกำหนดบริการ
| เวลาเทิร์นรอบโดยประมาณ | จำนวนสปีชีส์ | วิเคราะห์ |
| 30 วันทำการ | 6 - 12 | การจัดกลุ่มครอบครัวยีน การขยายตัวและการหดตัวของครอบครัวยีน การก่อสร้างต้นไม้สายวิวัฒนาการ การประมาณเวลาที่แตกต่าง (จำเป็นต้องมีการสอบเทียบฟอสซิล) เวลาแทรก LTR (สำหรับพืช) การทำซ้ำจีโนมทั้งหมด (สำหรับพืช) ความดันเลือก การวิเคราะห์ synteny |
การวิเคราะห์ทางชีวสารสนเทศศาสตร์
●ครอบครัวยีน
●สายวิวัฒนาการ
●เวลาที่แตกต่างกัน
●ความดันเลือก
●การวิเคราะห์ synteny
ข้อกำหนดตัวอย่างและการจัดส่ง
ข้อกำหนดตัวอย่าง:
เนื้อเยื่อหรือ DNA สำหรับการจัดลำดับจีโนมและการประกอบ
สำหรับเนื้อเยื่อ
| สายพันธุ์ | เนื้อเยื่อ | สำรวจ | Pacbio CCS |
| สัตว์ | เนื้อเยื่ออวัยวะภายใน | 0.5 ~ 1 กรัม | ≥ 3.5 กรัม |
| เนื้อเยื่อกล้ามเนื้อ | |||
| ≥ 5.0 กรัม | |||
| ≥ 5.0 มล. | |||
| เลือดสัตว์เลี้ยงลูกด้วยนม | |||
| ≥ 0.5 มล. | |||
| เลือดสัตว์ปีก/ปลา | |||
| ปลูก | ใบสด | 1 ~ 2 กรัม | ≥ 5.0 กรัม |
| กลีบดอก/ลำต้น | 1 ~ 2 กรัม | ≥ 10.0 กรัม | |
| ราก/เมล็ด | 1 ~ 2 กรัม | ≥ 20.0 กรัม | |
| เซลล์ | เซลล์เพาะเลี้ยง | - | ≥ 1 x 108 |
ข้อมูล
ไฟล์ลำดับจีโนม (.fasta) และไฟล์คำอธิบายประกอบ (.gff3) ของสปีชีส์ที่เกี่ยวข้องอย่างใกล้ชิด
งานบริการ

การออกแบบการทดลอง

การส่งตัวอย่าง

การก่อสร้างห้องสมุด

การเรียงลำดับ

การวิเคราะห์ข้อมูล

บริการหลังการขาย
*ผลการสาธิตที่แสดงที่นี่ทั้งหมดมาจากจีโนมที่ตีพิมพ์ด้วยเทคโนโลยีชีวภาพ
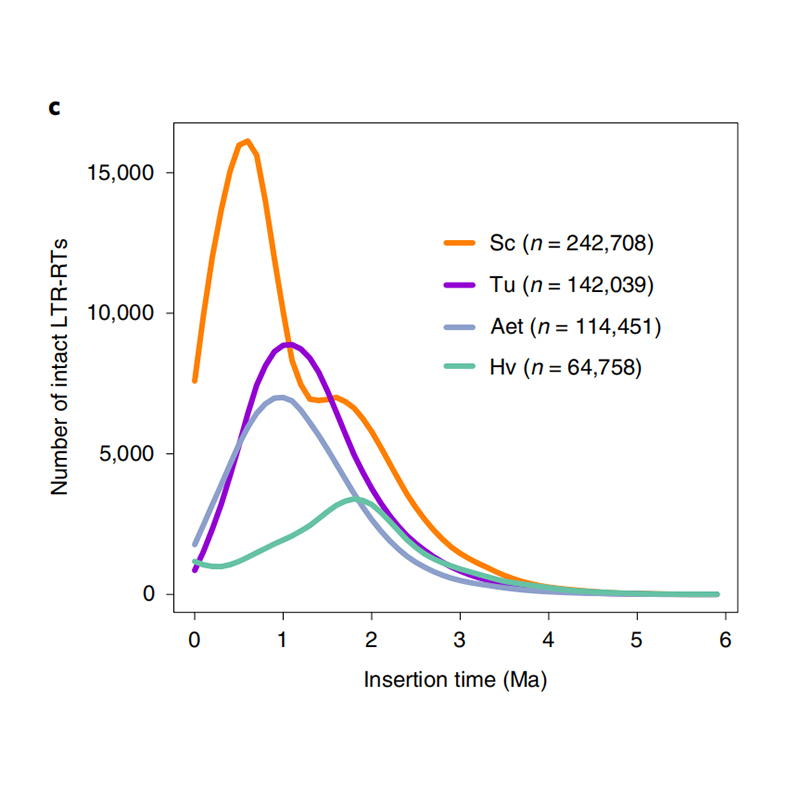
1. การประมาณเวลาแทรก LTR: รูปที่แสดงการกระจาย bimodal ที่เป็นเอกลักษณ์ในเวลาแทรก LTR-RTS ในจีโนมไรย์ที่มีไวอยู่เมื่อเทียบกับสายพันธุ์อื่น ๆ จุดสูงสุดล่าสุดปรากฏขึ้นเมื่อประมาณ 0.5 ล้านปีก่อน
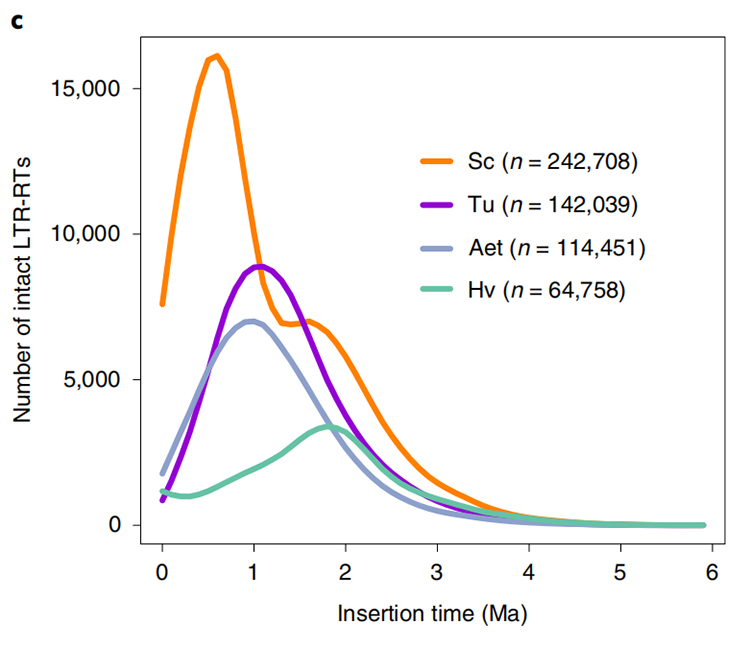
Li Guang et al.พันธุศาสตร์ธรรมชาติ, 2021
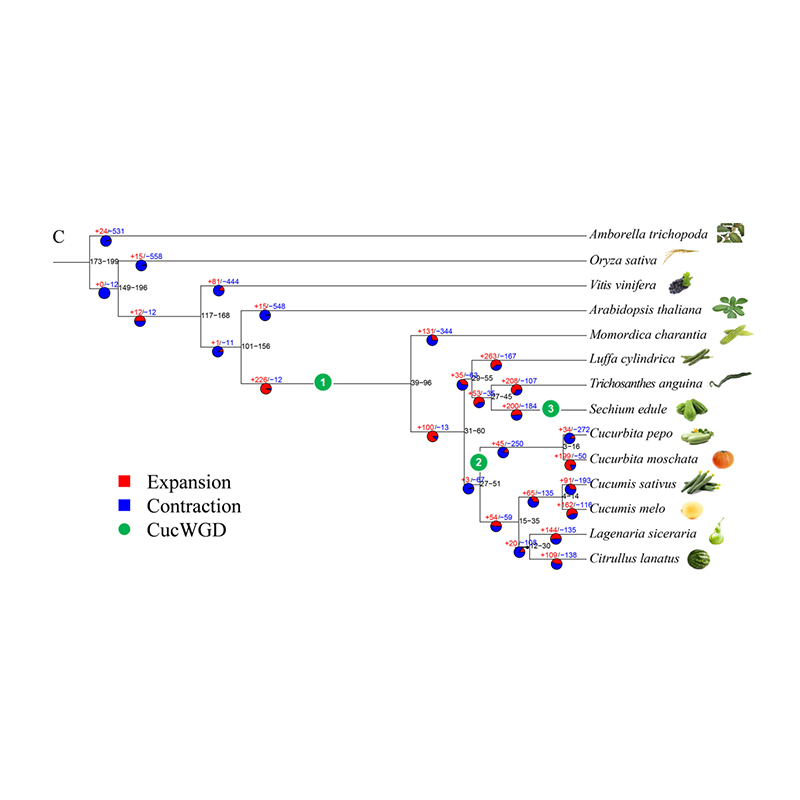
2.Phylogeny และการวิเคราะห์ตระกูลยีนเกี่ยวกับ Chayote (SECHIUM Edule): โดยการวิเคราะห์ chayote และอีก 13 สายพันธุ์ที่เกี่ยวข้องในตระกูลยีนพบว่ามีความสัมพันธ์อย่างใกล้ชิดที่สุดกับงูมะระ (Trichosanthes Anguina) Chayote ได้มาจากงูมะระในประมาณ 27-45 mya และการทำซ้ำจีโนมทั้งหมด (WGD) ถูกพบใน Chayote ใน 25 ± 4 mya ซึ่งเป็นเหตุการณ์ WGD ที่สามใน Cucuibitaceae
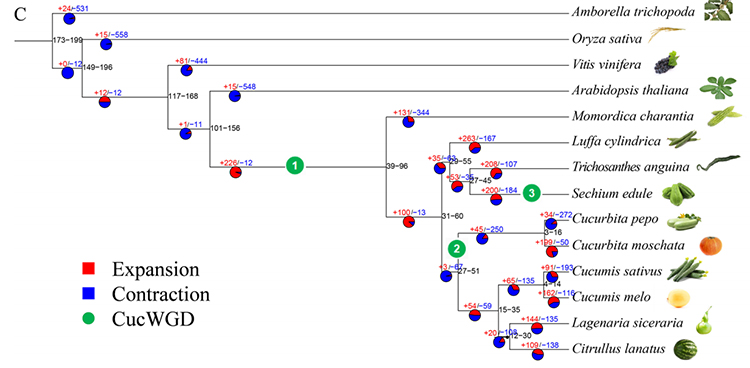
fu a et al.การวิจัยพืชสวน, 2021
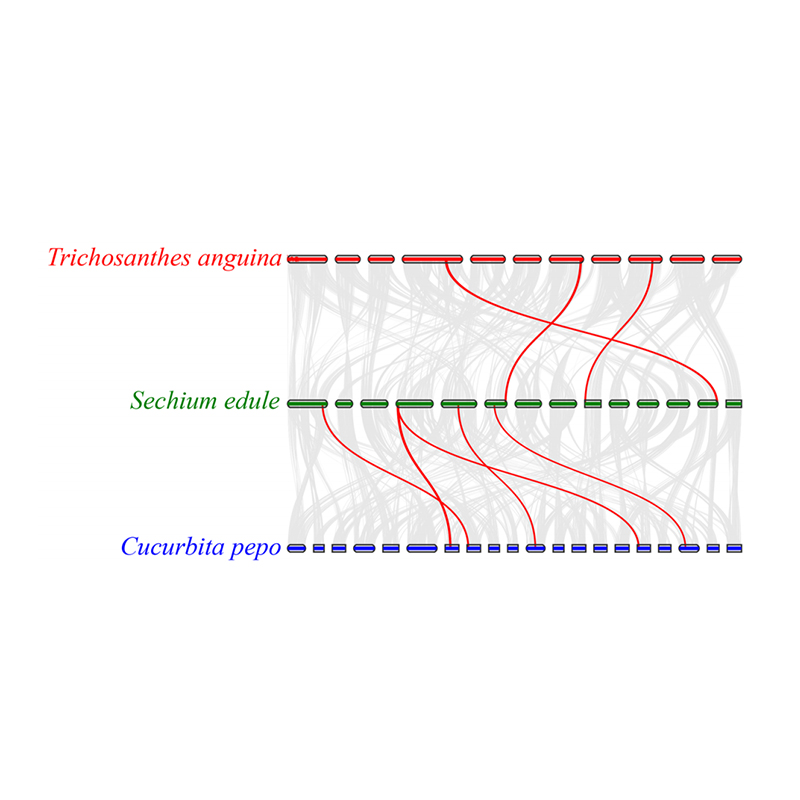
3.Synteny การวิเคราะห์: ยีนบางชนิดที่เกี่ยวข้องกับไฟโตฮอร์โมนในการพัฒนาผลไม้พบได้ใน chayote, งูมะระและสควอช ความสัมพันธ์ระหว่าง chayote และสควอชสูงกว่านั้นเล็กน้อยระหว่าง chayote และงูมะระ
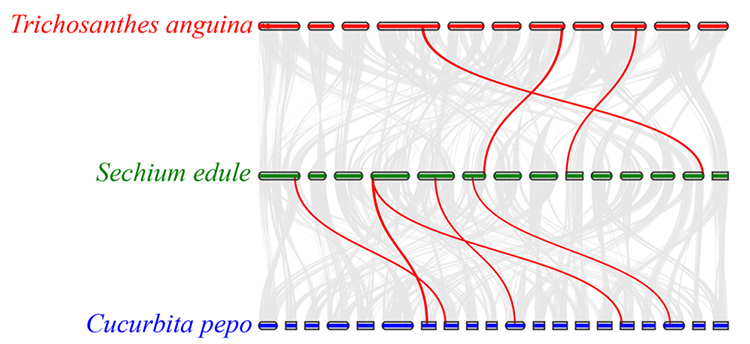
fu a et al.การวิจัยพืชสวน, 2021
4. การวิเคราะห์ครอบครัวของ Gene: การเพิ่มคุณค่าของ Kegg เกี่ยวกับการขยายตัวของครอบครัวยีนและการหดตัวใน G.thurberi และ G.davidsonii Genomes แสดงให้เห็นว่าการสังเคราะห์สเตียรอยด์และการสังเคราะห์ brassinosteroid biosynthesis
Yang Z et al.ชีววิทยา BMC, 2021
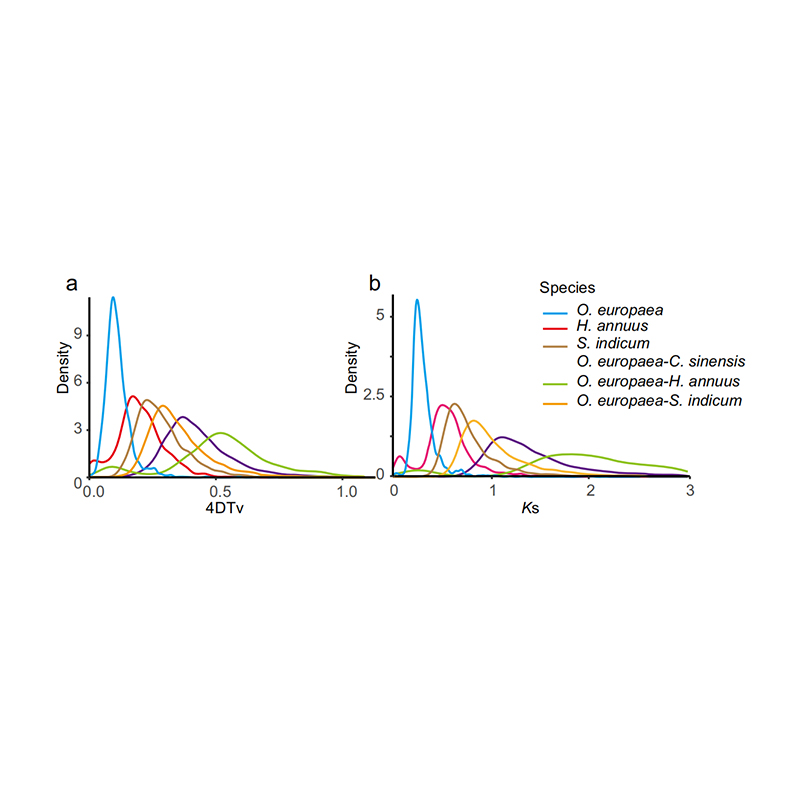
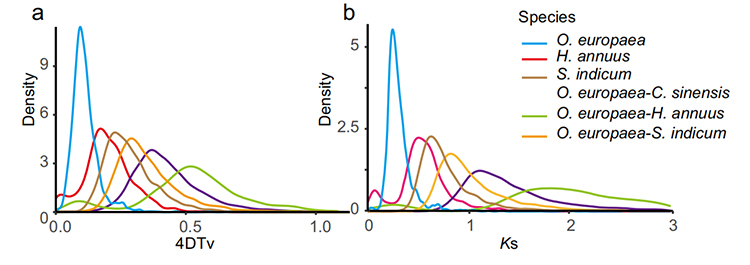
5. การวิเคราะห์การทำสำเนาจีโนมแบบ whole: การวิเคราะห์การกระจาย 4DTV และ KS แสดงเหตุการณ์การทำซ้ำจีโนมทั้งหมด ยอดของ intraspecies แสดงเหตุการณ์การทำซ้ำ ยอดของ interspecies แสดงเหตุการณ์ speciation การวิเคราะห์ชี้ให้เห็นว่าเมื่อเปรียบเทียบกับอีกสามสายพันธุ์ที่เกี่ยวข้องอย่างใกล้ชิด O. Europaea ผ่านการทำซ้ำของยีนขนาดใหญ่เมื่อเร็ว ๆ นี้

Rao G et al.การวิจัยพืชสวน, 2021
กรณี BMK
Rose Without Prickle: ข้อมูลเชิงลึกของจีโนมเชื่อมโยงกับการปรับความชื้น
ที่ตีพิมพ์: รีวิววิทยาศาสตร์แห่งชาติ, 2021
กลยุทธ์การเรียงลำดับ:
'Baseye'sไม่มีหนาม'(อาร์Wichurainan) จีโนม:
ประมาณ 93 x Pacbio + ประมาณ 90 x nanopore + 267 x Illumina
ผลลัพธ์ที่สำคัญ
1. จีโนมที่มีคุณภาพสูง R.Wichuraiana ถูกสร้างขึ้นโดยใช้เทคนิคการเรียงลำดับที่อ่านมานานซึ่งให้การประกอบ 530.07 MB (ขนาดจีโนมโดยประมาณประมาณ 525.9 MB โดยการไหลของไซโตเมทรีและ 525.5 โดยการสำรวจจีโนม; heterozygosity ประมาณ 1.03%) คะแนนโดยประมาณของ Busco คือ 93.9% เมื่อเปรียบเทียบกับ“ Blush เก่า” (Haploob) คุณภาพและความสมบูรณ์ของจีโนมนี้ได้รับการยืนยันโดยความแม่นยำฐานฐานเดี่ยวและดัชนีแอสเซมบลี LTR (LAI = 20.03) จีโนม R.Wichuraiana มียีนการเข้ารหัสโปรตีน 32,674 ยีน
2. การวิเคราะห์ร่วมกันของ Multi-Omics ซึ่งประกอบด้วยจีโนมเปรียบเทียบ, transcriptomics, การวิเคราะห์ QTL ของประชากรทางพันธุกรรม, เผยให้เห็นการเก็งกำไรที่สำคัญระหว่าง R. wichuraiana และ Rosa chinensis นอกจากนี้การเปลี่ยนแปลงการแสดงออกของยีนที่เกี่ยวข้องใน QTL มีแนวโน้มที่จะเกี่ยวข้องกับการสร้างลวดลายหนามของลำต้น

การเปรียบเทียบจีโนม anaysis ระหว่าง basye; s thornless และ rosa chinensis รวมถึงการวิเคราะห์ synteny, กลุ่มตระกูลยีน, การขยายตัวและการวิเคราะห์การหดตัวเผยให้เห็นการเปลี่ยนแปลงจำนวนมากซึ่งเกี่ยวข้องกับลักษณะสำคัญในดอกกุหลาบ การขยายตัวที่ไม่เหมือนใครในตระกูลยีน NAC และ FAR1/FRS มีแนวโน้มที่จะเกี่ยวข้องกับการต่อต้านจุดดำ
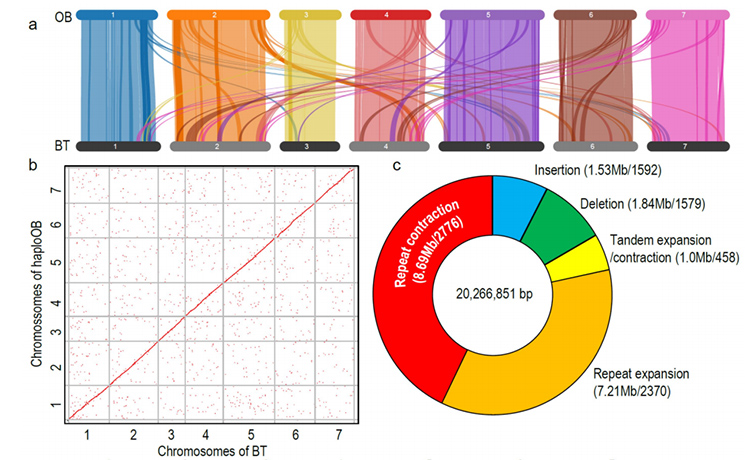

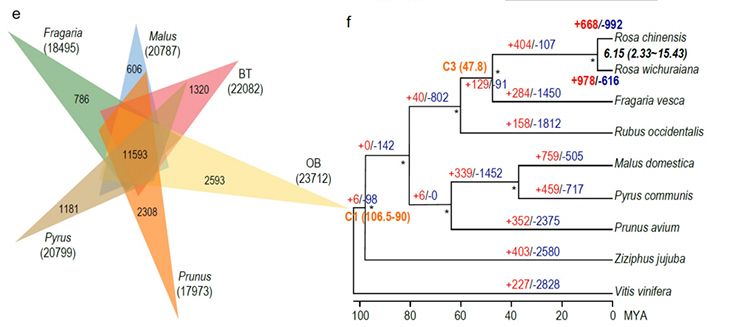
การวิเคราะห์จีโนมเปรียบเทียบระหว่าง BT และ Haploob Genomes
Zhong, M. , et al. “ Rose Without Prickle: ข้อมูลเชิงลึกของจีโนมเชื่อมโยงกับการปรับความชื้น”รีวิววิทยาศาสตร์แห่งชาติ, 2021;, NWAB092