Small RNA Sequencing-Illumina
Drag
● Biblioteksförberedelser inkluderar ett steg för val av storlek
● Bioinformatisk analys centrerad kring miRNA-förutsägelse och deras mål
Servicefördelar
●Omfattande bioinformatikanalys:Möjliggör identifiering av både kända och nya miRNA, identifiering av miRNA-mål och motsvarande funktionella anteckningar och anrikning med flera databaser (KEGG, GO)
●Rigorös kvalitetskontroll: Vi implementerar centrala kontrollpunkter över alla stadier, från prov- och biblioteksberedning till sekvensering och bioinformatik. Denna noggranna övervakning säkerställer leverans av konsekvent högkvalitativa resultat.
●Support efter försäljning: Vårt engagemang sträcker sig längre än att slutföra projekt med en 3-månaders serviceperiod efter försäljning. Under denna tid erbjuder vi projektuppföljning, felsökningshjälp och frågestunder för att ta itu med eventuella frågor relaterade till resultaten.
●Omfattande expertis: Med en meritlista av att framgångsrikt avsluta flera sRNA-projekt som täcker över 300 arter inom olika forskningsdomäner, tillför vårt team en mängd erfarenhet till varje projekt.
Exempel på krav och leverans
| Bibliotek | Plattform | Rekommenderad data | Data QC |
| Storlek vald | Illumina SE50 | 10M-20M avläsningar | Q30≥85 % |
Exempelkrav:
Nukleotider:
| Konc.(ng/μl) | Mängd (μg) | Renhet | Integritet |
| ≥ 80 | ≥ 0,8 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Begränsad eller ingen protein- eller DNA-kontamination visas på gelen. | RIN≥6,0; 5,0≥28S/18S≥1,0; begränsad eller ingen baslinjehöjning |
● Växter:
Rot, stjälk eller kronblad: 450 mg
Blad eller frö: 300 mg
Frukt: 1,2 g
● Djur:
Hjärta eller tarm: 450 mg
Inälvor eller hjärna: 240 mg
Muskel: 600 mg
Ben, hår eller hud: 1,5 g
● Leddjur:
Insekter: 9g
Kräftdjur: 450 mg
● Helblod: 2 rör
● Celler: 106 celler
● Serum och plasma:6 ml
Rekommenderad provleverans
Behållare: 2 ml centrifugrör (tunnfolie rekommenderas inte)
Provmärkning: Grupp+replikat t.ex. A1, A2, A3; B1, B2, B3.
Transport:
1. Torris: Proverna måste packas i påsar och grävas ner i torris.
2. RNAstabila rör: RNA-prover kan torkas i RNA-stabiliseringsrör (t.ex. RNAstable®) och skickas i rumstemperatur.
Service Arbetsflöde

Experimentdesign

Provleverans

RNA-extraktion

Byggande av bibliotek

Sekvensering

Dataanalys

Service efter försäljning
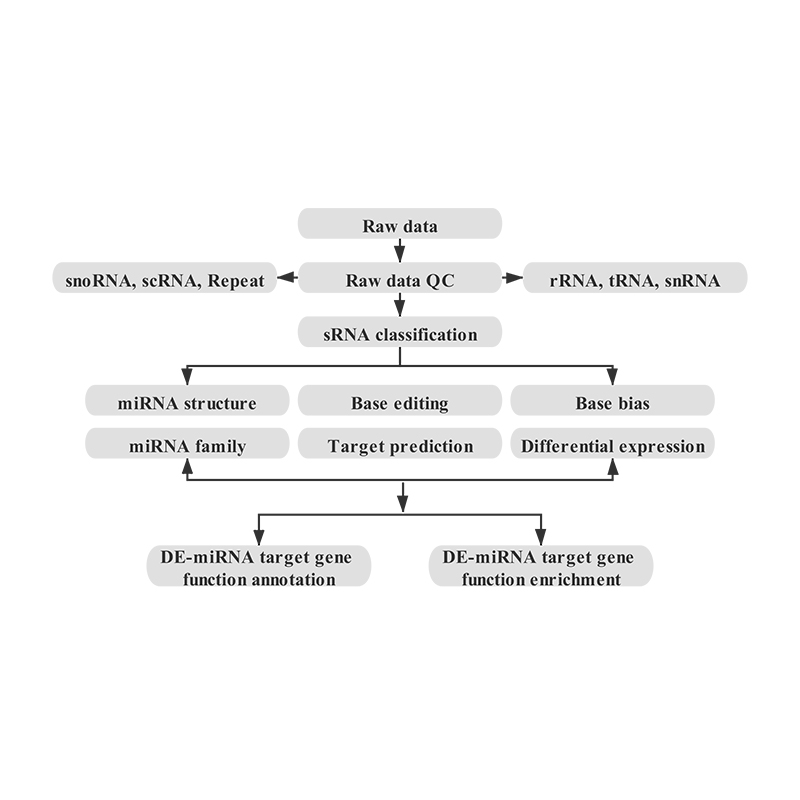
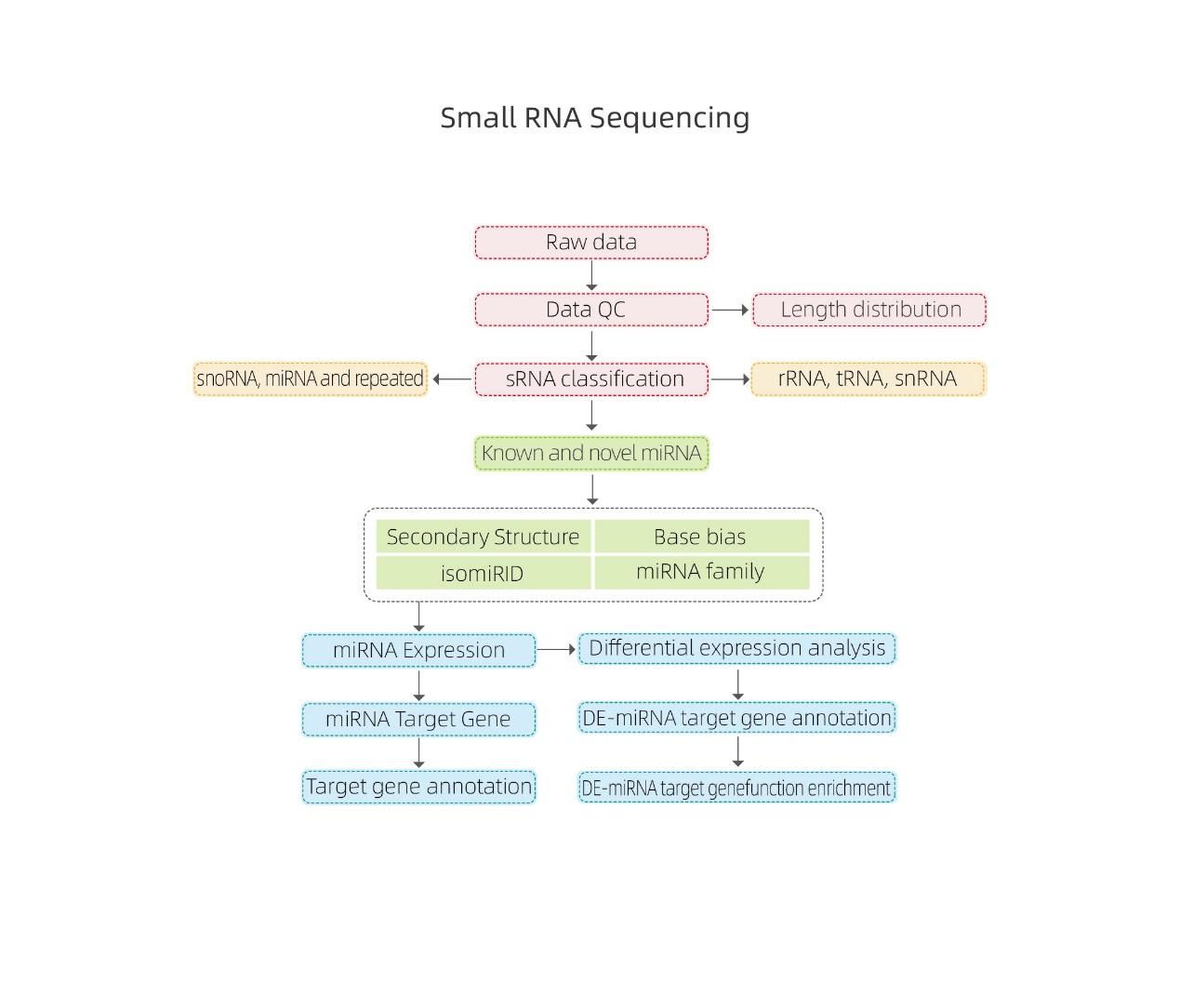
Bioinformatik
● sRNA-klassificering
● Anpassning till ett referensgenom
● Identifiering av känt och nytt miRNA
● Differentiell miRNA-expressionsanalys
● Funktionell annotering av miRNA-mål
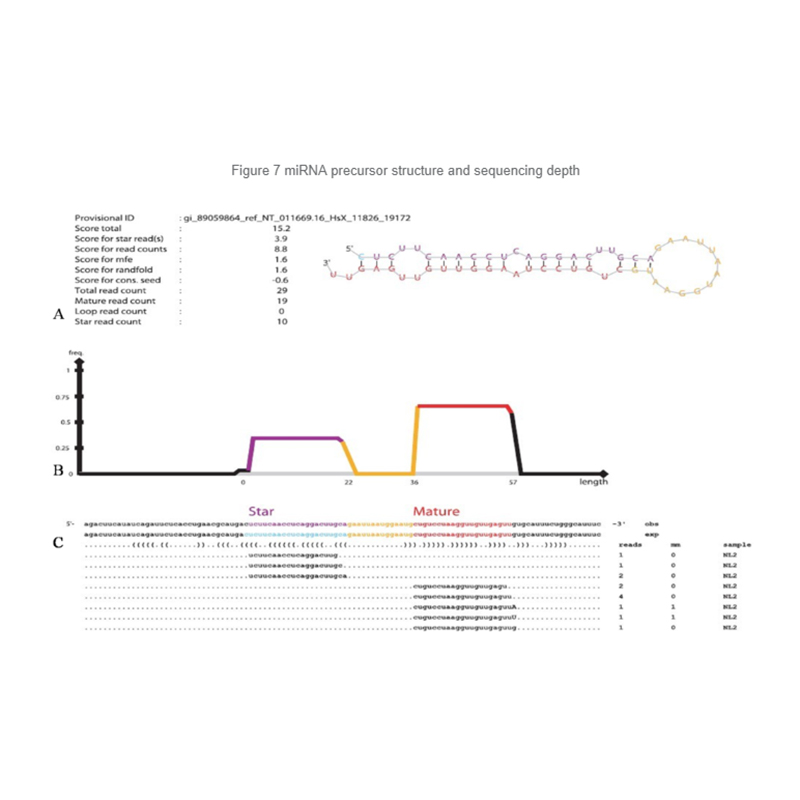
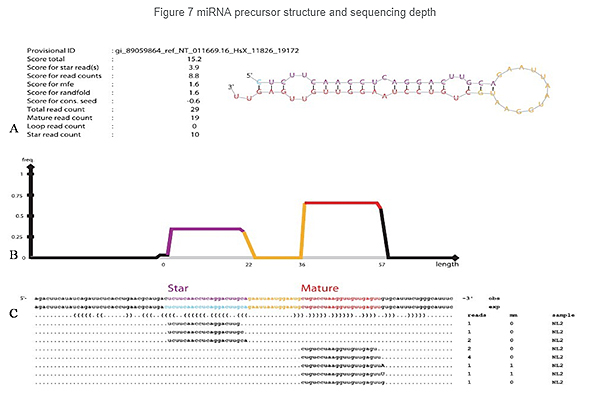
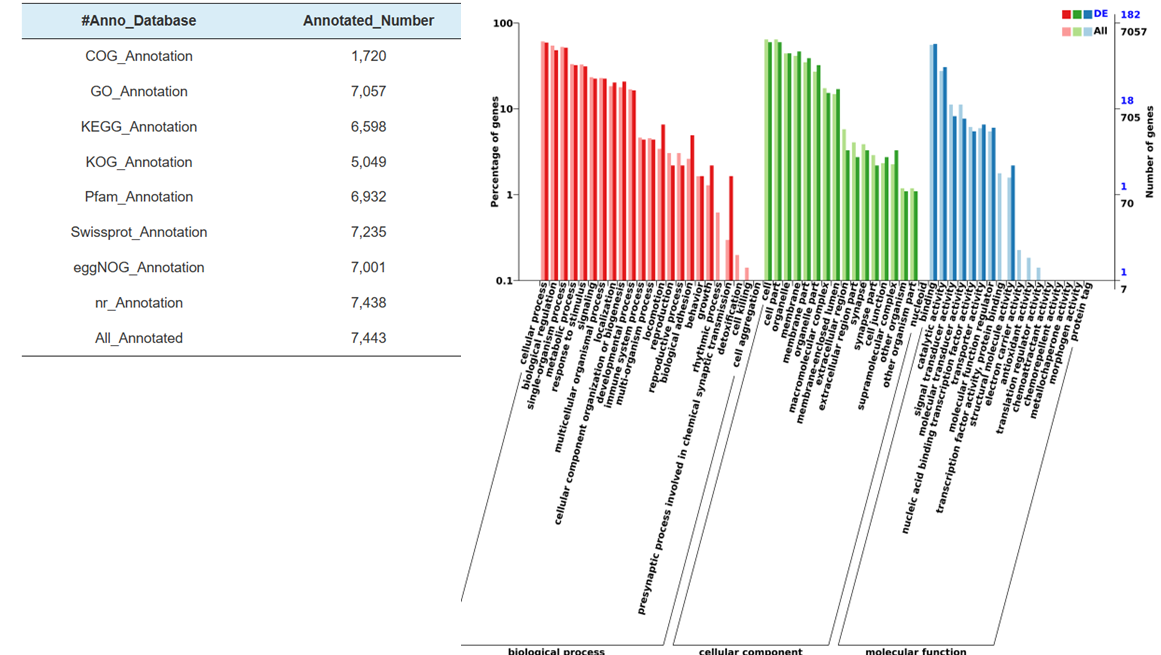
Identifiering av miRNA: struktur och djup
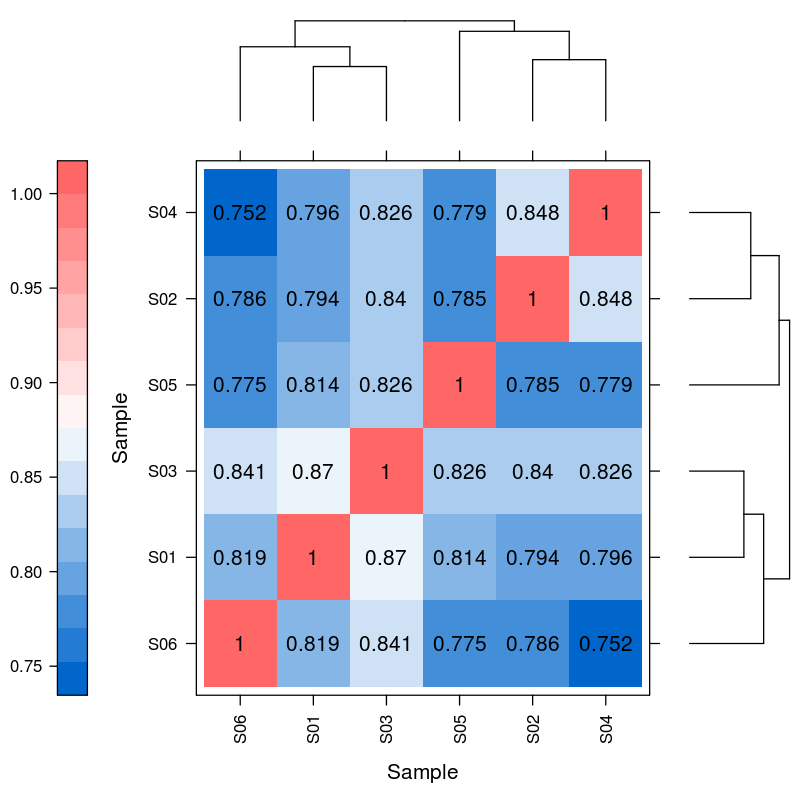
Differentiellt uttryck av miRNA – hierarkisk klustring
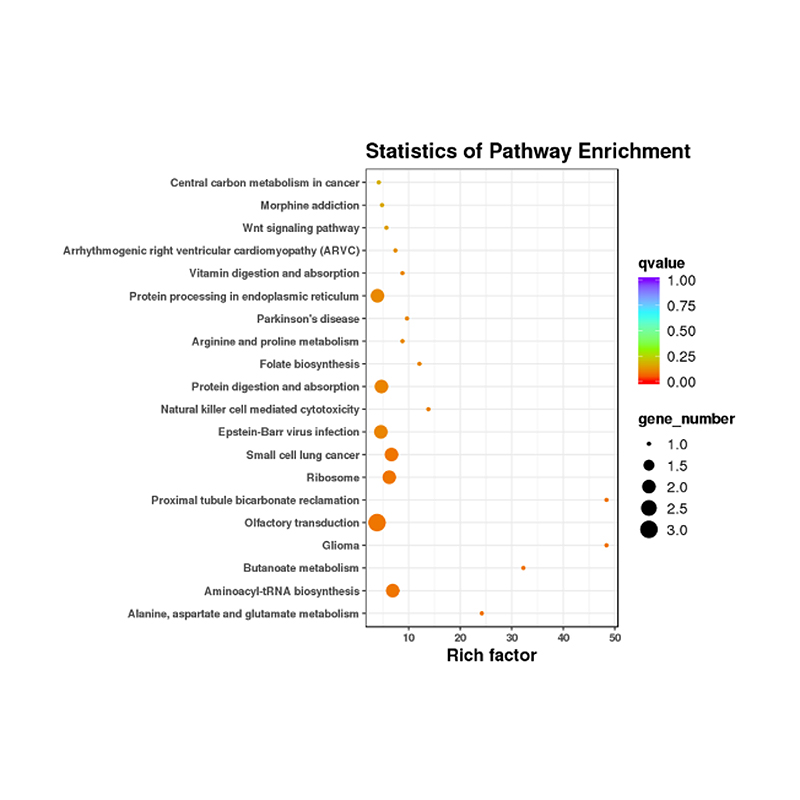
Funktionell annotering av mål för differentiellt uttryckta miRNA
Utforska forskningsframstegen som underlättas av BMKGene's sRNA-sekvenseringstjänster genom en kurerad samling publikationer.
Chen, H. et al. (2023) 'Viralinfektioner hämmar saponinbiosyntes och fotosyntes i Panax notoginseng', Plant Physiology and Biochemistry, 203, sid. 108038. doi: 10.1016/J.PLAPHY.2023.108038.
Li, H. et al. (2023) "Växten FYVE-domäninnehållande protein FREE1 associerar med mikroprocessorkomponenter för att undertrycka miRNA-biogenes", rapporterar EMBO, 24(1). doi: 10.15252/EMBR.202255037/SUPPL_FILE/EMBR202255037-SUP-0004-SDATAFIG4.TIF.
Yu, J. et al. (2023) "MicroRNA Ame-Bantam-3p kontrollerar utvecklingen av larverpupparna genom att rikta in sig på genen för flera epidermala tillväxtfaktorliknande domäner 8 (megf8) i honungsbiet, Apis mellifera", International Journal of Molecular Sciences, 24(6), s. . 5726. doi: 10.3390/IJMS24065726/S1.
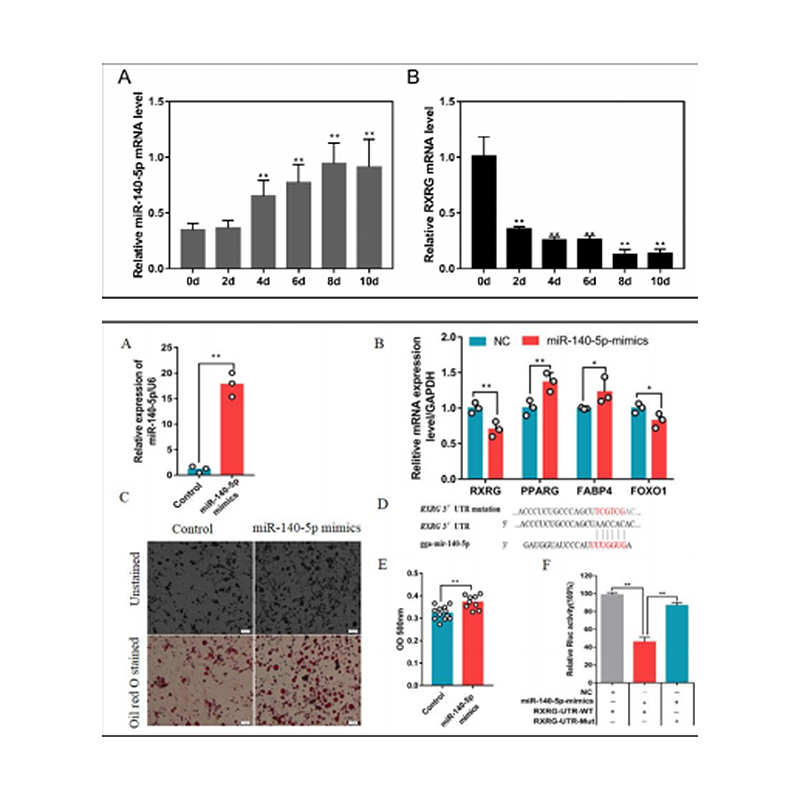
Zhang, M. et al. (2018) 'Integrerad analys av MiRNA och gener associerade med köttkvalitet avslöjar att Gga-MiR-140-5p påverkar intramuskulär fettavlagring hos kycklingar', Cellular Physiology and Biochemistry, 46(6), s. 2421–2433. doi: 10.1159/000489649.