-

Metagenomisk sekvensering -NGS
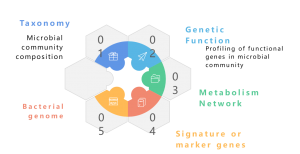
Ett metagenom är en samling av det totala genetiska materialet från en blandad gemenskap av organismer, såsom miljö- och mänskliga metagenomer. Den innehåller genom från både odlingsbara och oodlingsbara mikroorganismer. Hagelgevärs metagenomisk sekvensering med NGS möjliggör studiet av dessa intrikata genomiska landskap inbäddade i miljöprover genom att tillhandahålla mer än taxonomisk profilering, vilket också ger granulära insikter om arternas mångfald, överflödsdynamik och komplexa populationsstrukturer. Utöver taxonomiska studier erbjuder shotgun-metagenomik också ett funktionellt genomikperspektiv, vilket möjliggör utforskning av kodade gener och deras förmodade roller i ekologiska processer. Slutligen bidrar etableringen av korrelationsnätverk mellan genetiska element och miljöfaktorer till en holistisk förståelse av det invecklade samspelet mellan mikrobiella samhällen och deras ekologiska bakgrund. Sammanfattningsvis står metagenomisk sekvensering som ett centralt instrument för att reda ut de genomiska krångligheterna hos olika mikrobiella samhällen, vilket belyser de mångfacetterade förhållandena mellan genetik och ekologi inom dessa komplexa ekosystem.
Plattformar: Illumina NovaSeq och DNBSEQ-T7
-

Metagenomisk sekvensering-TGS
Ett metagenom är en samling av genetiskt material från en blandad gemenskap av organismer, såsom miljö- och mänskliga metagenomer. Den innehåller genom från både odlingsbara och oodlingsbara mikroorganismer. Metagenomisk sekvensering möjliggör studiet av dessa intrikata genomiska landskap inbäddade i ekologiska prover genom att tillhandahålla mer än taxonomisk profilering. Det erbjuder också ett funktionellt genomikperspektiv genom att utforska de kodade generna och deras förmodade roller i miljöprocesser. Medan traditionella hagelgevärsmetoder med Illumina-sekvensering har använts i stor utsträckning i metagenomiska studier, har tillkomsten av Nanopore och PacBio långläst sekvensering förändrat området. Nanopore och PacBio-teknologi förbättrar nedströms bioinformatiska analyser, särskilt metagenommontering, vilket säkerställer mer kontinuerliga sammansättningar. Rapporter indikerar att Nanopore-baserad och PacBio-baserad metagenomik framgångsrikt har genererat kompletta och slutna bakteriegenom från komplexa mikrobiomer (Moss, EL, et al., Nature Biotech, 2020). Att integrera Nanopore-läsningar med Illumina-läsningar ger ett strategiskt tillvägagångssätt för felkorrigering, vilket minskar Nanopores inneboende låga noggrannhet. Denna synergistiska kombination utnyttjar styrkorna hos varje sekvenseringsplattform och erbjuder en robust lösning för att övervinna potentiella begränsningar och förbättra precisionen och tillförlitligheten i metagenomiska analyser.
Plattform: Nanopore PromethION 48, Illumia och PacBio Revio
-

Helgenom-bisulfitsekvensering (WGBS)
Whole Genome Bisulfite Sequencing (WGBS) står som guldstandardmetoden för djupgående utforskning av DNA-metylering, närmare bestämt den femte positionen i cytosin (5-mC), en pivotal regulator av genuttryck och cellulär aktivitet. Principen bakom WGBS involverar bisulfitbehandling, vilket inducerar omvandlingen av ometylerade cytosiner till uracil (C till U), samtidigt som metylerade cytosiner lämnas oförändrade. Denna teknik erbjuder enbasupplösning, vilket gör det möjligt för forskare att omfattande undersöka metylomen och upptäcka onormala metyleringsmönster förknippade med olika tillstånd, särskilt cancer. Genom att använda WGBS kan forskare få oöverträffade insikter i genomomfattande metyleringslandskap, vilket ger en nyanserad förståelse för de epigenetiska mekanismer som ligger till grund för olika biologiska processer och sjukdomar.
-

Analys för transposas-tillgängligt kromatin med hög genomströmningssekvensering (ATAC-seq)
ATAC-seq är en sekvenseringsteknik med hög genomströmning som används för genomomfattande kromatintillgänglighetsanalys. Det ger en djupare förståelse för de komplexa mekanismerna för global epigenetisk kontroll över genuttryck. Metoden använder ett hyperaktivt Tn5-transposas för att samtidigt fragmentera och tagga öppna kromatinregioner genom att infoga sekvenseringsadaptrar. Efterföljande PCR-amplifiering resulterar i skapandet av ett sekvenseringsbibliotek, som möjliggör en omfattande identifiering av öppna kromatinregioner under specifika rum-tidsförhållanden. ATAC-seq ger en holistisk bild av tillgängliga kromatinlandskap, till skillnad från metoder som enbart fokuserar på transkriptionsfaktorbindningsställen eller specifika histonmodifierade regioner. Genom att sekvensera dessa öppna kromatinregioner avslöjar ATAC-seq regioner som är mer benägna att aktiva regulatoriska sekvenser och potentiella transkriptionsfaktorbindningsställen, vilket ger värdefulla insikter om den dynamiska moduleringen av genuttryck över genomet.
-

16S/18S/ITS Amplicon Sequencing-PacBio
16S och 18S rRNA-generna, tillsammans med Internal Transcribed Spacer (ITS)-regionen, fungerar som pivotala molekylära fingeravtrycksmarkörer på grund av deras kombination av mycket konserverade och hypervariabla regioner, vilket gör dem till ovärderliga verktyg för att karakterisera prokaryota och eukaryota organismer. Amplifiering och sekvensering av dessa regioner erbjuder en isoleringsfri metod för att undersöka den mikrobiella sammansättningen och mångfalden över olika ekosystem. Medan Illumina-sekvensering vanligtvis riktar sig till korta hypervariabla regioner som V3-V4 av 16S och ITS1, har det visats att överlägsen taxonomisk annotering kan uppnås genom att sekvensera hela längden av 16S, 18S och ITS. Detta omfattande tillvägagångssätt resulterar i högre procentandelar av exakt klassificerade sekvenser, vilket uppnår en upplösningsnivå som sträcker sig till artidentifiering. PacBios Single-Molecule Real-Time (SMRT) sekvenseringsplattform utmärker sig genom att tillhandahålla mycket exakta långa avläsningar (HiFi) som täcker fullängdsamplikonerna, vilket konkurrerar med precisionen hos Illumina-sekvensering. Denna förmåga tillåter forskare att uppnå en oöverträffad fördel - en panoramautsikt över det genetiska landskapet. Den utökade täckningen höjer avsevärt upplösningen i artanteckningar, särskilt inom bakterie- eller svampsamhällen, vilket möjliggör en djupare förståelse av mikrobiella populationers krångligheter.
-

16S/18S/ITS Amplicon Sequencing-NGS
Amplikonsekvensering med Illumina-teknologi, specifikt inriktad på 16S, 18S och ITS genetiska markörer, är en kraftfull metod för att reda ut fylogenin, taxonomi och artöverflöd inom mikrobiella samhällen. Detta tillvägagångssätt involverar sekvensering av de hypervariabla regionerna av genetiska markörer för hushållning. Ursprungligen introducerades som ett molekylärt fingeravtryck avWoeses et al1977 har denna teknik revolutionerat mikrobiomprofilering genom att möjliggöra isoleringsfria analyser. Genom sekvensering av 16S (bakterier), 18S (svampar) och Internal Transcribed Spacer (ITS, svampar), kan forskare identifiera inte bara rikliga arter utan också sällsynta och oidentifierade. Amplikonsekvensering har blivit allmänt antagen som ett centralt verktyg och har blivit avgörande för att urskilja differentiella mikrobiella sammansättningar i olika miljöer, inklusive människans mun, tarmar, avföring och vidare.
-

Omsekvensering av hela genomet av bakterier och svampar
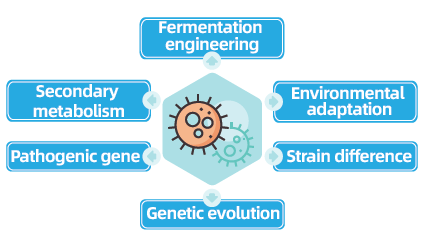
Projekt för återsekvensering av helgenom av bakterier och svamp är avgörande för att främja mikrobiell genomik genom att möjliggöra komplettering och jämförelse av mikrobiella genom. Detta underlättar fermenteringsteknik, optimering av industriella processer och utforskning av sekundära metabolismvägar. Dessutom är omsekvensering av svampar och bakterier avgörande för att förstå miljöanpassning, optimera stammar och avslöja genetisk evolutionsdynamik, med breda implikationer inom medicin, jordbruk och miljövetenskap.
-

PacBio-fullängds 16S/18S/ITS Amplicon Sequencing
Amplicon (16S/18S/ITS) plattform är utvecklad med många års erfarenhet av mikrobiell mångfaldsprojektanalys, som innehåller standardiserad grundläggande analys och personlig analys: grundläggande analys täcker det vanliga analysinnehållet i aktuell mikrobiell forskning, analysinnehållet är rikt och omfattande, och analysresultat presenteras i form av projektrapporter; Innehållet i personlig analys är mångsidigt. Prover kan väljas och parametrar kan ställas in flexibelt i enlighet med den grundläggande analysrapporten och forskningssyfte, för att realisera personliga krav. Windows operativsystem, enkelt och snabbt.
-

PacBio-fullängdstranskriptom (icke-referens)
Med Pacific Biosciences (PacBio) Isoform-sekvenseringsdata som indata, kan den här appen identifiera transkriptionssekvenser i full längd (utan montering). Genom att kartlägga fullängdssekvenser mot referensgenom kan transkript optimeras av kända gener, transkript, kodande regioner, etc. I detta fall kan mer exakt identifiering av mRNA-strukturer, såsom alternativ splitsning, etc, uppnås. Gemensam analys med NGS-transkriptomsekvenseringsdata möjliggör mer omfattande annotering och mer exakt kvantifiering av uttryck på transkriptnivå, vilket till stor del gynnar nedströms differentiellt uttryck och funktionell analys.
-

Reduced Representation Bisulfite Sequencing (RRBS)
Reduced Representation Bisulfite Sequencing (RRBS) har dykt upp som ett kostnadseffektivt och effektivt alternativ till Whole Genome Bisulfite Sequencing (WGBS) inom DNA-metyleringsforskning. Medan WGBS ger omfattande insikter genom att undersöka hela arvsmassan vid enkelbasupplösning, kan dess höga kostnad vara en begränsande faktor. RRBS mildrar denna utmaning strategiskt genom att selektivt analysera en representativ del av genomet. Denna metod bygger på anrikningen av CpG-örika regioner genom MspI-klyvning följt av storleksval av 200-500/600 bps fragment. Följaktligen sekvenseras endast regioner proximala till CpG-öar, medan de med avlägsna CpG-öar exkluderas från analysen. Denna process, i kombination med bisulfitsekvensering, möjliggör högupplöst detektering av DNA-metylering, och sekvenseringsmetoden, PE150, fokuserar specifikt på ändarna av skären snarare än mitten, vilket ökar effektiviteten av metyleringsprofilering. RRBS är ett ovärderligt verktyg som möjliggör kostnadseffektiv DNA-metyleringsforskning och främjar kunskapen om epigenetiska mekanismer.
-

Prokaryotisk RNA-sekvensering
RNA-sekvensering möjliggör en omfattande profilering av alla RNA-transkript i celler under specifika förhållanden. Denna banbrytande teknologi fungerar som ett kraftfullt verktyg som avslöjar invecklade genuttrycksprofiler, genstrukturer och molekylära mekanismer förknippade med olika biologiska processer. RNA-sekvensering, som är allmänt antagen inom grundforskning, klinisk diagnostik och läkemedelsutveckling, ger insikter i krångligheterna med cellulär dynamik och genetisk reglering. Vår prokaryota RNA-provbearbetning är skräddarsydd för prokaryota transkriptomer, som involverar rRNA-utarmning och riktningsbiblioteksberedning.
Plattform: Illumina NovaSeq
-

Metatranskriptomsekvensering
Med hjälp av Illuminas sekvenseringsteknologi avslöjar BMKGENEs metatranskriptomsekvenseringstjänst det dynamiska genuttrycket av en mångfald av mikrober, som sträcker sig över eukaryoter till prokaryoter och virus, inom naturliga miljöer som jord, vatten, hav, avföring och tarmarna. Vår omfattande tjänst ger forskare möjlighet att fördjupa sig i komplexa mikrobiella samhällens fullständiga genuttrycksprofiler. Utöver taxonomisk analys underlättar vår metatranskriptomsekvenseringstjänst utforskning av funktionell anrikning, och belyser differentiellt uttryckta gener och deras roller. Upptäck en mängd biologiska insikter när du navigerar i de komplexa landskapen av genuttryck, taxonomisk mångfald och funktionell dynamik inom dessa olika miljönischer.
-

De novo Fungal Genome Assembly
BMKGENE erbjuder mångsidiga lösningar för svampgenom, som tillgodoser olika forskningsbehov och önskad genomkompletthet. Att använda enbart kortläst Illumina-sekvensering möjliggör generering av ett utkast till genom. Kortläsningar och långlästa sekvensering med Nanopore eller Pacbio kombineras för ett mer förfinat svampgenom med längre sammansättningar. Integrering av Hi-C-sekvensering förbättrar dessutom kapaciteten ytterligare, vilket möjliggör uppnåendet av ett fullständigt kromosomnivågenom.