Mänsklig hela exome -sekvensering
Servicefunktioner
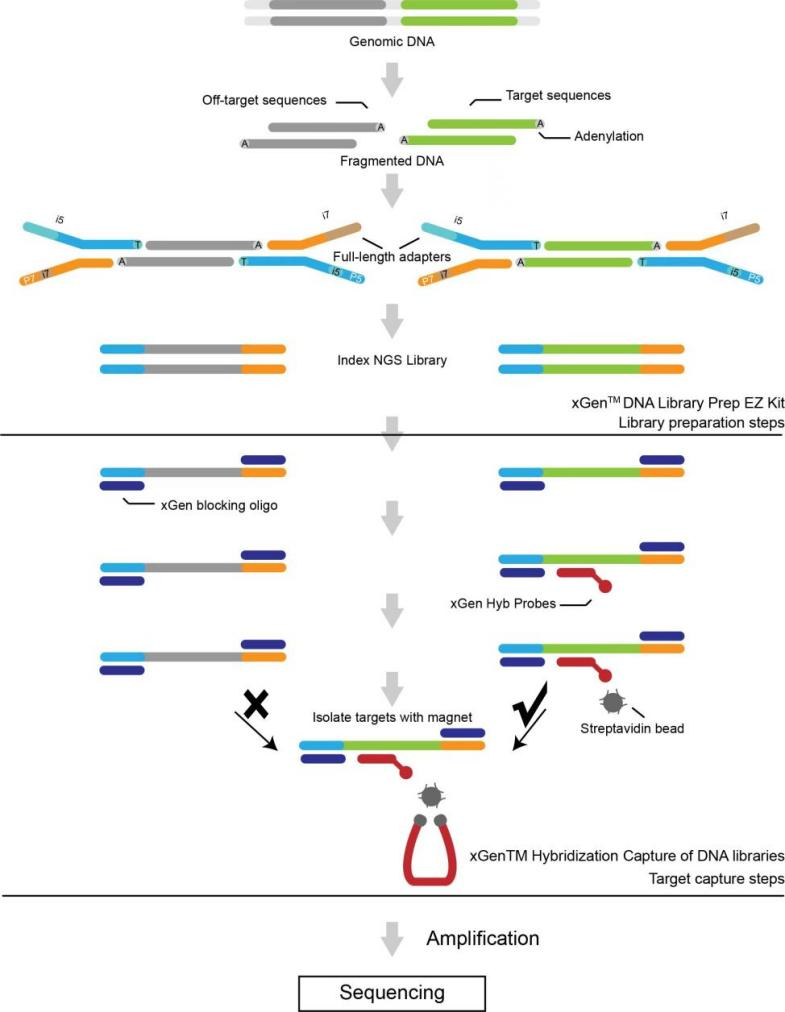
● Två exome -paneler tillgängliga baserade på målberikning med sonder: Sure Select Human All Exon V6 (Agilent) och Xgen Exome Hybridization Panel V2 (IDT).
● Sekvensering på Illumina Novaseq.
● Bioinformatisk rörledning riktad mot sjukdomsanalys eller tumöranalys.
Servicefördelar
●Mål från proteinkodningsregionen: Genom att fånga och sekvensera proteinkodningsregioner används HWES för att avslöja varianter relaterade till proteinstruktur.
●Kostnadseffektivt:HWE: er ger cirka 85% av de mänskliga sjukdomsassocierade mutationerna från 1% av det mänskliga genomet.
●Hög noggrannhet: Med högt sekvenseringsdjup underlättar HWES upptäckten av både vanliga varianter och sällsynta varianter med frekvenser lägre än 1%.
●Rigorös kvalitetskontroll: Vi implementerar fem kärnkontrollpunkter över alla steg, från prov- och biblioteksförberedelser till sekvensering och bioinformatik. Denna noggranna övervakning säkerställer leverans av resultat av hög kvalitet.
●Omfattande bioinformatikanalys: Vår rörledning går utöver att identifiera variationer till referensgenomet, eftersom det innehåller avancerad analys utformad för att specifikt ta itu med forskningsfrågor relaterade till genetiska aspekter av sjukdomar eller tumöranalys.
●Support efter försäljning:Vårt åtagande sträcker sig utöver projektets slutförande med en 3-månaders serviceperiod efter försäljning. Under denna tid erbjuder vi projektuppföljning, felsökningsassistans och Q & A-sessioner för att hantera eventuella frågor relaterade till resultaten.
Provspecifikationer
| Exon fångststrategi | Sekvenseringsstrategi | Rekommenderad datautgång |
| Sure Select Human All Exon V6 (Agilent) eller xgen exome hybridiseringspanel V2 (IDT)
| Illumina Novaseq PE150 | 5 -10 GB För Mendelian -störningar/sällsynta sjukdomar:> 50x För tumörprover: ≥ 100x |
Provkrav
| Provtyp
| Belopp(Qubit®)
| Koncentration | Volym
| Renhet (NanoDrop ™) |
|
Genomisk DNA
| ≥ 50 ng | ≥ 6 ng/μl | ≥ 15 μl | OD260/280 = 1,8-2,0 Ingen nedbrytning, ingen förorening
|
Bioinformatik

Bioinformatisk analys av HWES-sjukdomsprover inkluderar:
● Sekvenseringsdata QC
● Referensgenomjustering
● Identifiering av SNP: er och indelar
● Funktionell kommentar av SNP: er och indeck

Bioinformatisk analys av tumörprover inkluderar:
● Sekvenseringsdata QC
● Referensgenomjustering
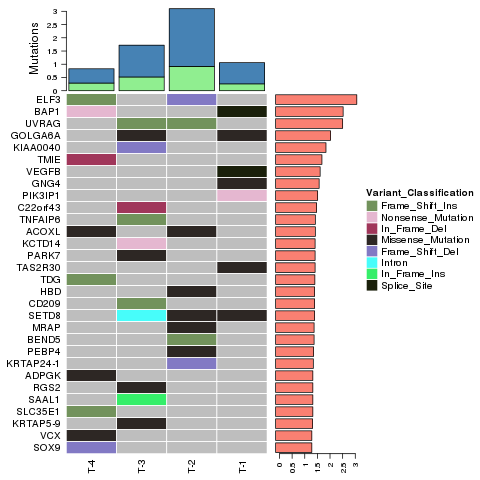
● Identifiering av SNP: er, indelar och somatiska variationer
● Identifiering av bakterievarianter
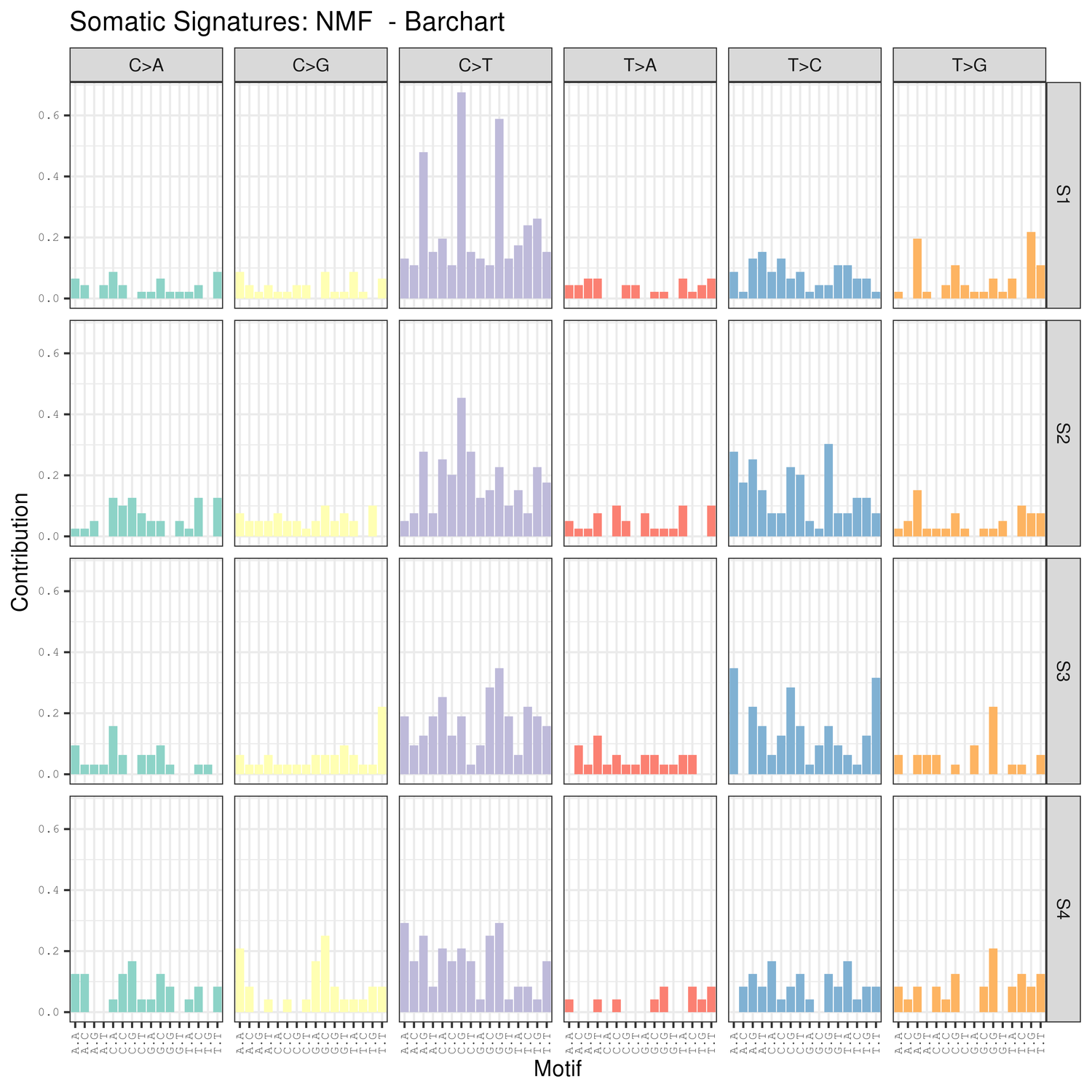
● Analys av mutationssignaturer
● Identifiering av drivgener baserade på förstärkningsmutationer
● Mutation Annotation på nivån av läkemedelskänslighet
● Heterogenitetsanalys - Beräkning av renhet och ploidy
Servicearbetsflöde

Provleverans

DNA -extraktion

Bibliotekskonstruktion

Sekvensering

Dataanalys

Dataleverans

Efterförsäljningstjänster
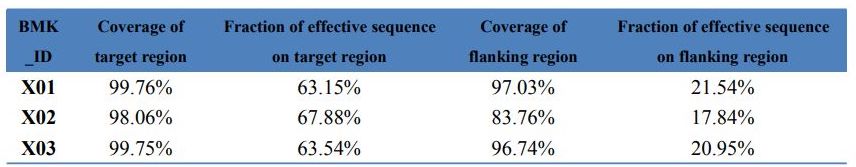
Data QC - Statistik över exome -fångst
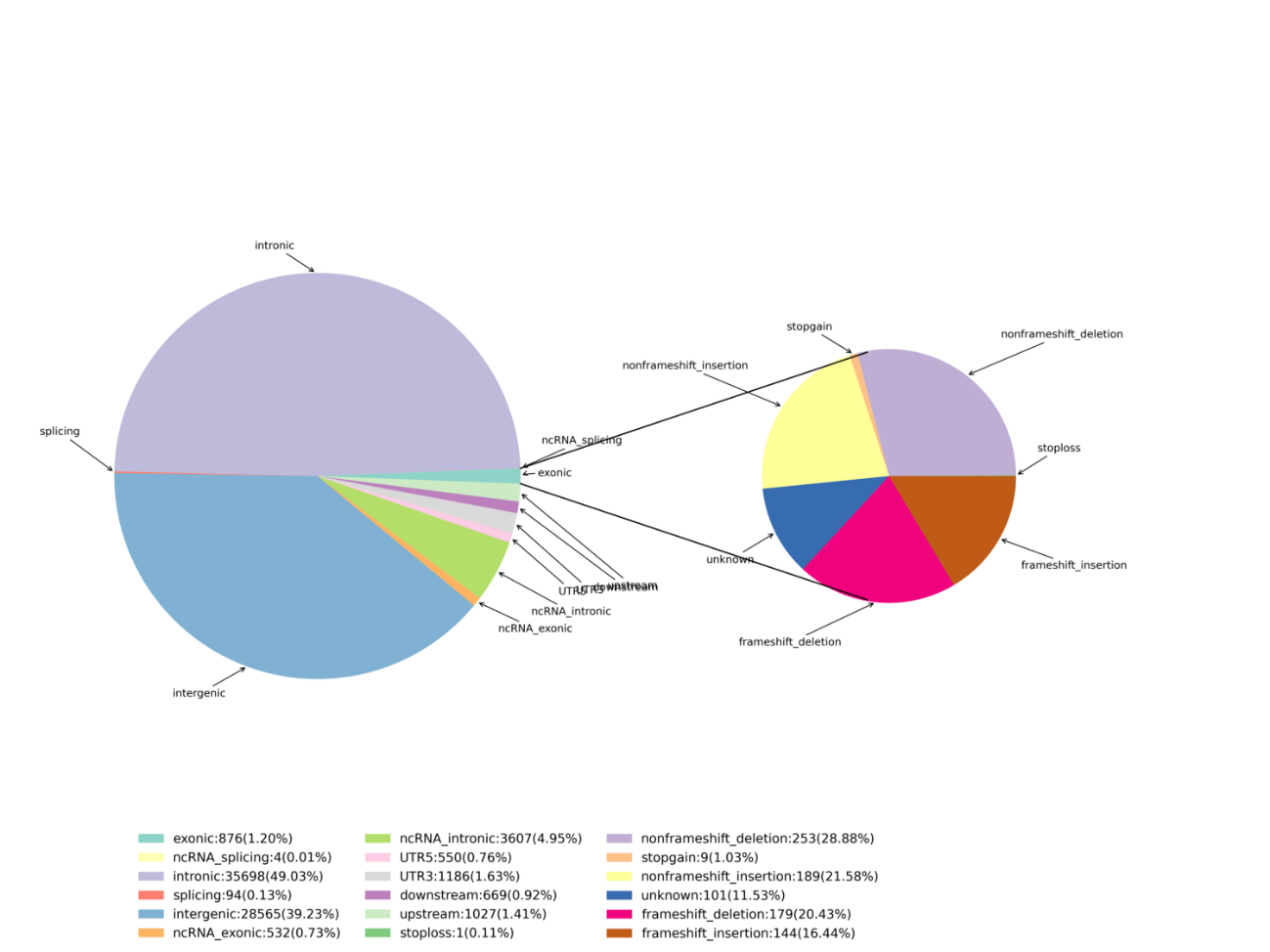
Variantidentifiering - Indels
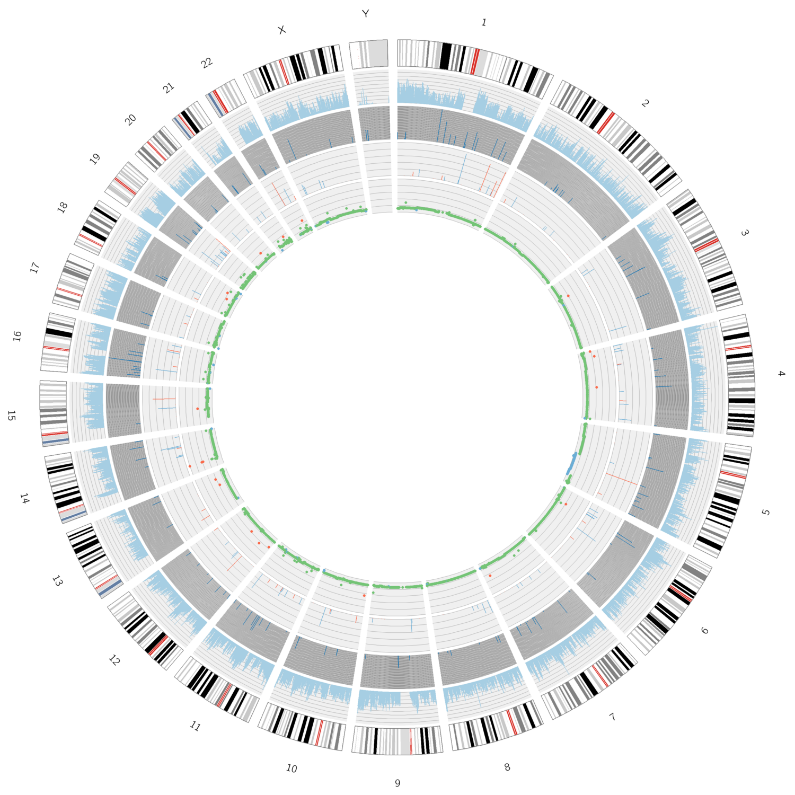
Avancerad analys: Identifiering och distribution av skadliga SNP: er/Indels - Cirkosplott
Tumöranalys: Identifiering och distribution av somatiska mutationer - Circos plot
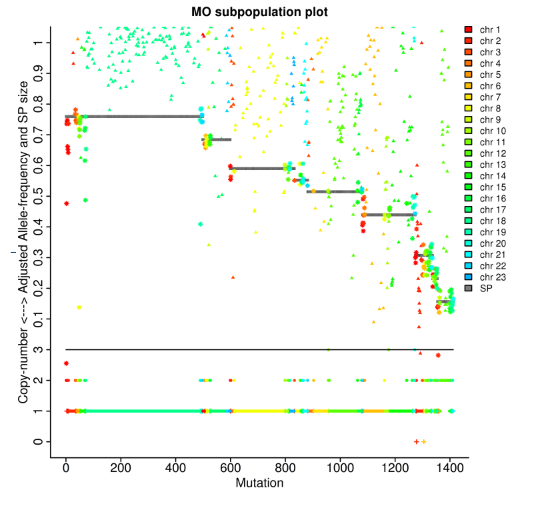
Tumöranalys: Klonala linjer