Hi-C Based Genome Assembly
Servicefunktioner
● Sekvensering på Illumina NovaSeq med PE150.
● Service kräver vävnadsprover, istället för extraherade nukleinsyror, för att tvärbinda med formaldehyd och bevara DNA-proteininteraktionerna.
● Hi-C-experimentet involverar restriktion och slutreparation av de klibbiga ändarna med biotin, följt av cirkularisering av de resulterande trubbiga ändarna samtidigt som interaktionerna bevaras. DNA:t dras sedan ner med streptavidinkulor och renas för efterföljande biblioteksberedning.
Servicefördelar

Översikt över Hi-C
(Lieberman-Aiden E et al.,Vetenskap, 2009)
●Eliminera behovet av genetiska populationsdata:Hi-C ersätter den väsentliga information som krävs för contig förankring.
●Hög markördensitet:vilket leder till ett högt förankringsförhållande över 90 %.
●Omfattande expertis och publikationsregister:BMKGene har stor erfarenhet av mer än 2000 fall av Hi-C Genome Assembly från 1000 olika arter och olika patent. Över 200 publicerade fall har en ackumulerad effektfaktor på över 2000.
●Mycket kompetent bioinformatikteam:med interna patent och mjukvaruupphovsrätt för Hi-C-experiment och dataanalys, möjliggör den egenutvecklade visualiseringsdatamjukvaran manuell blockflyttning, reversering, återkallelse och omställning.
●Support efter försäljning:Vårt engagemang sträcker sig längre än att slutföra projekt med en 3-månaders serviceperiod efter försäljning. Under denna tid erbjuder vi projektuppföljning, felsökningshjälp och frågestunder för att ta itu med eventuella frågor relaterade till resultaten.
●Omfattande anteckning: vi använder flera databaser för att funktionellt kommentera generna med identifierade variationer och utföra motsvarande anrikningsanalys, vilket ger insikter om flera forskningsprojekt.
Servicespecifikationer
| Biblioteksförberedelser | Sekvenseringsstrategi | Rekommenderad datautgång | Kvalitetskontroll |
| Hi-C bibliotek | Illumina NovaSeq PE150 | 100x | Q30 ≥ 85 % |
Exempel på krav
| Vävnad | Erforderligt belopp |
| Djurens inälvor | ≥ 2 g |
| Djurmuskel | |
| Däggdjursblod | ≥ 2 ml |
| Fjäderfä/fiskblod | |
| Växt- färska blad | ≥ 3 g |
| Odlade celler | ≥ 1x107 |
| Insekt | ≥ 2 g |
Service Arbetsflöde

Experimentdesign

Provleverans

Byggande av bibliotek

Sekvensering

Dataanalys

Service efter försäljning
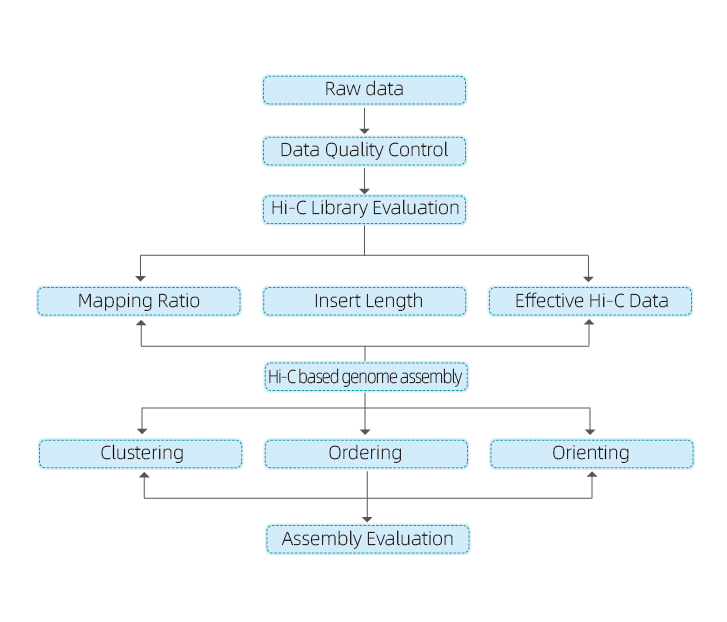
1) Rådata QC
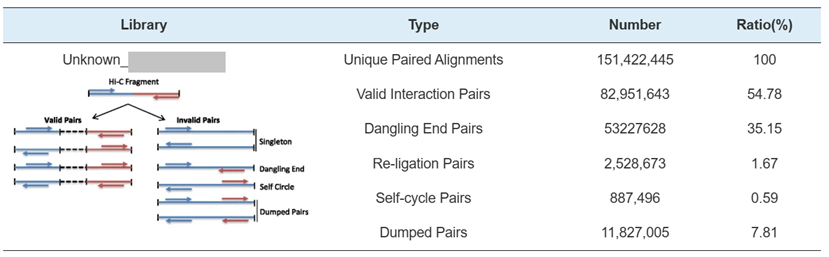
2) Hi-C-bibliotek QC: uppskattning av giltiga Hi-C-interaktioner
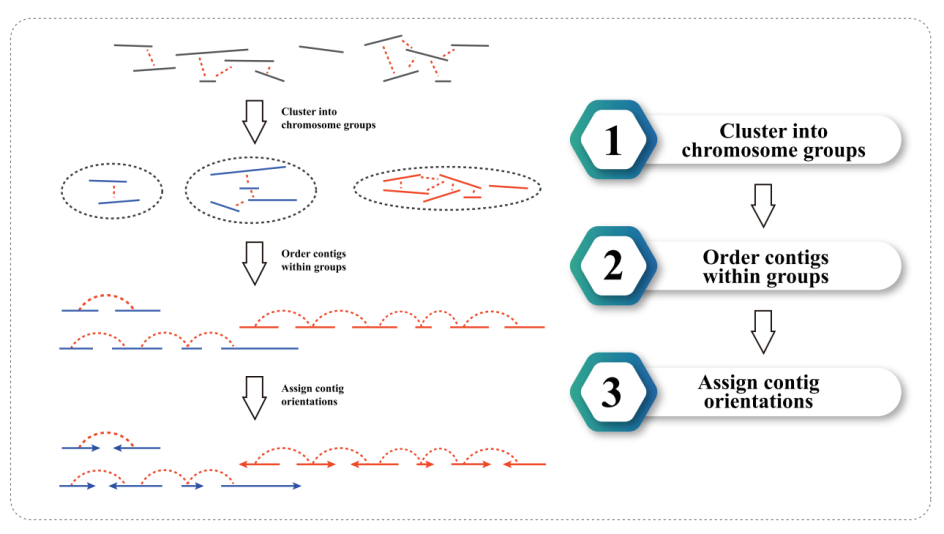
3) Hi-C-montering: klustring av contigs i grupper, följt av contig-ordning inom varje grupp och tilldelning av contig-orientering
4) Hi-C utvärdering
Hi-C Library QC – uppskattning av Hi-C giltiga interaktionspar
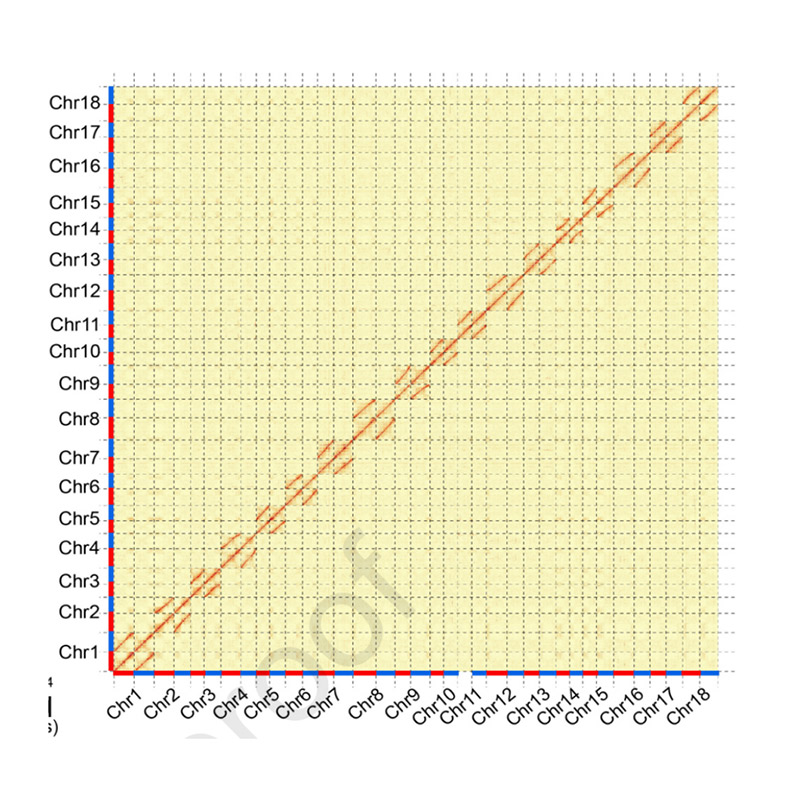
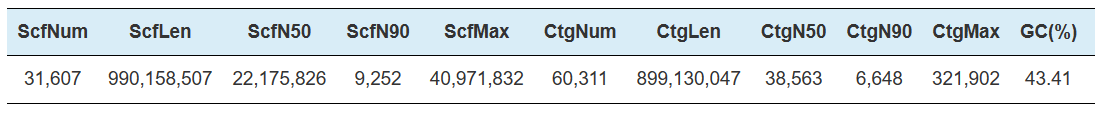
Hi-C Assembly – statistik
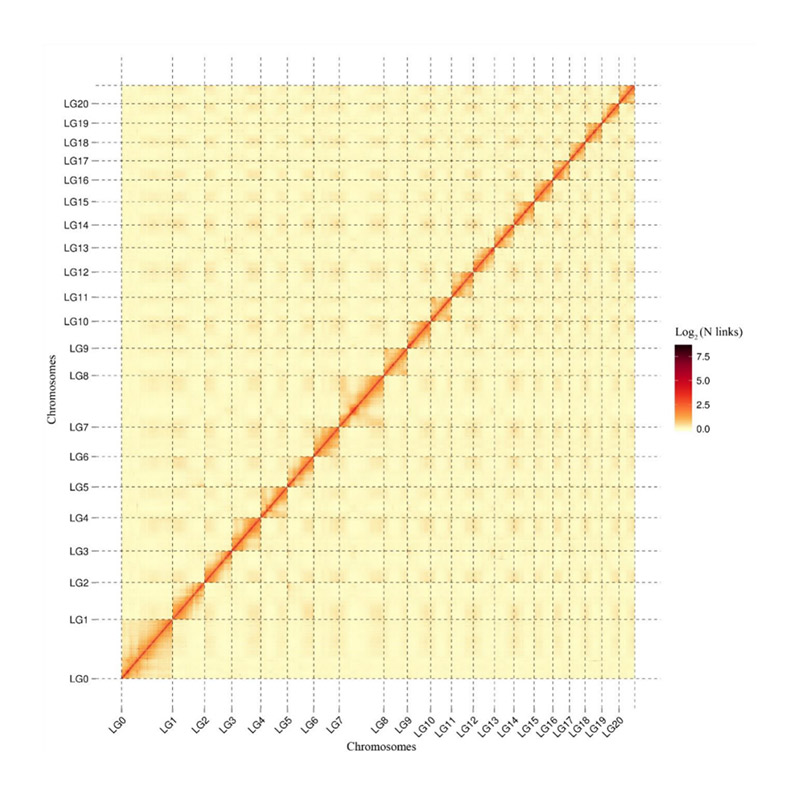
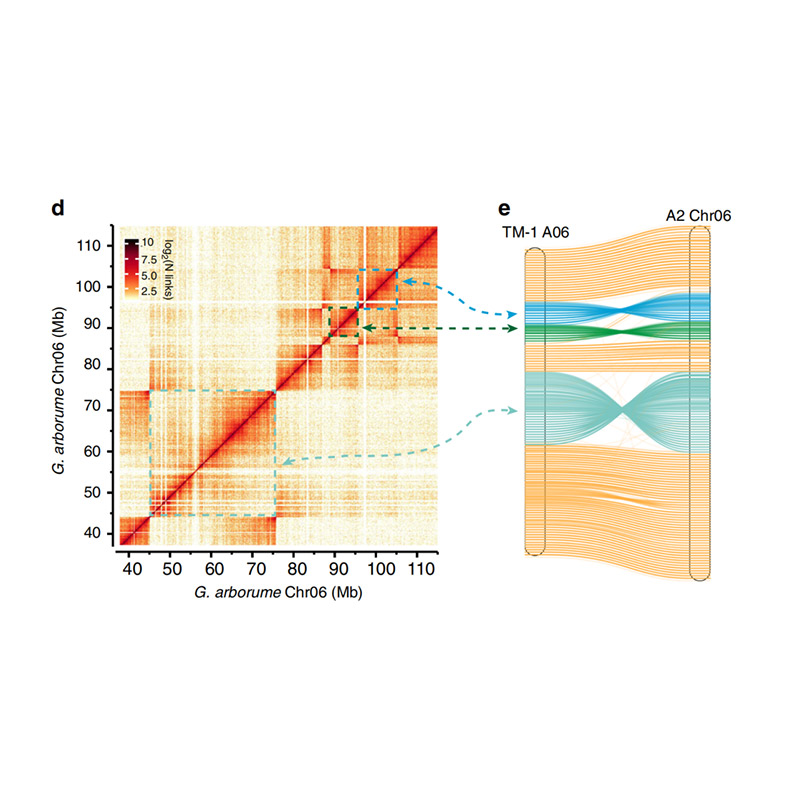
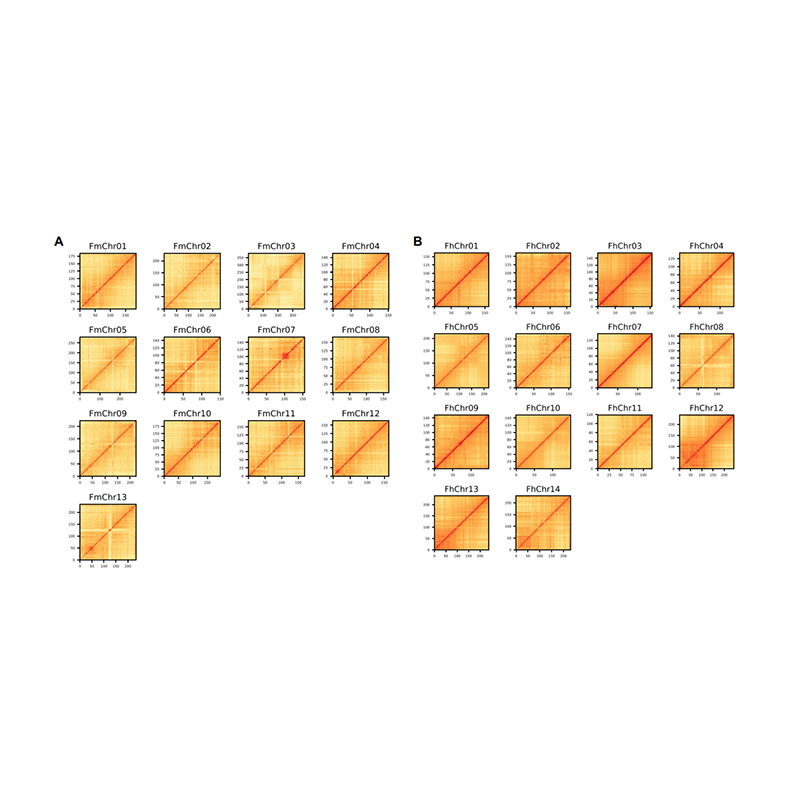
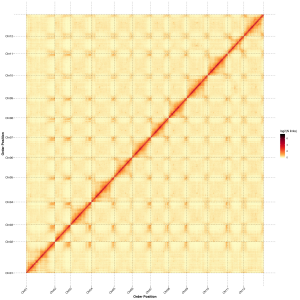
Utvärdering efter montering – värmekarta över signalintensitet mellan fack
Utforska framstegen som underlättas av BMKGenes Hi-C-monteringstjänster genom en utvald samling publikationer.
Tian, T. et al. (2023) 'Genomsammansättning och genetisk dissektion av en framträdande torka-resistent majsgroddplasma', Nature Genetics 2023 55:3, 55(3), s. 496–506. doi: 10.1038/s41588-023-01297-y.
Wang, ZL et al. (2020) 'A Chromosome-Scale Assembly of the Asian Honeybee Apis cerana Genome', Frontiers in Genetics, 11, sid. 524140. doi: 10.3389/FGENE.2020.00279/BIBTEX.
Zhang, F. et al. (2023) 'Avslöjar utvecklingen av tropanalkaloidbiosyntes genom att analysera två genom i familjen Solanaceae', Nature Communications 2023 14:1, 14(1), s. 1–18. doi: 10.1038/s41467-023-37133-4.
Zhang, X. et al. (2020) "Genomer från Banyanträdet och pollinatorgetingen ger insikter i Fig-getingsamverkan", Cell, 183(4), s. 875-889.e17. doi: 10.1016/J.CELL.2020.09.043