Genomomfattande associationsanalys
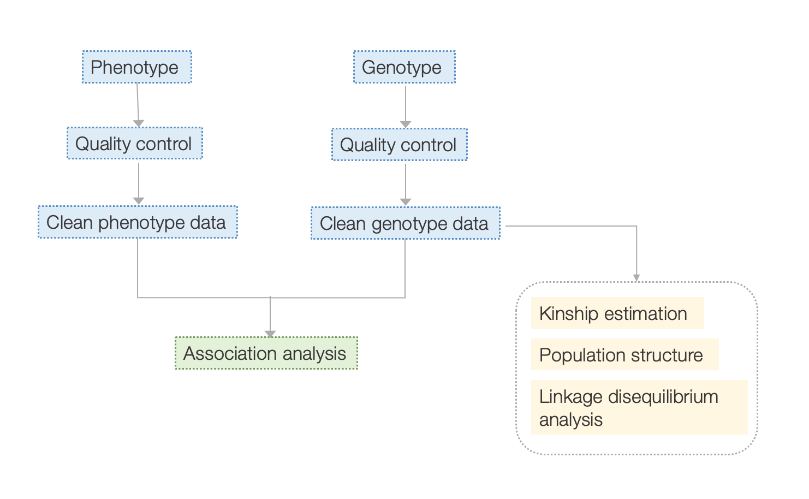
Arbetsflöde

Servicefördelar
●Omfattande expertis och publikationsregister: med samlad erfarenhet av GWAS har BMKGene genomfört hundratals artprojekt inom populations-GWAS-forskning, hjälpt forskare att publicera mer än 100 artiklar och den kumulativa effektfaktorn nådde 500.
● Omfattande bioinformatikanalys: arbetsflöde inkluderar SNP-egenskapsassociationsanalys, som levererar en uppsättning kandidatgener och deras motsvarande funktionella anteckning.
●Mycket kompetent bioinformatikteam och kort analyscykel: med stor erfarenhet av avancerad genomikanalys, levererar BMKGenes team omfattande analyser med en snabb handläggningstid.
●Support efter försäljning:Vårt engagemang sträcker sig längre än att slutföra projekt med en 3-månaders serviceperiod efter försäljning. Under denna tid erbjuder vi projektuppföljning, felsökningshjälp och frågestunder för att ta itu med eventuella frågor relaterade till resultaten.
Servicespecifikationer och krav
| Typ av sekvensering | Rekommenderad befolkningsskala | Sekvenseringsstrategi | Nukleotidkrav |
| Helgenomsekvensering | 200 prover | 10x | Koncentration: ≥ 1 ng/µL Total mängd ≥ 30ng Begränsad eller ingen nedbrytning eller kontaminering |
| Specific-Locus Amplified Fragment (SLAF) | Taggdjup: 10x Antal taggar: < 400 Mb: WGS rekommenderas < 1 Gb: 100 000 taggar 1 Gb > 2Gb: 300K taggar Max 500 000 taggar | Koncentration ≥ 5 ng/µL Total mängd ≥ 80 ng Nanodrop OD260/280=1,6-2,5 Agarosgel: ingen eller begränsad nedbrytning eller kontaminering
|
Materialval



Olika sorter, underarter, lantraser/genbanker/blandade familjer/vilda resurser
Olika sorter, underarter, lantraser
Halvsyskonfamilj/helsyskonfamilj/vilda resurser
Service Arbetsflöde

Experimentdesign

Provleverans

RNA-extraktion

Byggande av bibliotek

Sekvensering

Dataanalys

Service efter försäljning
Innehåller följande analys:
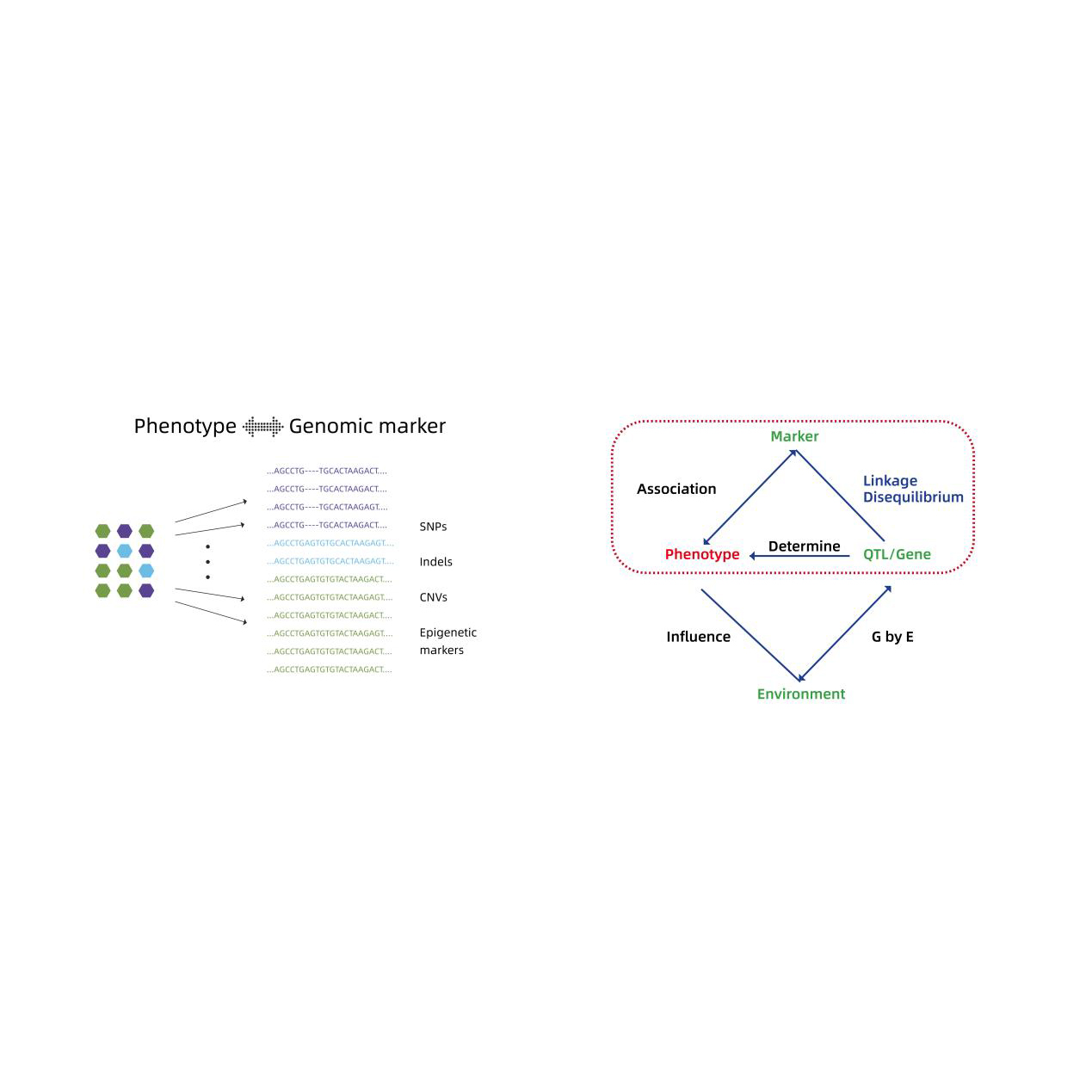
- Genomomfattande associationsanalys: LM, LMM, EMMAX, FASTLMM modell
- Funktionell annotering av kandidatgener
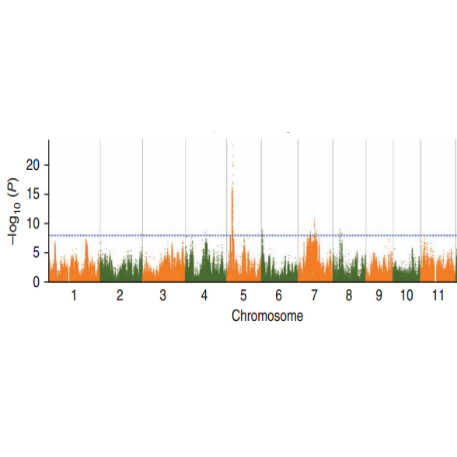
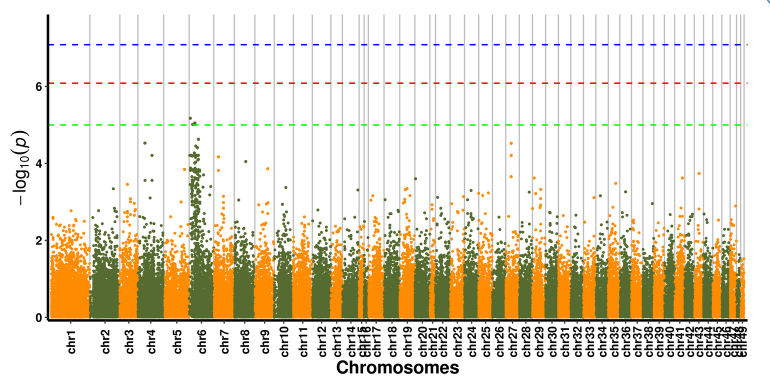
SNP-drag associationsanalys – Manhattan plot
SNP-drag associationsanalys – QQ plot
Utforska framstegen som underlättas av BMKGene's de GWAS-tjänster genom en utvald samling publikationer:
Lv, L. et al. (2023) "Insikt i den genetiska grunden för ammoniaktolerans hos rakmussla Sinonovacula constricta genom genomomfattande associationsstudie",Vattenbruk569, sid. 739351. doi: 10.1016/J.AQUACULTURE.2023.739351.
Li, X. et al. (2022) "Multiomics-analyser av 398 rävsvanshirsaccessioner avslöjar genomiska regioner associerade med domesticering, metabolitegenskaper och antiinflammatoriska effekter",Molekylär växt, 15(8), s. 1367–1383. doi: 10.1016/j.molp.2022.07.003.
Li, J. et al. (2022) 'Genomomfattande associationskartläggning av hulless knappt fenotyper i torkamiljö',Frontiers in Plant Science, 13, sid. 924892. doi: 10.3389/FPLS.2022.924892/BIBTEX.
Zhao, X. et al. (2021) "GmST1, som kodar för ett sulfotransferas, ger resistens mot sojabönmosaikvirusstammarna G2 och G3",Växt, Cell & Miljö, 44(8), s. 2777–2792. doi: 10.1111/PCE.14066.