De novo Fungal Genome Assembly
Servicefunktioner
Med tre möjliga alternativ att välja mellan beroende på önskad grad av genomets fullständighet:
● Draft Genome Option: Kortläst sekvensering med Illumina NovaSeq PE150.
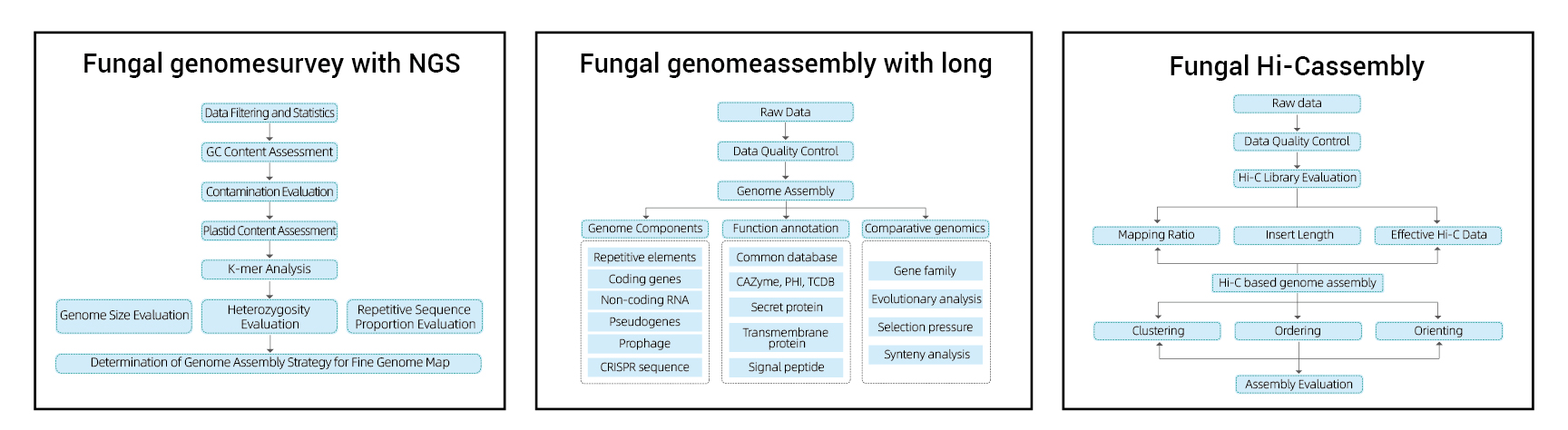
● Alternativ för svampfina genom:
Genomundersökning: Illumina NovaSeq PE150.
Genomförsamling: PacBio Revio (HiFi-läsning) eller Nanopore PromethION 48.
● Svampgenom på kromosomnivå:
Genomundersökning: Illumina NovaSeq PE150.
Genomförsamling: PacBio Revio (HiFi-läsning) eller Nanopore PromethION 48.
Kontig förankring med Hi-C montering.
Servicefördelar
●Flera sekvenseringsstrategier tillgängliga: För olika forskningsmål och krav på genomets fullständighet
●Komplett arbetsflöde för bioinformatik:Detta inkluderar genomsammansättning och förutsägelse av flera genomiska element, funktionell genannotering och contig-förankring.
●Omfattande expertis: Med över 12 000 mikrobiella genom samlade har vi över ett decenniums erfarenhet, ett mycket skickligt analysteam, omfattande innehåll och utmärkt support efter försäljning.
●Support efter försäljning:Vårt engagemang sträcker sig längre än att slutföra projekt med en 3-månaders serviceperiod efter försäljning. Under denna tid erbjuder vi projektuppföljning, felsökningshjälp och frågestunder för att ta itu med eventuella frågor relaterade till resultaten.
Servicespecifikationer
| Service | Sekvenseringsstrategi | Kvalitetskontroll |
| Utkast till genom | Illumina PE150 100x | Q30≥85 % |
| Fint Genom | Genomundersökning: Illumina PE150 50 x Montering: PacBio HiFi 30x eller Nanopore 100x | contig N50 ≥1Mb(Pacbio Unicellular) contig N50 ≥2Mb(ONT encellig) contig N50 ≥500kb(Övriga) |
| Genomet på kromosomnivå | Genomundersökning: Illumina PE150 50 x Montering: PacBio HiFi 30x eller Nanopore 100x Hi-C montering 100x | Contig förankringsförhållande >90 %
|
Servicekrav
| Koncentration (ng/µL) | Total mängd (µg) | Volym (µL) | OD260/280 | OD260/230 | |
| PacBio | ≥20 | ≥2 | ≥20 | 1,7-2,2 | ≥1,6 |
| Nanopore | ≥40 | ≥2 | ≥20 | 1,7-2,2 | 1,0-3,0 |
| Illumina | ≥1 | ≥0,06 | ≥20 | - | - |
Encellig svamp: ≥3,5x1010 celler
Makrosvamp: ≥10 g
Service Arbetsflöde

Provleverans

Byggande av bibliotek

Sekvensering

Dataanalys

Service efter försäljning
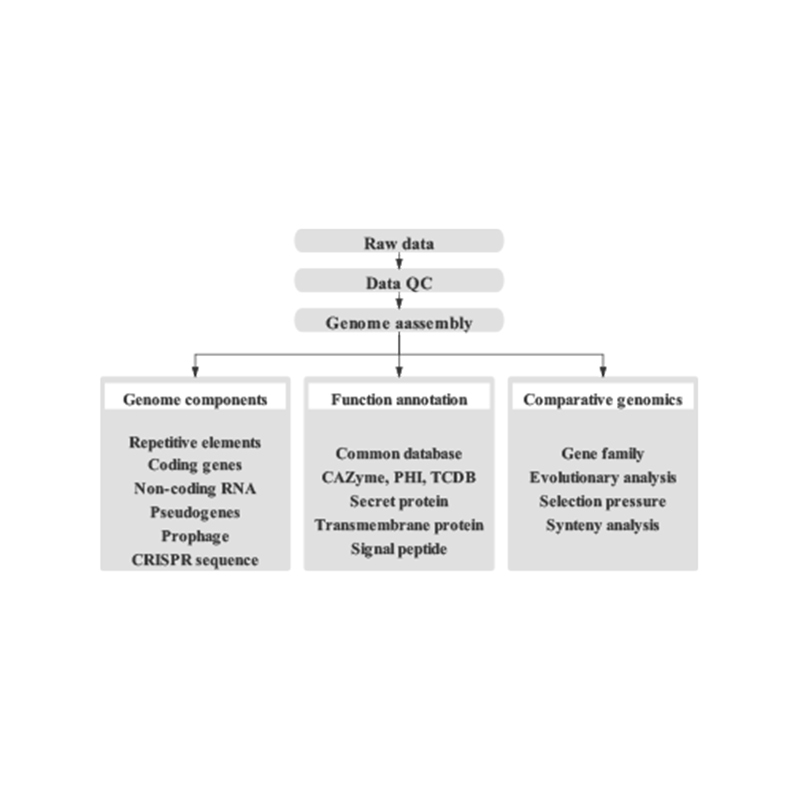
Inkluderar följande analys:
Genomundersökning:
- Sekvenseringsdatakvalitetskontroll
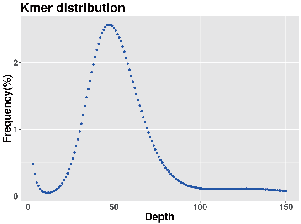
- Genomuppskattning: storlek, heterozygositet, repetitiva element
Fine Genome Assembly:
- Sekvensering av datakvalitetskontroll
- De novoMontering
- Genomkomponentanalys: Förutsägelse av CDS och flera genomiska element
- Funktionell anteckning med flera allmänna databaser (GO, KEGG, etc.) och avancerade databaser (CARD, VFDB, etc.)
Hi-C-montering:
- Hi-C biblioteksbedömning.
- Contigs förankring klustring, ordning och orientering
- Utvärdering av HI-C Assembly: Baserat på referensgenom och värmekarta
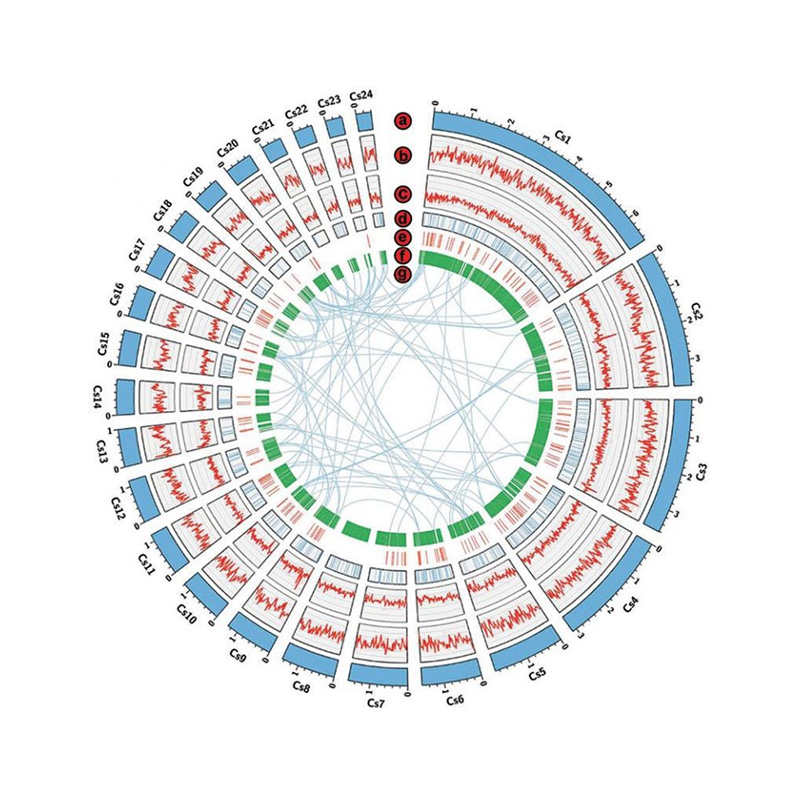
Genomundersökning: k-mer distribution
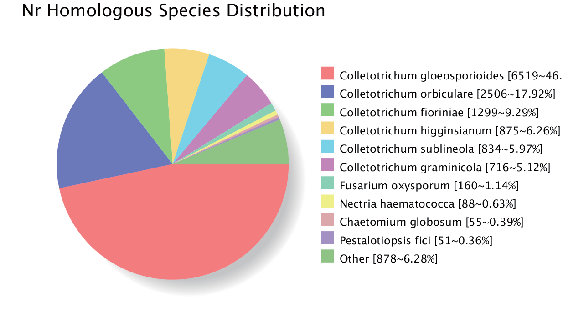
Genomsammansättning: genhomolog annotering (NR-databas)
Genomsammansättning: funktionell genannotering (GO)
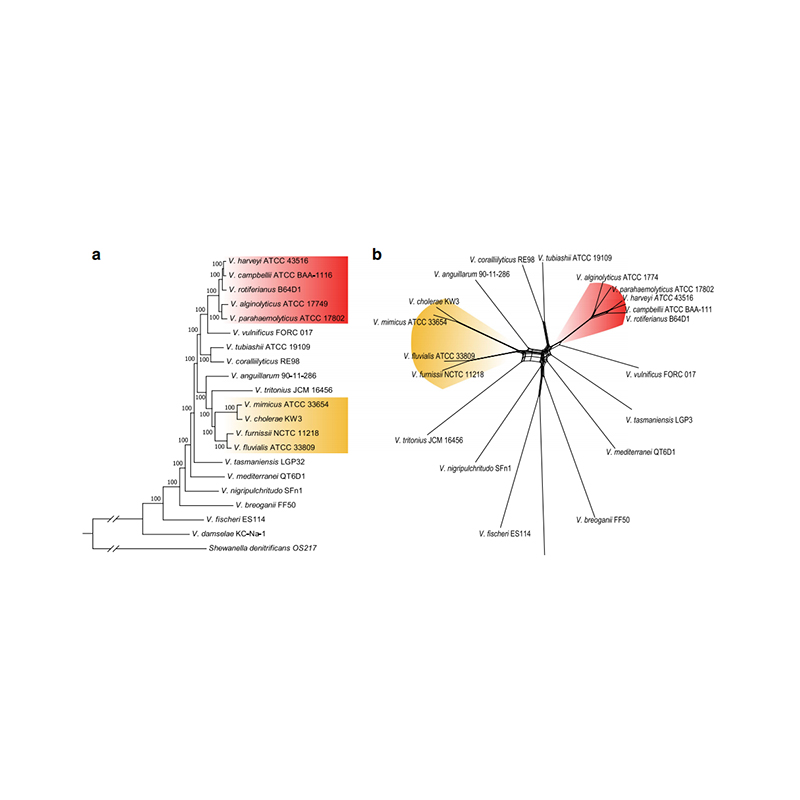
Utforska framstegen som underlättas av BMKGenes svampgenomsättningstjänster genom en utvald samling publikationer.
Hao, J. et al. (2023) "Integrerad omisk profilering av den medicinska svampen Inonotus obliquus under nedsänkta förhållanden",BMC Genomics, 24(1), s. 1–12. doi: 10.1186/S12864-023-09656-Z/FIGURE/3.
Lu, L. et al. (2023) 'Genomsekvensering avslöjar utvecklingen och patogena mekanismerna hos patogenen Rhizoctonia cerealis med vassa ögonfläckar av vete',The Crop Journal, 11(2), s. 405–416. doi: 10.1016/J.CJ.2022.07.024.
Zhang, H. et al. (2023) 'Genomresurser för fyra Clarireedia-arter som orsakar dollarfläckar på olika gräsgräs',Växtsjuka, 107(3), s. 929–934. doi: 10.1094/PDIS-08-22-1921-A
Zhang, SS et al. (2023) "Genetiska och molekylära bevis för ett tetrapolärt parningssystem i den ätliga svampen Grifola frondosa",Journal of Fungi9(10), sid. 959. doi: 10.3390/JOF9100959/S1.