DNBSEQ förgjorda bibliotek
Drag
●Plattform:MGI-DNBSEQ-T7
●Sekvenseringslägen:PE150
●Överföring av Illumina-bibliotek tillMGI:möjliggör sekvensering av stora datamängder till låg kostnad.
●Kvalitetskontroll av bibliotek före sekvensering.
●Sekvenseringsdata QC och leverans:leverans av QC-rapport och rådata i fastq-format efter demultiplexering och filtrering av Q30-läsningar.
Fördelar
●Mångsidighet av sekvenseringstjänster:kunden kan välja att sekvensera efter fil eller mängd data.
●Hög datautgång:1500 Gb/bana
●Leverans av sekvenserings-QC-rapport:med kvalitetsmått, datanoggrannhet och övergripande prestanda för sekvenseringsprojektet.
●Mogen sekvenseringsprocess:med kort handläggningstid.
●Rigorös kvalitetskontroll: vi implementerar strikta kvalitetskontrollkrav för att garantera leverans av konsekvent högkvalitativa resultat.
Exempel på krav
| Datamängd (X) | Koncentration (qPCR/nM) | Volym | |
| Delvis körfält
| X ≤ 10 Gb | ≥ 1nM | ≥ 25 μl |
| 10 Gb < X ≤ 50 Gb | ≥ 2 nM | ≥ 25 μl | |
| 50 Gb < X ≤ 100 Gb | ≥ 3 nM | ≥ 25 μl | |
| X > 100 Gb | ≥ 4 nM | ||
| Enkel körfält | Per Lane | ≥ 1,5 nM / bibliotekspool | ≥ 25 μl / bibliotekspool |
Förutom koncentration och total mängd krävs även ett lämpligt toppmönster.
Obs: Lane-sekvensering av lågdiversitetsbibliotek kräver PhiX spik-in för att säkerställa robusta basanrop.
Vi rekommenderar att du skickar in förpoolade bibliotek som exempel. Om du behöver BMKGENE för att utföra bibliotekspoolning, vänligen se
bibliotekskrav för partiell körfältssekvensering.
Bibliotekets storlek (toppkarta)
Huvudtoppen bör ligga inom 300-450 bp.
Bibliotek bör ha en enda huvudtopp, ingen adapterkontamination och inga primerdimerer.
Service arbetsflöde




Bibliotek QC rapport
En rapport om kvaliteten på biblioteket tillhandahålls före sekvensering, bedömning av biblioteksmängd och fragmentering.
Sekvenserings-QC-rapport
Tabell 1. Statistik över sekvenseringsdata.
| Prov-ID | BMKID | Rå läser | Rådata (bp) | Ren läsning (%) | Q20(%) | Q30(%) | GC(%) |
| C_01 | BMK_01 | 22,870,120 | 6 861 036 000 | 96,48 | 99,14 | 94,85 | 36,67 |
| C_02 | BMK_02 | 14,717,867 | 4,415,360,100 | 96,00 | 98,95 | 93,89 | 37.08 |
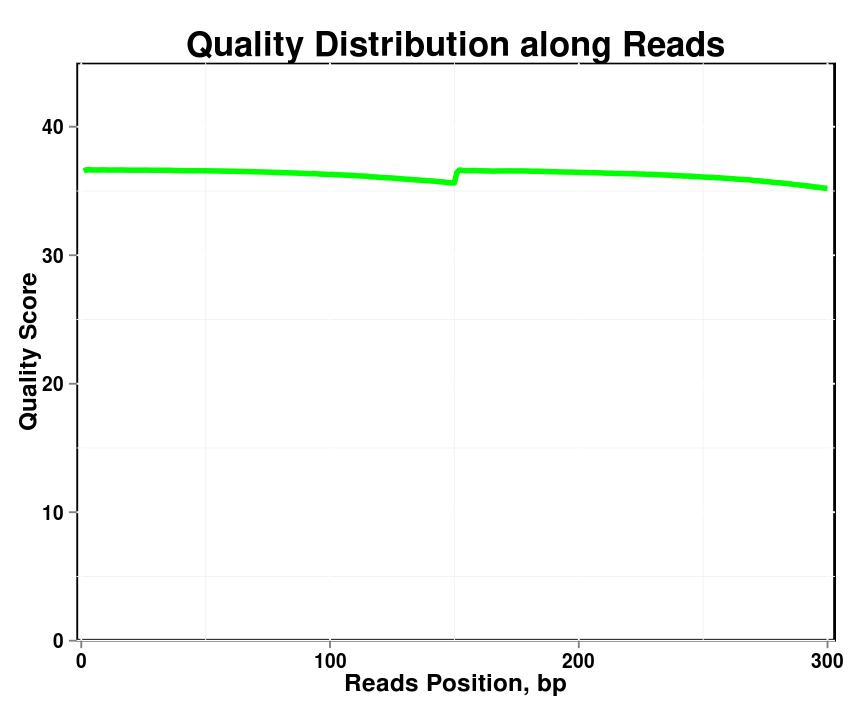
Figur 1. Kvalitetsfördelning längs läsningar i varje prov
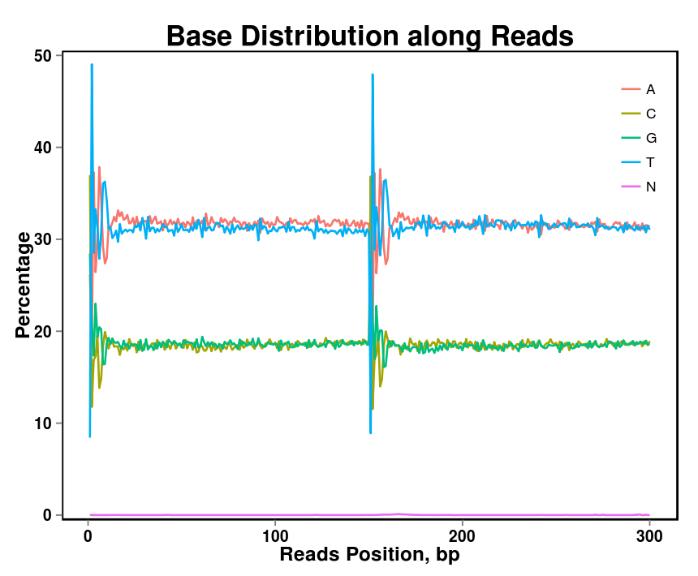
Figur 2. Basinnehållsfördelning
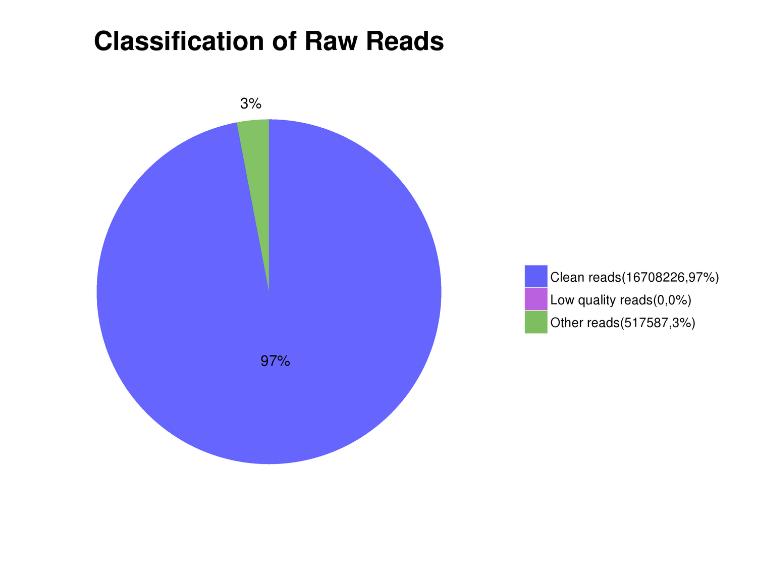
Figur 3. Fördelning av läst innehåll i sekvenseringsdata