BMKMANU S1000 Spatial Transcriptome
BMKMANU S1000 Spatial Transcriptome Technical Scheme
Drag
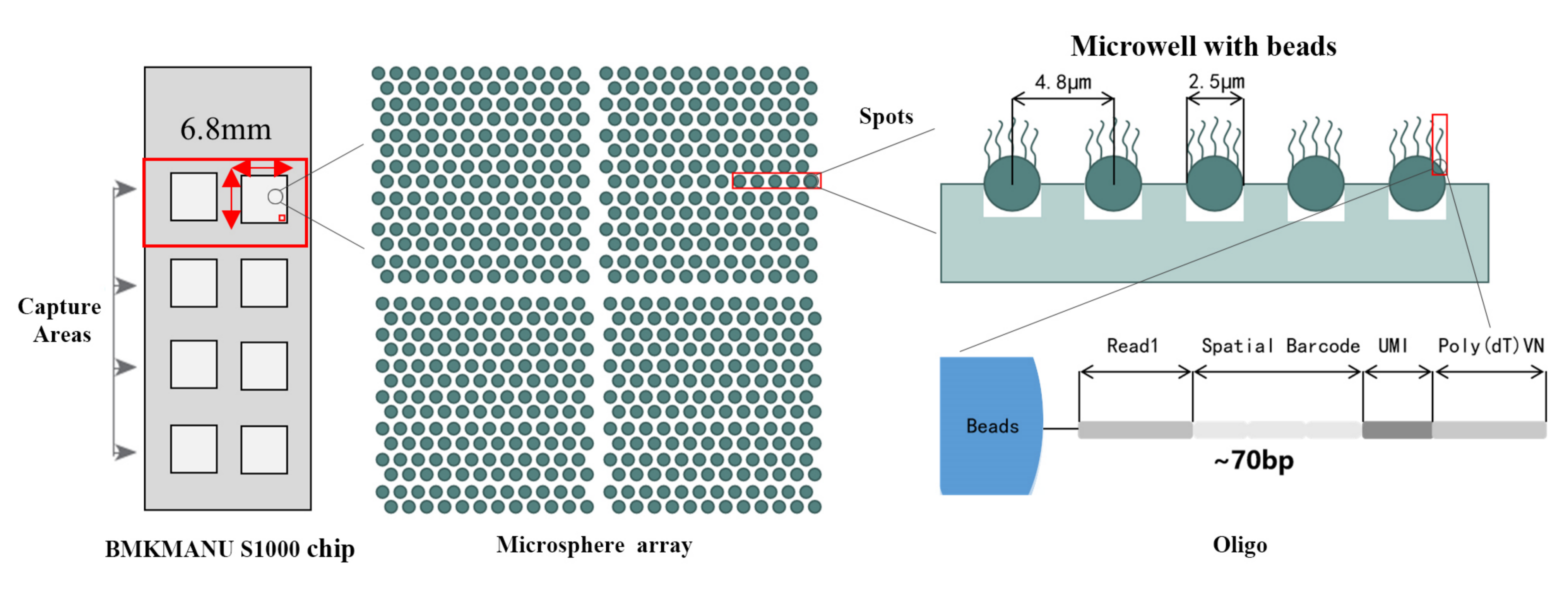
● Upplösning: 5 µM
● Punktdiameter: 2,5 µM
● Antal platser: cirka 2 miljoner
● 3 möjliga inspelningsområdesformat: 6,8 mm * 6,8 mm, 11 mm * 11 mm eller 15 mm * 20 mm
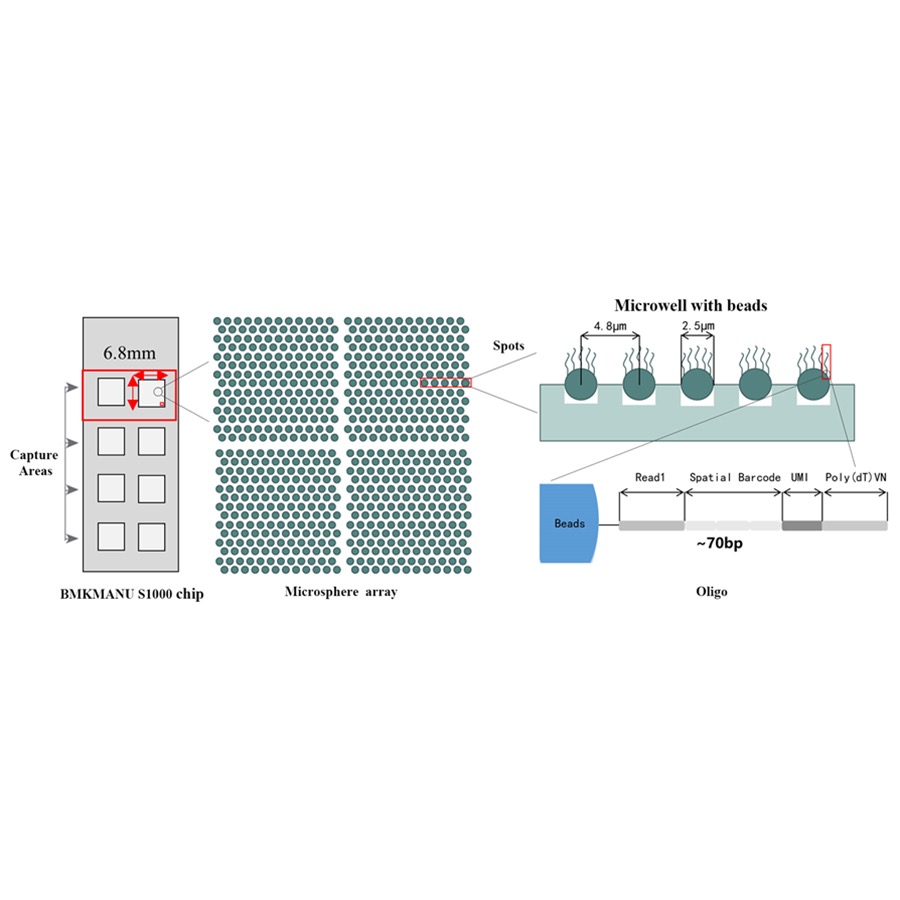
● Varje streckkodad pärla är laddad med primers som består av 4 sektioner:
poly(dT)-svans för mRNA-priming och cDNA-syntes
Unique Molecular Identifier (UMI) för att korrigera amplifieringsbias
Rumslig streckkod
Bindningssekvens för partiell läs 1 sekvenseringsprimer
● H&E och fluorescerande färgning av sektioner
● Möjlighet att användacellsegmenteringsteknik: integration av H&E-färgning, fluorescerande färgning och RNA-sekvensering för att bestämma gränserna för varje cell och korrekt tilldela genuttryck till varje cell.
Fördelar med BMKMANU S1000
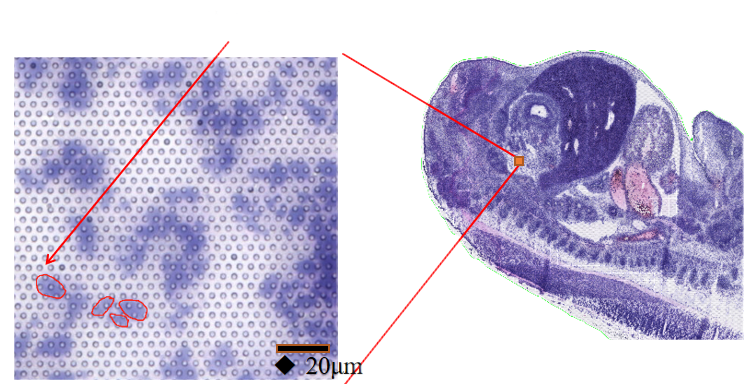
●Subcellulär upplösning: Varje infångningsområde innehöll >2 miljoner rumsliga streckkodade fläckar med en diameter på 2,5 µm och ett avstånd på 5 µm mellan fläckcentra, vilket möjliggör rumslig transkriptomanalys med subcellulär upplösning (5 µm).
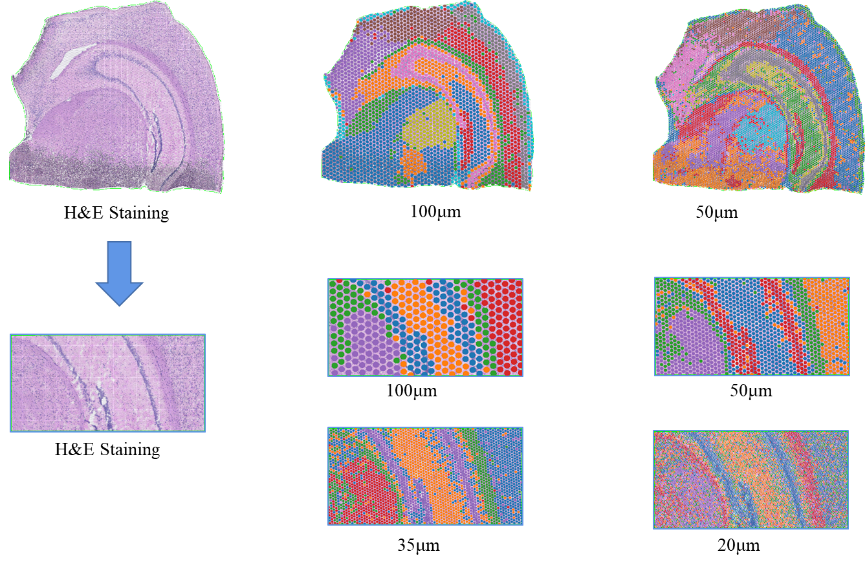
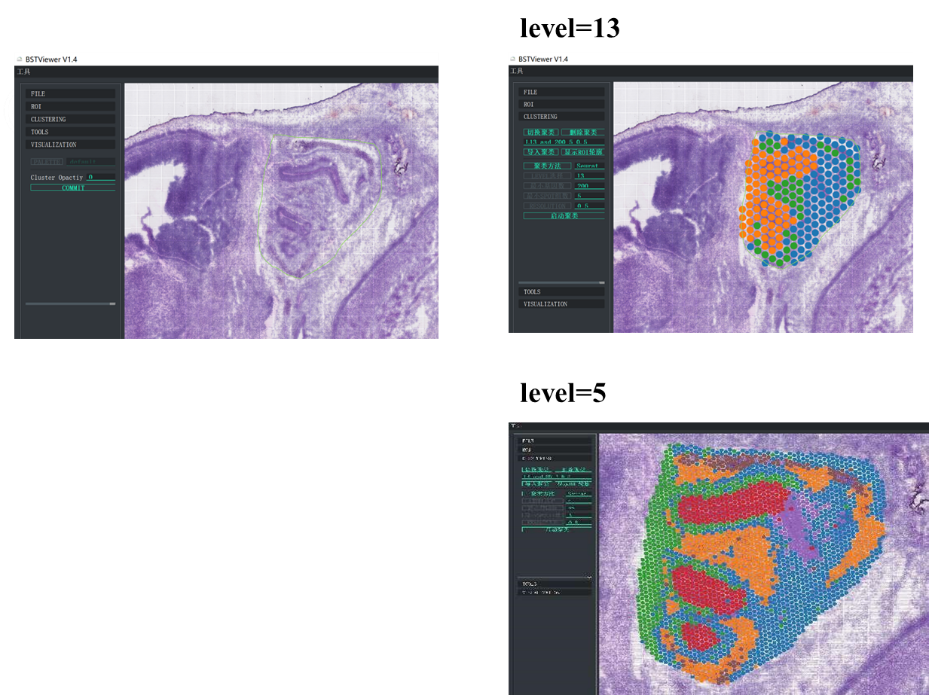
●Flernivåupplösningsanalys:Flexibel analys på flera nivåer från 100 μm till 5 μm för att lösa olika vävnadsfunktioner med optimal upplösning.
●Möjlighet att använda "Tre i en bild" cellsegmenteringsteknik:Genom att kombinera fluorescensfärgning, H&E-färgning och RNA-sekvensering på ett enda objektglas, möjliggör vår "tre-i-ett"-analysalgoritm identifieringen av cellgränser för efterföljande cellbaserad transkriptomik.
●Kompatibel med flera sekvenseringsplattformar: Både NGS och långläst sekvensering tillgängliga.
●Flexibel design av 1-8 Active Capture Area: Storleken på infångningsområdet är flexibel, det går att använda 3 format (6,8 mm * 6,8 mm., 11 mm * 11 mm och 15 mm * 20 mm)
●One-stop service: Den integrerar alla erfarenhets- och färdighetsbaserade steg, inklusive kryosektionering, färgning, vävnadsoptimering, rumslig streckkodning, biblioteksförberedelse, sekvensering och bioinformatik.
●Omfattande bioinformatik och användarvänlig visualisering av resultat:Paketet innehåller 29 analyser och 100+ högkvalitativa figurer, kombinerat med användning av egenutvecklad mjukvara för att visualisera och anpassa celldelning och punktklustring.
●Anpassad dataanalys och visualisering: tillgänglig för olika forskningsförfrågningar
●Mycket kompetent tekniskt team: med erfarenhet av över 250 vävnadstyper och 100+ arter inklusive människor, mus, däggdjur, fiskar och växter.
●Uppdateringar i realtid på hela projektet: med full kontroll över experimentella framsteg.
●Valfri gemensam analys med encellig mRNA-sekvensering
Servicespecifikationer
|
Prov Krav
| Bibliotek |
Sekvenseringsstrategi
| Data rekommenderas | Kvalitetskontroll |
| OCT-inbäddade kryoprover, 3 block per prov | S1000 cDNA-bibliotek | Illumina PE150 (andra plattformar tillgängliga) | 100K PE-avläsningar per 100 uM ( 60-150 Gb ) | RIN>7 |
För mer information om vägledning för provförberedelser och servicearbetsflöde är du välkommen att prata med aBMKGENE expert
Service arbetsflöde
I provberedningsfasen utförs ett första bulk-RNA-extraktionsförsök för att säkerställa att ett högkvalitativt RNA kan erhållas. I vävnadsoptimeringssteget färgas och visualiseras sektionerna och permeabiliseringsförhållandena för mRNA-frisättning från vävnad optimeras. Det optimerade protokollet tillämpas sedan under bibliotekskonstruktion, följt av sekvensering och dataanalys.
Det kompletta tjänstearbetsflödet innefattar uppdateringar i realtid och klientbekräftelser för att upprätthålla en responsiv återkopplingsslinga, vilket säkerställer smidigt projektgenomförande.
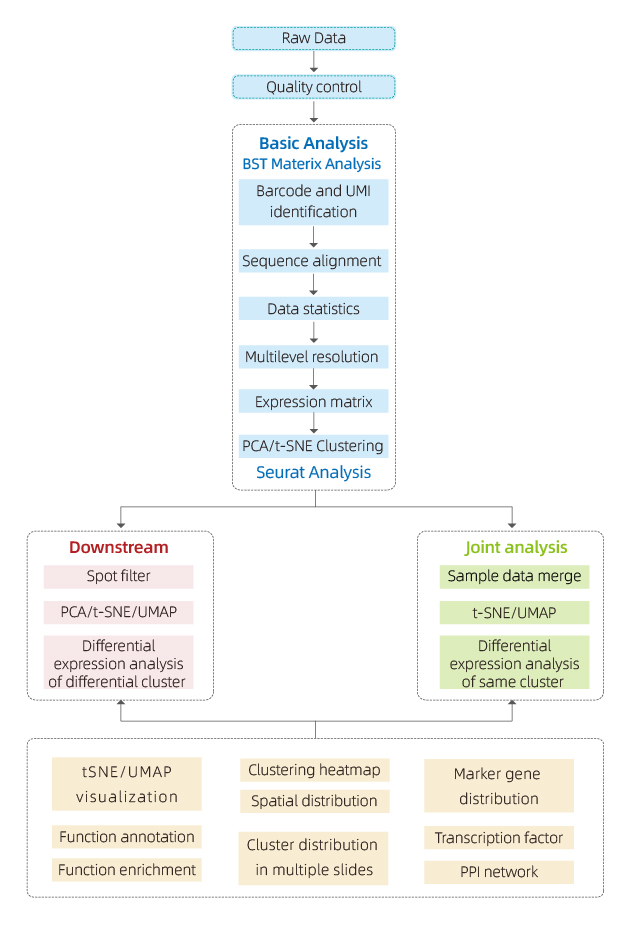
Data som genereras av BMKMANU S1000 analyseras med hjälp av programvaran "BSTMatrix", som är oberoende designad av BMKGENE och genererar en genuttrycksmatris. Därifrån genereras en standardrapport som inkluderar datakvalitetskontroll, internprovanalys och intergruppsanalys.
● Datakvalitetskontroll:
Datautgång och distribution av kvalitetspoäng
Gendetektion per plats
Vävnadstäckning
● Analys av inre prov:
Genrikedom
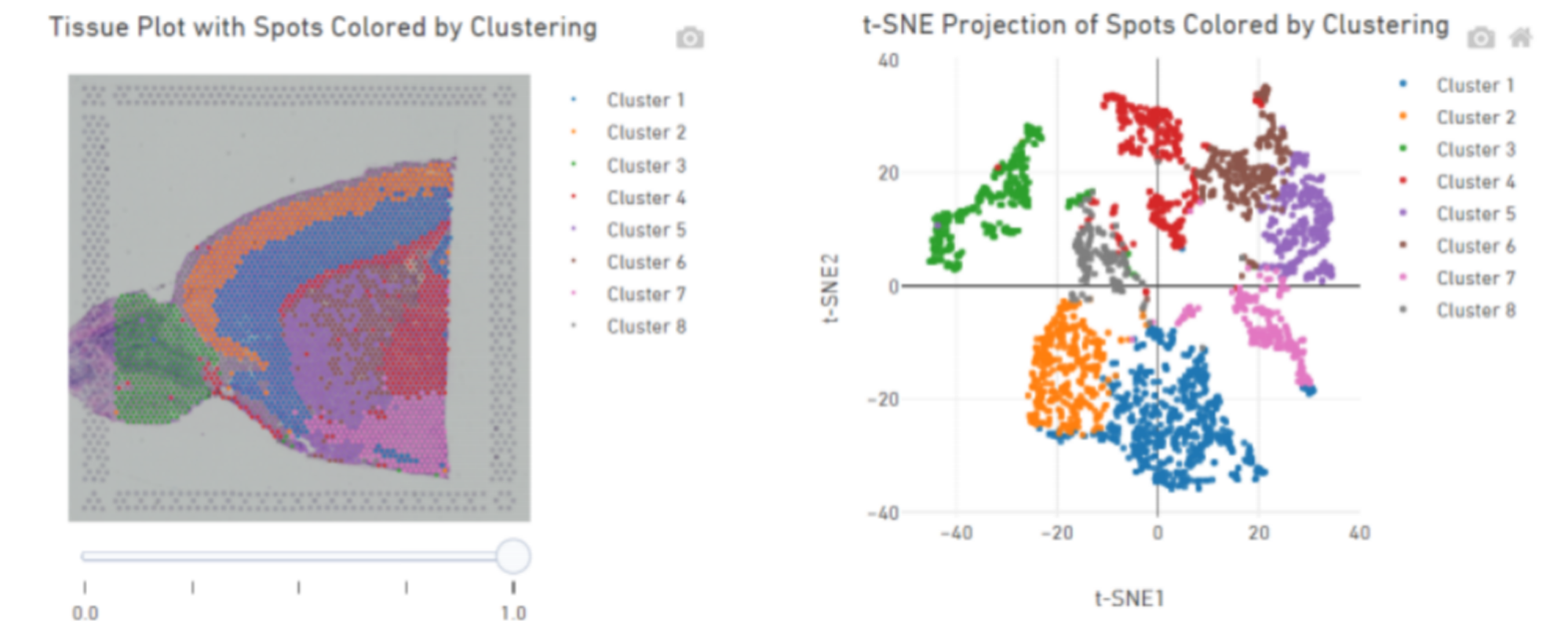
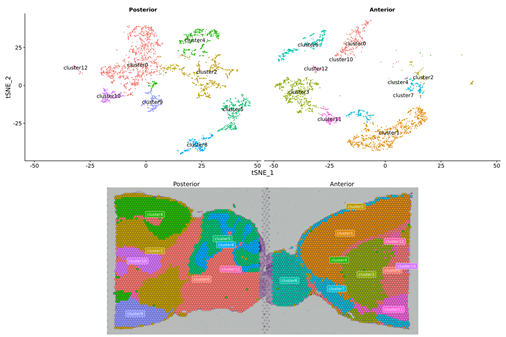
Punktklustring, inklusive reducerad dimensionsanalys
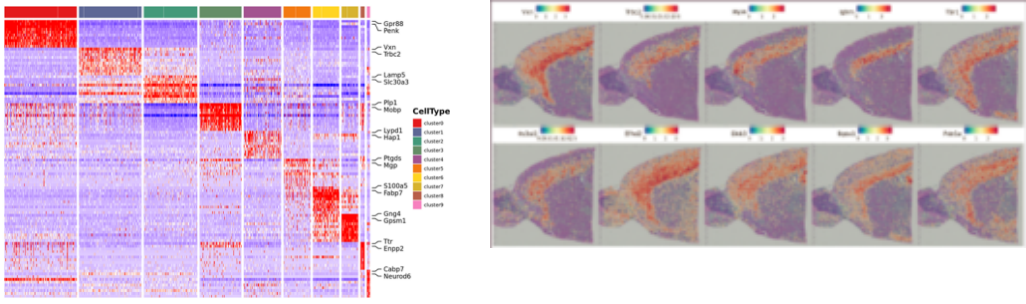
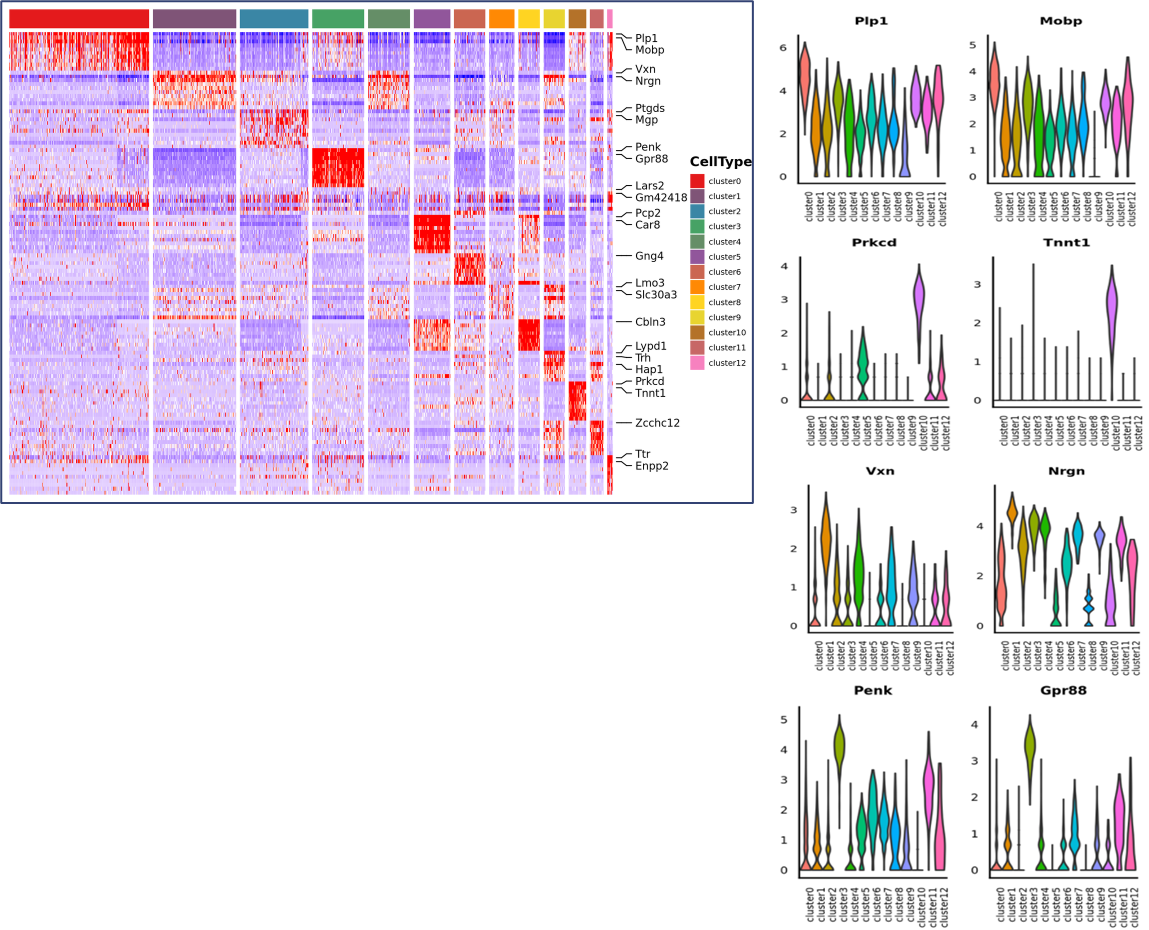
Differentiell expressionsanalys mellan kluster: identifiering av markörgener
Funktionell annotering och anrikning av markörgener
● Intergruppsanalys:
Återkombination av fläckar från både prover (t.ex. sjuka och kontroll) och re-kluster
Identifiering av markörgener för varje kluster
Funktionell annotering och anrikning av markörgener
Differentiellt uttryck för samma kluster mellan grupper
Dessutom utvecklade BMKGENE "BSTViewer" är ett användarvänligt verktyg som gör det möjligt för användaren att visualisera genuttrycket och fläckklustring i olika upplösningar.
BMKGene utvecklade mjukvara för användarvänlig visualisering
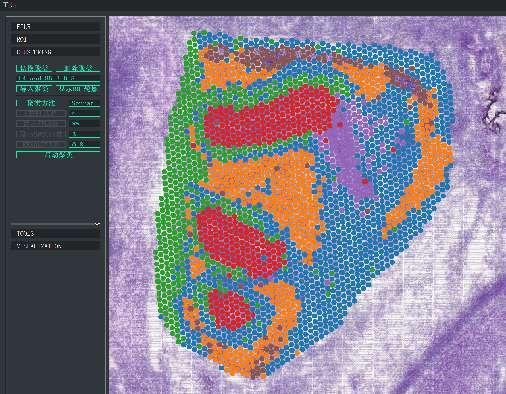
BSTViewer-punktklustring med flernivåupplösning
BSTcellViewer: automatisk och manuell celldelning
Analys av inre prov
Punktklustring:
Identifiering av markörgener och rumslig fördelning:
Intergruppsanalys
Datakombination från båda grupperna och re-kluster:
Markörgener för nya kluster:
Utforska framstegen som underlättas av BMKGenes rumsliga transkriptomiktjänster med BMKManu S1000-teknologin i denna utvalda publikation:
Song, X. et al. (2023) "Spatial transcriptomics avslöjar ljusinducerade klorenkymceller involverade i att främja skottregenerering i tomatkallus",Proceedings of the National Academy of Sciences of the United States of America120(38), sid. e2310163120. doi: 10.1073/pnas.2310163120
Du, Y. et al. (2023) "Systematisk jämförelse av sekvenseringsbaserade rumsliga transkriptomiska metoder",bioRxiv, sid. 2023.12.03.569744. doi: 10.1101/2023.12.03.569744.