Majelis Génom Dumasar Hi-C
Fitur Service
● Sequencing on Illumina NovaSeq kalawan PE150.
● Service merlukeun sampel jaringan, tinimbang asam nukléat sari, cross-link jeung formaldehida tur conserve interaksi DNA-protéin.
● Percobaan Hi-C ngalibatkeun pangwatesan jeung perbaikan tungtung tungtung caket jeung biotin, dituturkeun ku circularization tina tungtung Blunt hasilna bari preserving interaksi. DNA ieu lajeng ditarik ka handap kalawan manik streptavidin sarta dimurnikeun pikeun persiapan perpustakaan saterusna.
Kaunggulan Service

Tinjauan Hi-C
(Lieberman-Aiden E et al.,Élmu, 2009)
●Ngaleungitkeun Kabutuhan Data Populasi Genetik:Hi-C substitutes informasi penting diperlukeun pikeun contig anchoring.
●Kapadetan spidol luhur:ngarah kana rasio anchoring contig tinggi luhur 90%.
●Kaahlian éksténsif sareng catetan publikasi:BMKGene boga pangalaman vast kalawan leuwih ti 2000 kasus Hi-C Genome Majelis ti 1000 spésiés béda jeung sagala rupa patén-patén. Langkung ti 200 kasus anu diterbitkeun gaduh faktor dampak akumulatif langkung ti 2000.
●Tim bioinformatika anu terampil:kalayan patén-patén sareng hak cipta parangkat lunak pikeun percobaan Hi-C sareng analisa data, parangkat lunak data visualisasi anu dikembangkeun nyalira ngamungkinkeun blok manual mindahkeun, ngabalikeun, nyabut, sareng ngulang deui.
●Dukungan Pasca Penjualan:Komitmen urang ngalegaan saluareun parantosan proyék kalayan periode jasa saatos-jualan 3-bulan. Salila ieu, kami nawiskeun nurutan proyék, bantuan ngungkulan, sareng sesi Q&A pikeun ngajawab patarosan naon waé anu aya hubunganana sareng hasil.
●Anotasi Komprehensif: kami nganggo sababaraha basis data pikeun sacara fungsional ngémutan gen sareng variasi anu diidentifikasi sareng ngalaksanakeun analisa pengayaan anu saluyu, masihan wawasan ngeunaan sababaraha proyék panalungtikan.
Spésifikasi Service
| Persiapan perpustakaan | Stratégi sequencing | Disarankeun kaluaran data | Kontrol kualitas |
| Perpustakaan Hi-C | Illumina NovaSeq PE150 | 100x | Q30 ≥ 85% |
Sarat Sampel
| Tisu | Jumlah diperlukeun |
| Sato Viscera | ≥ 2 g |
| Otot sasatoan | |
| Getih Mamalia | ≥ 2 ml |
| Getih Unggas/Lauk | |
| Tutuwuhan- Daun seger | ≥ 3 g |
| Sél Kultur | ≥ 1x107 |
| Serangga | ≥ 2 g |
Aliran Gawé Palayanan

Desain ékspérimén

pangiriman sampel

Pangwangunan perpustakaan

Sequencing

Analisis data

jasa saatos-diobral
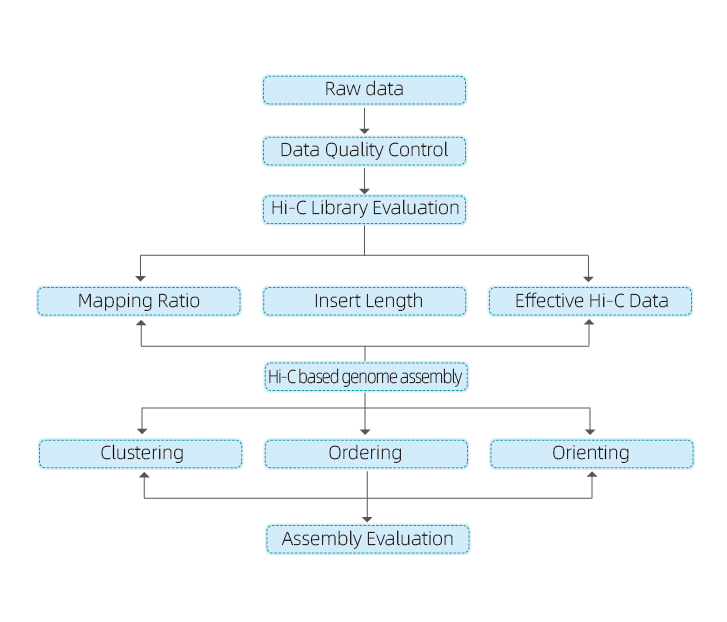
1) QC data atah
2) perpustakaan Hi-C QC: estimasi interaksi Hi-C valid
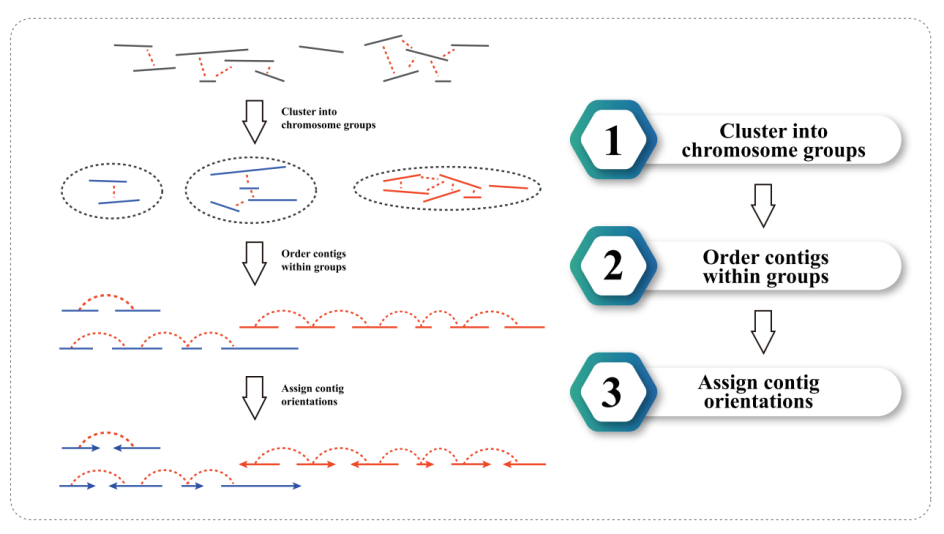
3) Hi-C assembly: clustering of contigs dina grup, dituturkeun ku contig ordering dina unggal grup jeung assigning contig orientasi
4) Evaluasi Hi-C
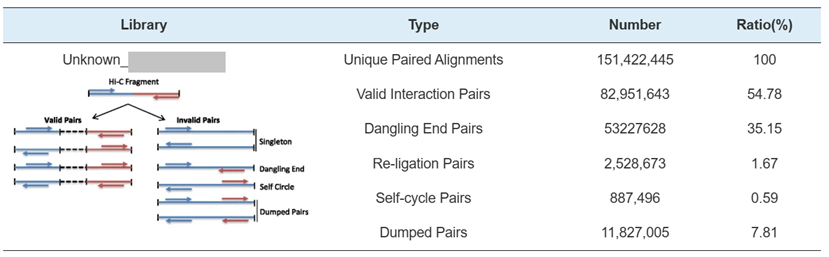
Hi-C Library QC - estimasi Hi-C pasangan interaksi valid
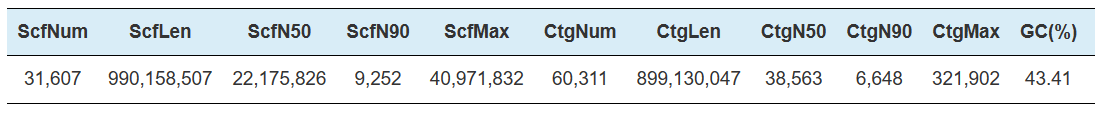
Hi-C Majelis - statistik
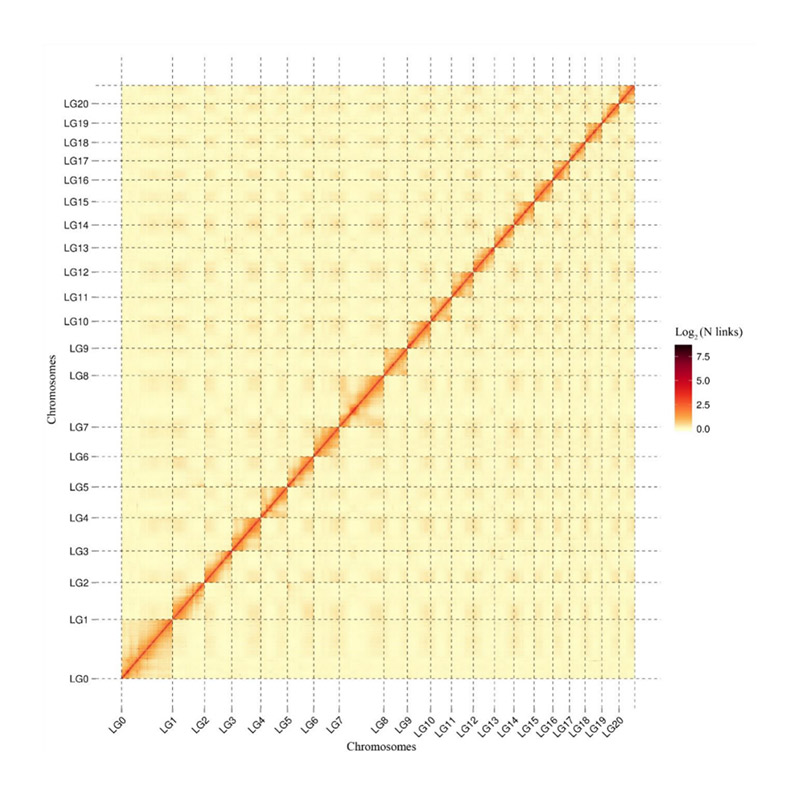
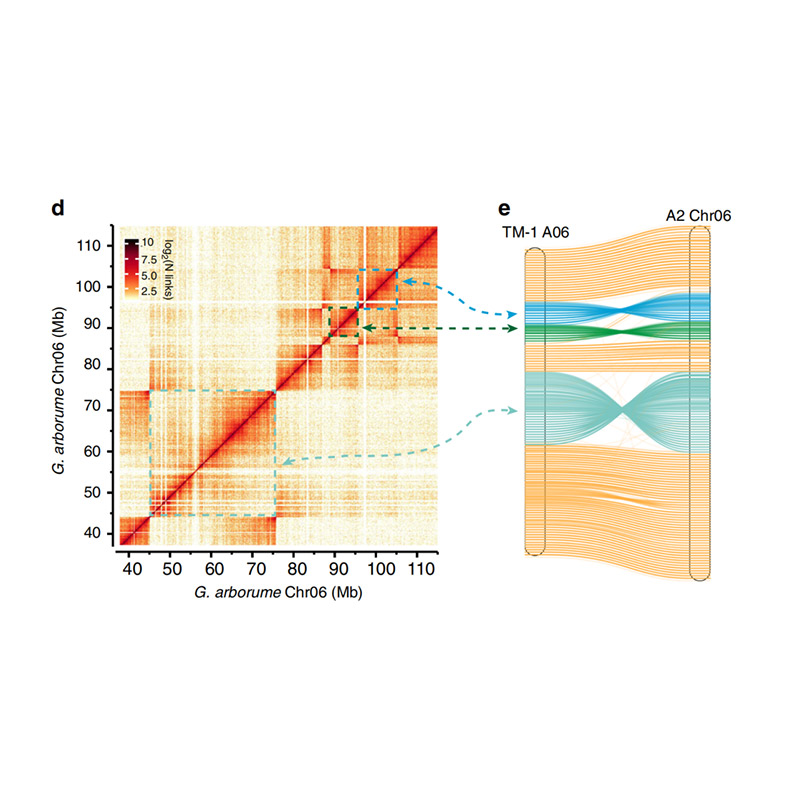
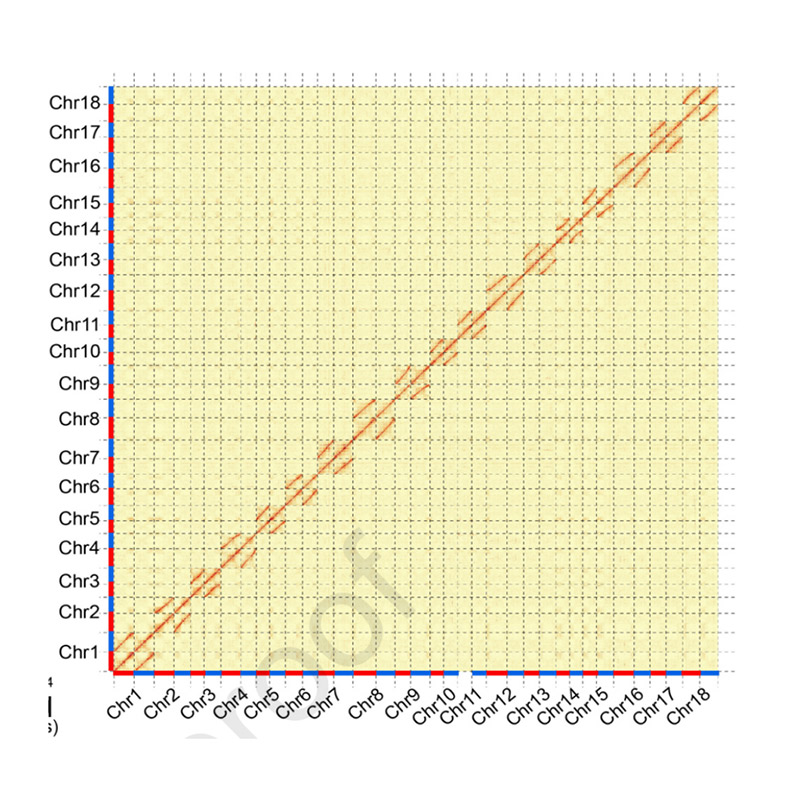
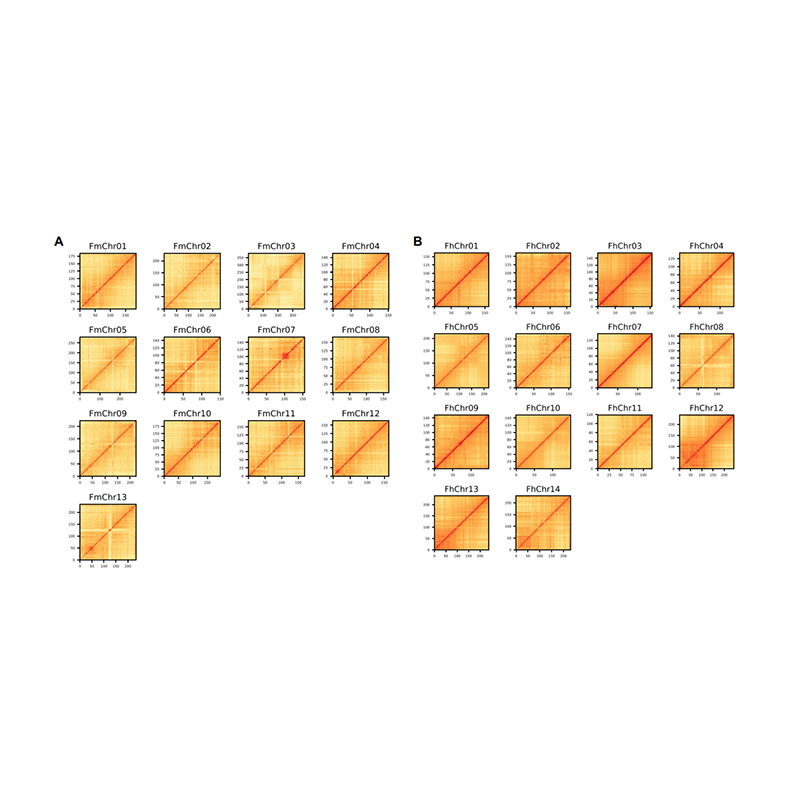
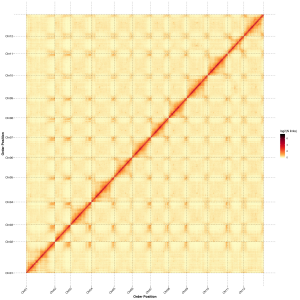
Evaluasi post-assembly - heatmap inténsitas sinyal antara bins
Jelajahi kamajuan anu difasilitasi ku jasa rakitan Hi-C BMKGene ngalangkungan koleksi publikasi anu disusun.
Tian, T. et al. (2023) 'Pangumpulan génom sareng dissection genetik tina germplasm jagung tahan halodo anu menonjol', Nature Genetics 2023 55: 3, 55 (3), pp. 496-506. Doi: 10.1038 / s41588-023-01297-y.
Wang, ZL et al. (2020) 'A Majelis Skala Kromosom tina Asian Honeybee Apis cerana Génom', Frontiers in Genetics, 11, p. 524140. doi: 10.3389 / FGENE.2020.00279 / BIBTEX.
Zhang, F. et al. (2023) 'Nungkabkeun évolusi biosintésis alkaloid tropana ku nganalisis dua génom dina kulawarga Solanaceae', Nature Communications 2023 14:1, 14(1), pp. 1–18. Doi: 10.1038 / s41467-023-37133-4.
Zhang, X. dkk. (2020) 'Genomes of the Banyan Tree and Pollinator Wasp Nyadiakeun Wawasan ngeunaan Fig-Wasp Coevolution', Cell, 183(4), pp. 875-889.e17. doi: 10.1016/J.CELL.2020.09.043