Bakterial dhe kërpudhat i tërë gjenomit të rregullimit
Avantazhet e shërbimit
>Analiza bioinformatike përfshin Thirrjen Variant:Sigurimi i pasqyrave funksionale për gjenomet e rivlerësuara.
● Ekspertizë e gjerë: Me mijëra projekte të rregullimit të mikrobeve të kryera çdo vit, ne sjellim një përvojë dekade, një ekip analize shumë të aftë, përmbajtje gjithëpërfshirëse dhe mbështetje të shkëlqyeshme pas shitjes.
>Mbështetje pas shitjes:Angazhimi ynë shtrihet përtej përfundimit të projektit me një periudhë shërbimi pas shitjes 3-mujore. Gjatë kësaj kohe, ne ofrojmë ndjekjen e projektit, ndihmën e zgjidhjes së problemeve dhe seancat e Q&A për të adresuar çdo pyetje që lidhen me rezultatet.
Specifikimet e shërbimit
| Platformë e sekuencave | Strategjia e sekuencave | Të dhënat e rekomanduara | Kontroll i cilësisë |
| Illumina Novaseq | PE150 | Thellësia prej 100x | Q30≥85% |
Kërkesat e mostrës
| Përqendrimi (ng/μl) | Shuma totale (NG) | Vëllimi (μl) |
| ≥1 | ≥60 | ≥20 |
Bakteret: ≥1x107 qelizë
Kërpudhat njëqelizore: ≥5x106-1x107 qelizë
Makro kërpudhat: ≥4g
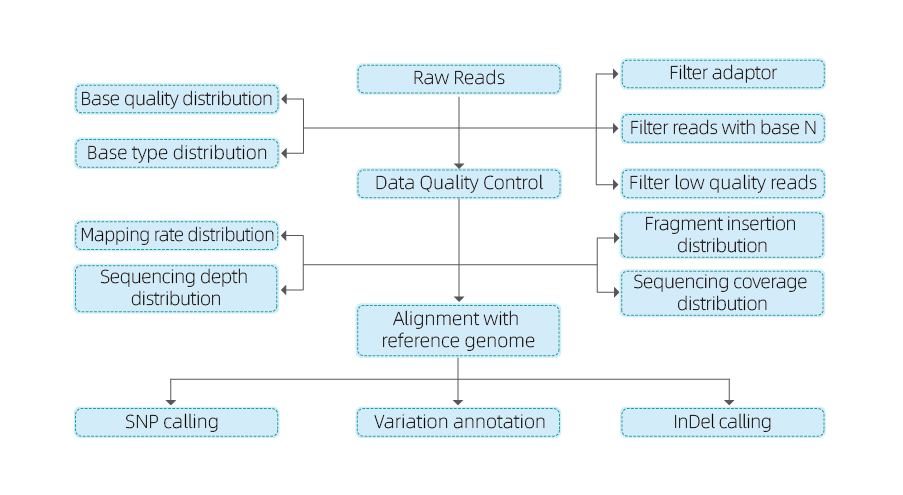
Rrjedha e punës së shërbimit

Dorëzimi i mostrës

Ndërtim bibliotekash

Sekuencim

Analiza e të dhënave

Shërbime pas shitjes
Përfshinë analizën e mëposhtme:
- Sekuencimi i kontrollit të cilësisë së të dhënave
- Përafrimi me gjenomin e referencës
- Thirrja Variant: SNP dhe Indel
- Shënim variant
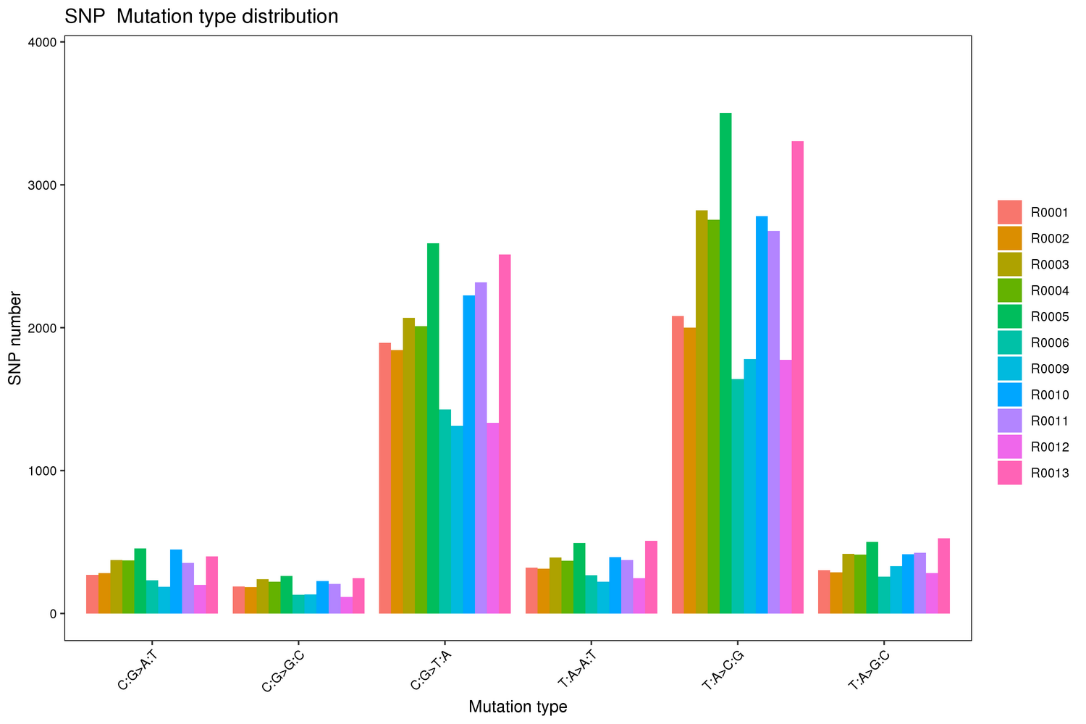
Thirrja Variant: Llojet e SNP
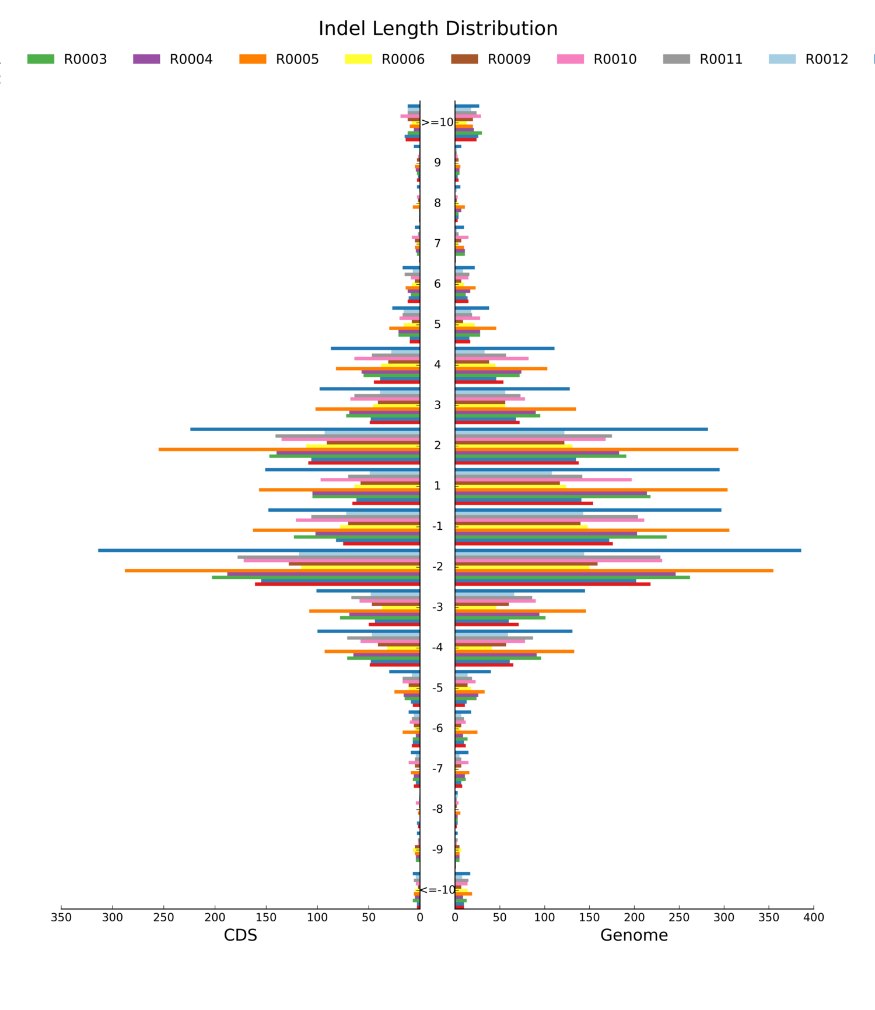
Thirrja Variant: Shpërndarja e gjatësisë së indel
Eksploroni përparimet e lehtësuara nga shërbimet e rregullimit të gjenomit mikrobik të BMKGene përmes një koleksioni të kuruar të botimeve.
Jia, Y. et al. (2023) 'Kombinimi i transkriptomit dhe i tërë i gjenomit të ri-rregullimit në gjenet e rezistencës ndaj sëmundjeve në ekran për bunt xhuxhin e grurit',,Revista Ndërkombëtare e Shkencave Molekulare, 24 (24). doi: 10.3390/ijms242417356.
Jiang, M. et al. (2023) 'Metabolizmi i glukozës i kontrolluar nga ampicilina manipulon kalimin nga toleranca në rezistencë në baktere',Përparimet e Shkencës, 9 (10). doi: 10.1126/sciadv.ade8582/furnizues_file/sciadv.ade8582_sm.pdf.
Yang, M. et al. (2022) 'Aliidiomarina Halalkaliphila sp. Nov., Një bakter haloalkalhilic i izoluar nga një liqen sode në rajonin e brendshëm autonome të Mongolisë, Kinë ', int. J. Syst. Evol.Mikrobiol, 72, f. 5263. Doi: 10.1099/ijsem.0.005263.
Zhu, Z., Wu, R. dhe Wang, G.-H. (2024) 'Sekuenca e gjenomit të Staphylococcus nepalensis ZZ-2023A, e izoluar nga Nasonia vitripennis',Njoftime për burimet e mikrobiologjisë. doi: 10.1128/MRA.00802-23.