Človeško celotno zaporedje eksome
Servisne funkcije
● Dve eksome plošči, ki sta na voljo na podlagi ciljne obogatitve s sondami: Seveda izberite Human All Exon V6 (Agilent) in XGen Exome Hibridizacijska plošča V2 (IDT).
● Zaporedje na Illumina Novaseq.
● Bioinformatski cevovod, usmerjen v analizo bolezni ali analizo tumorja.
Prednosti storitev
●Cilja območje kodiranja beljakovin: Z zajemanjem in zaporedjem regij kodiranja beljakovin se HWE uporablja za razkritje različic, povezanih s strukturo beljakovin.
●Stroškovno učinkovito:HWES prinaša približno 85% mutacij, povezanih s človeško boleznijo, iz 1% človeškega genoma.
●Visoka natančnost: Z visoko globino zaporedja HWES olajša odkrivanje skupnih različic in redkih različic s frekvencami, nižjimi od 1%.
●Strog nadzor kakovosti: V vseh fazah izvajamo pet jedrnih kontrolnih točk, od priprave vzorcev in knjižnic do zaporedja in bioinformatike. To natančno spremljanje zagotavlja dosledno kakovostne rezultate.
●Celovita analiza bioinformatike: Naš cevovod presega prepoznavanje sprememb referenčnega genoma, saj vključuje napredno analizo, namenjeno posebej obravnavanju raziskovalnih vprašanj, povezanih z genetskimi vidiki bolezni ali analizo tumorja.
●Podpora po prodaji:Naša zaveza se presega zaključek projekta s trimesečnim poprodajnim obdobjem. V tem času ponujamo nadaljnje spremljanje projektov, odpravljanje pomoči in vprašanj in vprašanj za reševanje vseh poizvedb, povezanih z rezultati.
Vzorčne specifikacije
| Strategija zajemanja eksona | Strategija zaporedja | Priporočeni izhod podatkov |
| Seveda izberite Human All Exon V6 (agilent) ali hibridizacijska plošča XGen Exome V2 (IDT)
| Illumina Novaseq PE150 | 5 -10 GB Za Mendelove motnje/redke bolezni:> 50x Za vzorce tumorja: ≥ 100x |
Vzorčne zahteve
| Vrsta vzorca
| Znesek(Qubit®)
| Koncentracija | Glasnost
| Čistost (Nanodrop ™) |
|
Genomska DNK
| ≥ 50 ng | ≥ 6 ng/μl | ≥ 15 μl | OD260/280 = 1,8-2,0 Brez degradacije, brez kontaminacije
|
Bioinformatika

Bioinformatična analiza vzorcev HWES-diaze vključuje:
● Podatki o zaporedju QC
● Referenčna poravnava genoma
● Identifikacija SNP -jev in indelov
● Funkcionalno pripisovanje SNP -jev in Indelov

Bioinformatična analiza vzorcev tumorjev vključuje:
● Podatki o zaporedju QC
● Referenčna poravnava genoma
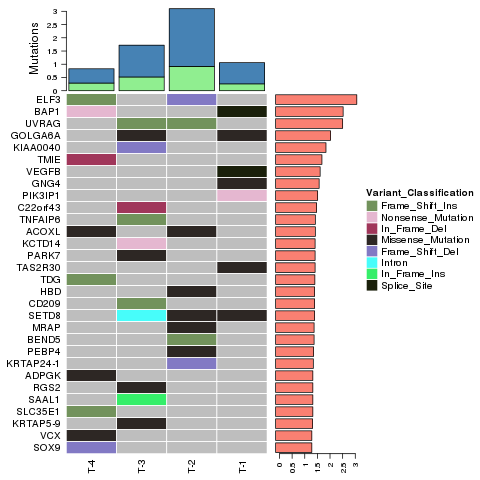
● Identifikacija SNP -jev, indelov in somatskih različic
● Identifikacija variant zarodne linije
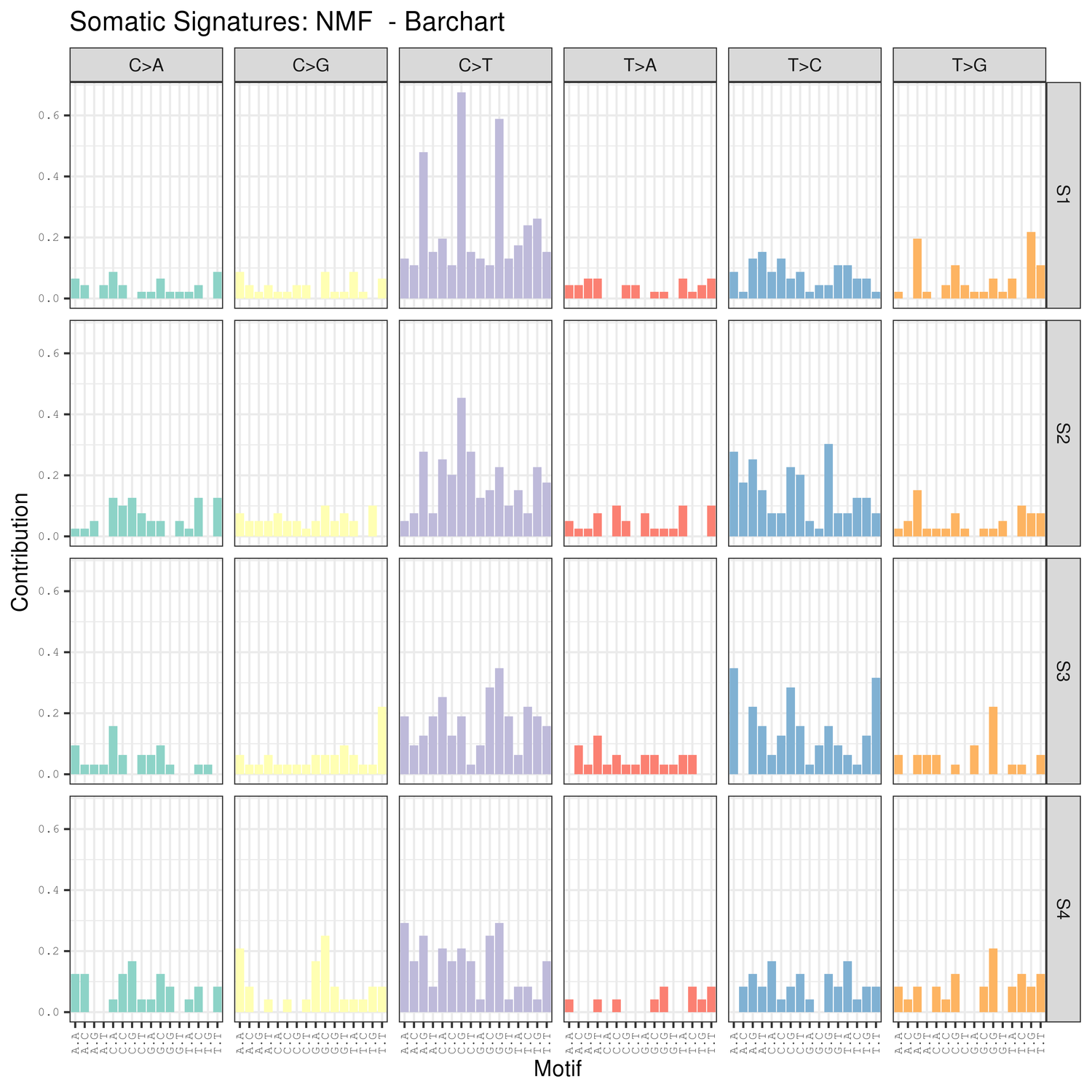
● Analiza podpisov mutacij
● Identifikacija pogonskih genov na podlagi mutacij pridobivanja funkcije
● Pripomba mutacije na ravni dovzetnosti za zdravila
● Analiza heterogenosti - izračun čistosti in ploodije
Servisni delovni tok

Dostava vzorca

Ekstrakcija DNK

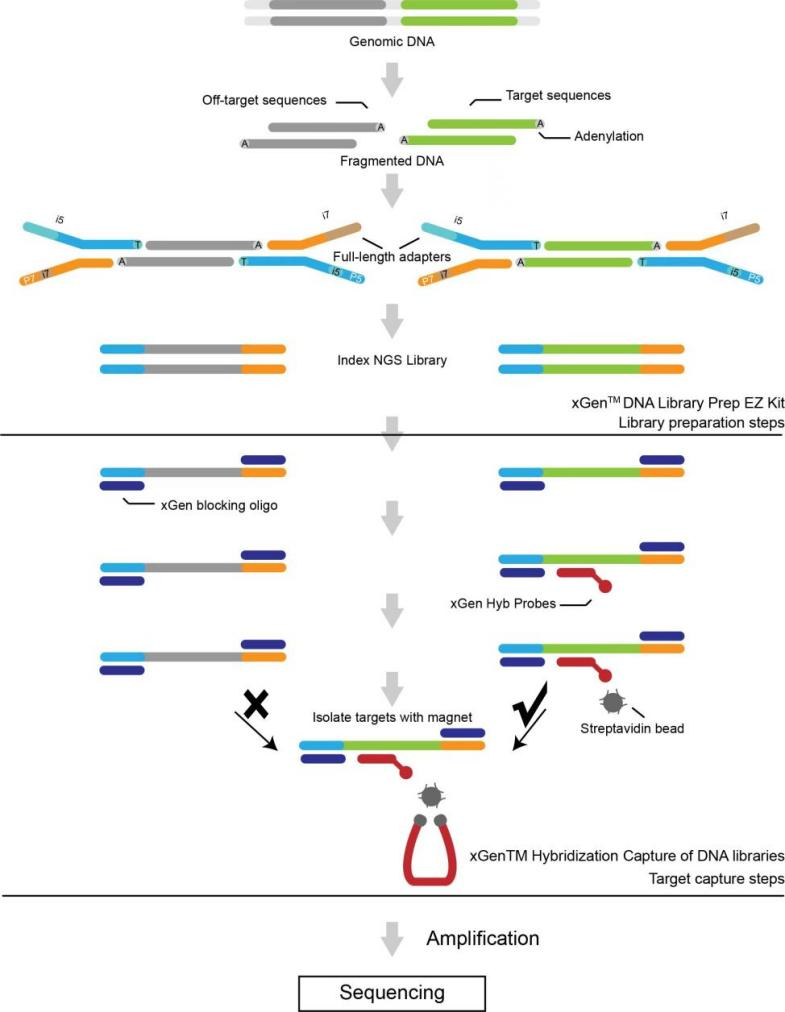
Konstrukcija knjižnice

Zaporedje

Analiza podatkov

Dostava podatkov

Po prodajnih storitvah
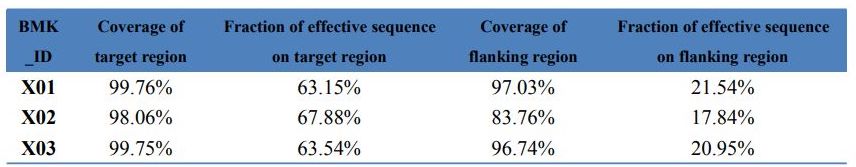
Podatki QC - Statistika zajemanja eksome
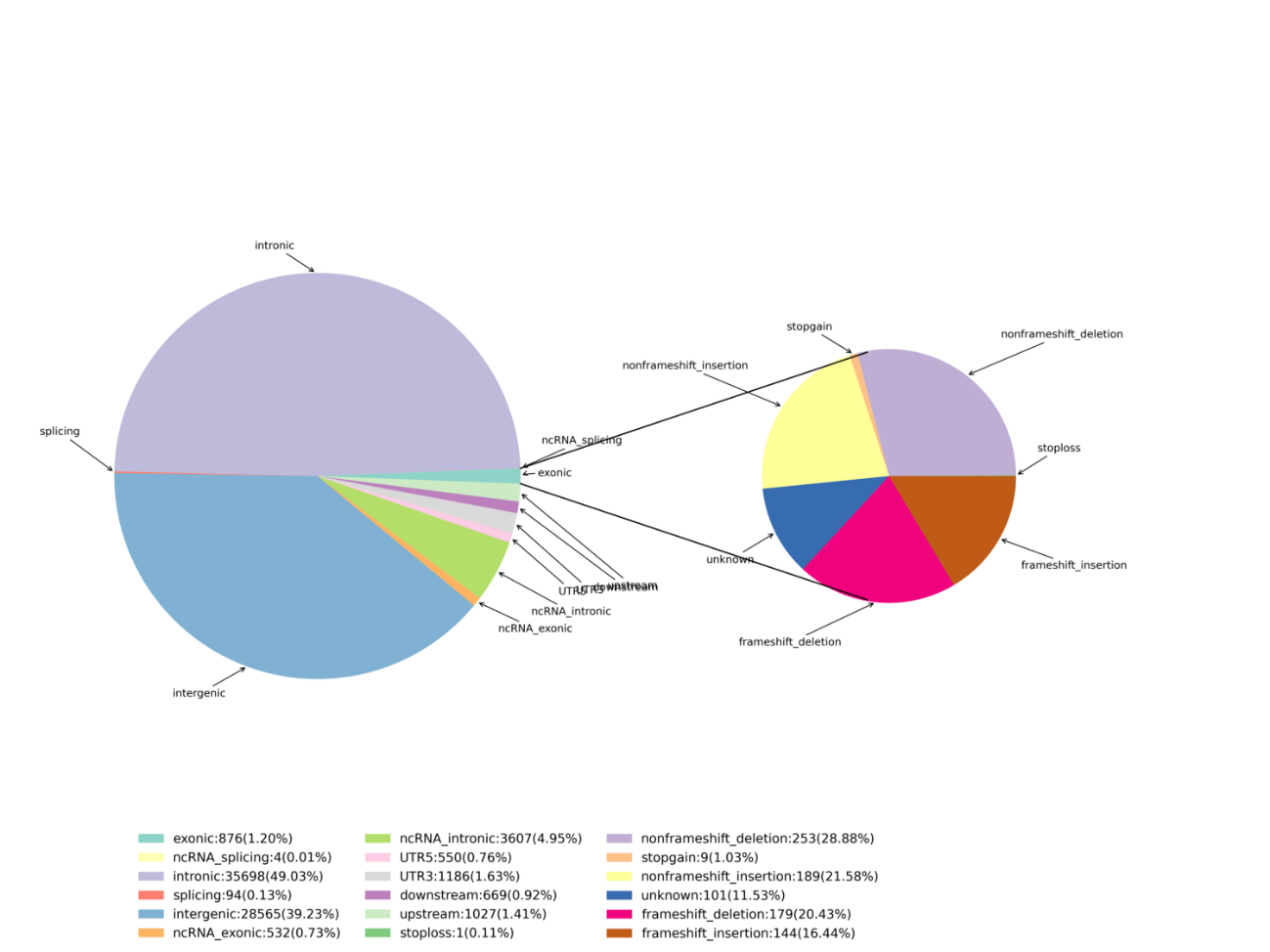
Identifikacija variante - Indeli
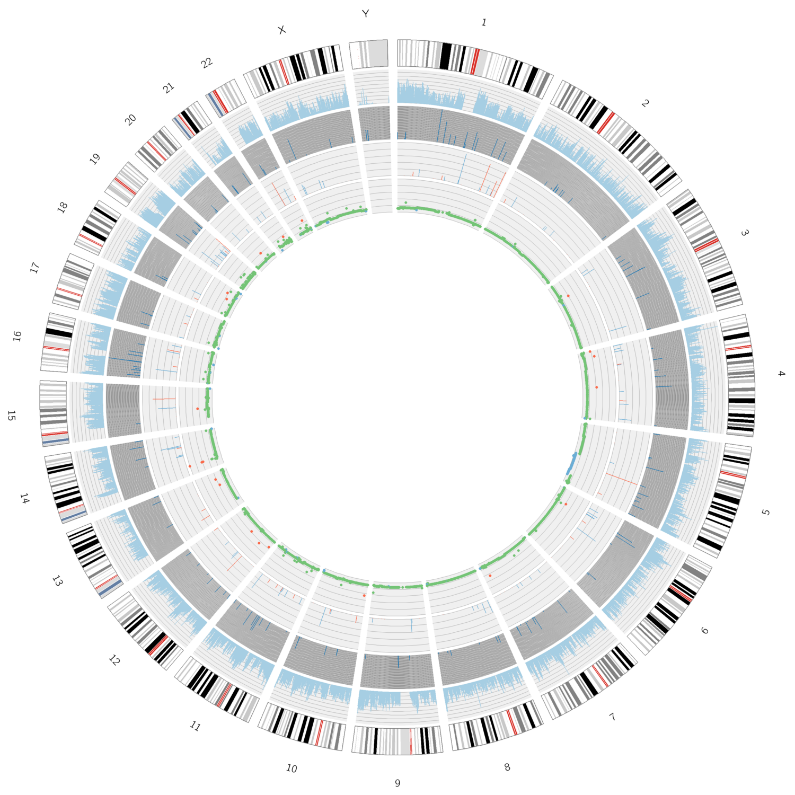
Napredna analiza: Identifikacija in porazdelitev škodljivih SNPS/INDELS - Cirsos Plot
Analiza tumorja: identifikacija in porazdelitev somatskih mutacij - cirsos ploskev
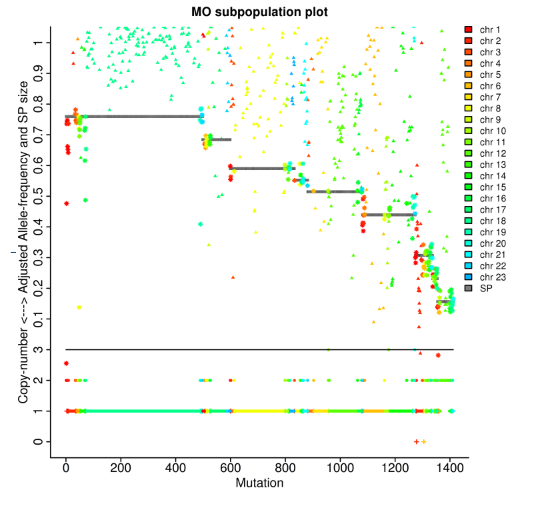
Analiza tumorja: klonske rodove