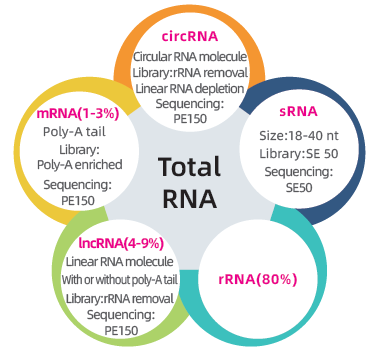
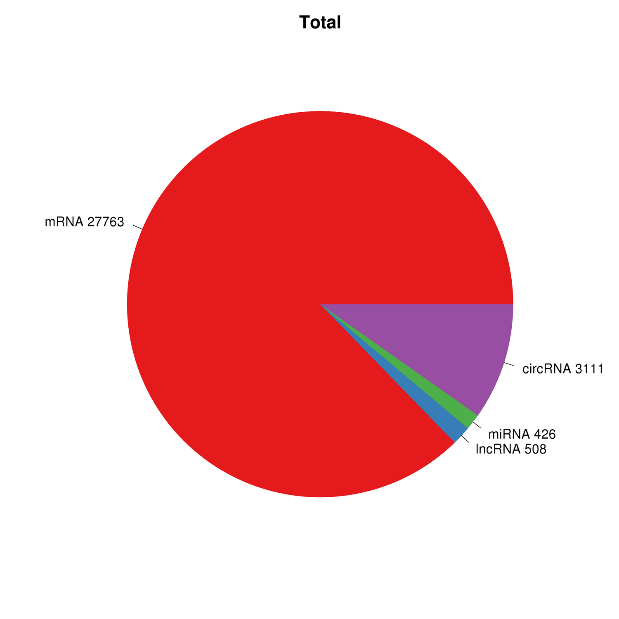
Секвенирование всего транскриптома - Illumina
Функции
● Двойная библиотека для последовательности Полного транскриптома: истощение рРНК с последующим подготовкой библиотеки PE150 и выбором размера с последующей подготовкой библиотеки SE50
● Полный анализ биоинформатики мРНК, LNCRNA, CIRCRNA и miRNA в отдельных отчетах о биоинформатике
● Совместный анализ всей экспрессии РНК в комбинированном отчете, включая анализ сети CERNA.
Сервисные преимущества
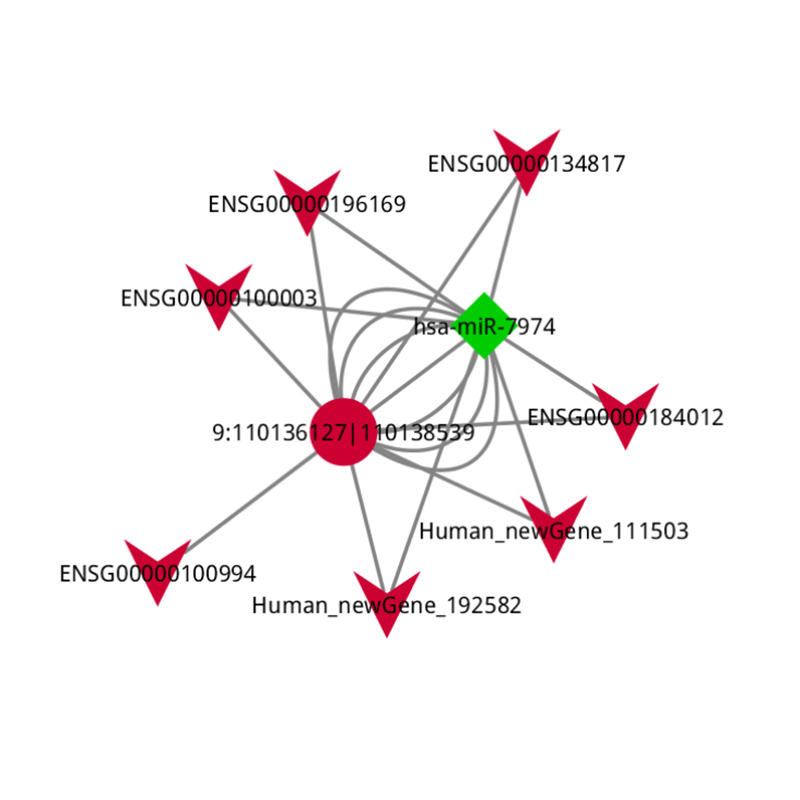
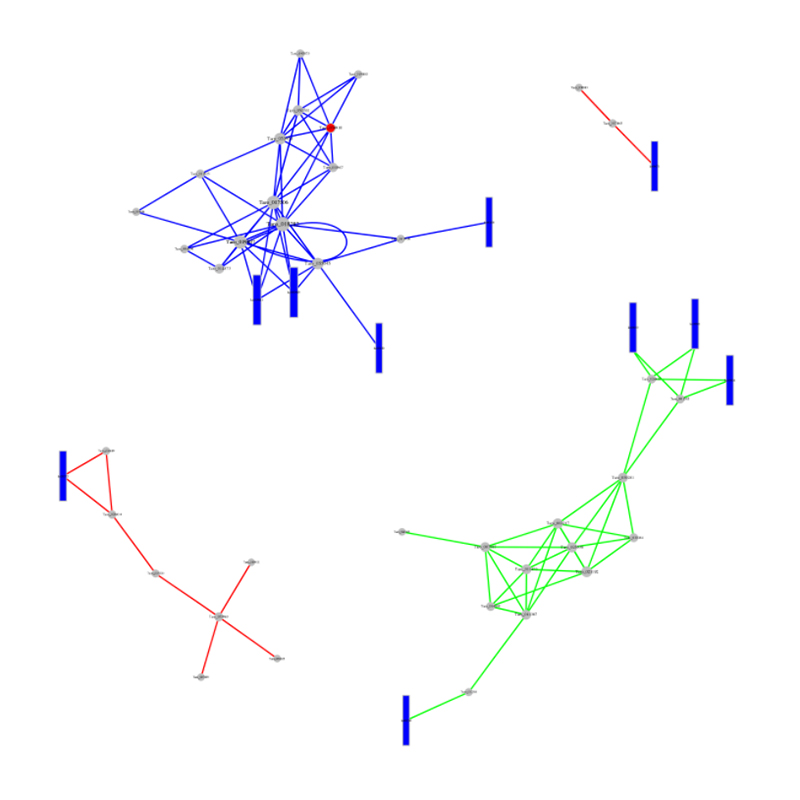
●Углубленный анализ нормативных сетей: Анализ сети CERNA включен путем совместного секвенирования мРНК, LNCRNA, CIRCRNA и miRNA и исчерпывающим биоинформатическим рабочим процессом.
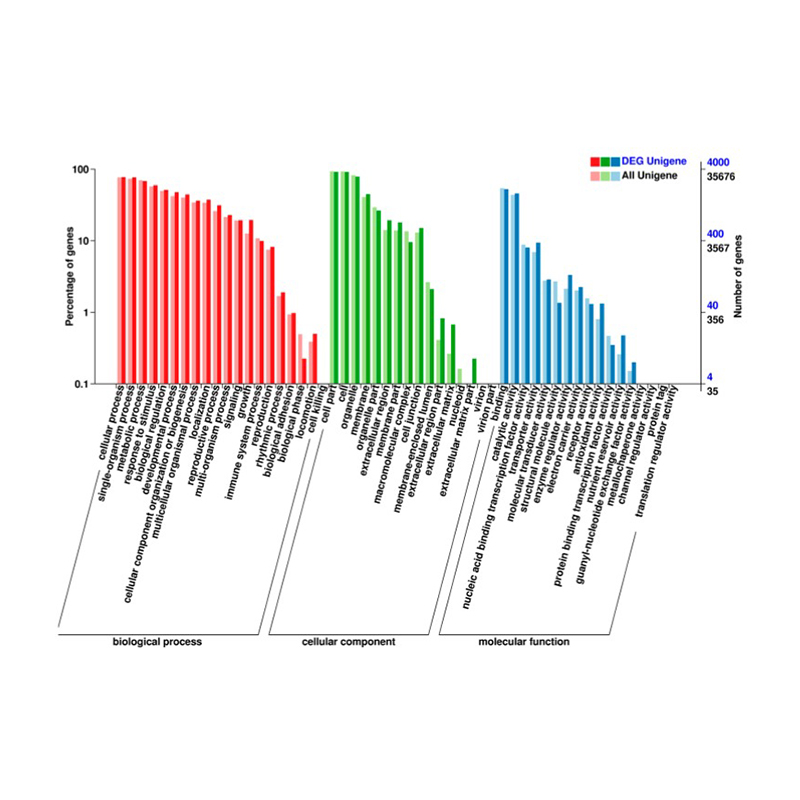
●Комплексная аннотация: Мы используем несколько баз данных для функциональной аннотации дифференциально экспрессируемых генов (DEGS) и выполняем соответствующий анализ обогащения, предоставляя понимание клеточных и молекулярных процессов, лежащих в основе реакции транскриптома.
●Обширный опыт: С учетом успешного закрытия более 2100 целых транскриптомных проектов в различных исследовательских областях наша команда приносит богатый опыт в каждый проект.
●Строгий контроль качества: Мы реализуем контрольные точки основного контроля на всех этапах, от подготовки образца и библиотеки до секвенирования и биоинформатики. Этот тщательный мониторинг обеспечивает доставку неизменно качественных результатов.
●Пост-продажа поддержка: Наше обязательство выходит за рамки завершения проекта с 3-месячным периодом послепродажного обслуживания. В течение этого времени мы предлагаем последующее наблюдение за проектом, помощь в устранении неполадок и сеансы вопросов и ответов для решения любых запросов, связанных с результатами
Требования к выборке и доставка
| Библиотека | Стратегия секвенирования | Данные рекомендуются | Контроль качества |
| РРНК истощается | Illumina PE150 | 16 Гб | Q30≥85% |
| Выбранный размер | Illumina SE50 | 10-20M читает |
Требования к образцу:
Нуклеотиды:
| Конц. (Нг/мкл) | Количество (мкг) | Чистота | Честность |
| ≥ 80 | ≥ 1,6 | OD260/280 = 1,7-2,5 OD260/230 = 0,5-2,5 Ограниченное или нет белка или загрязнения ДНК, показанного на геле. | Rin≥6.0 5,0≥28S/18Sц 1,0; ограниченная или нет базовой высоты |
Рекомендуемая доставка образца
Контейнер: 2 мл центрифужная трубка (оловянная фольга не рекомендуется)
Образец маркировки: группа+репликация, например, A1, A2, A3; B1, B2, B3.
Отгрузка:
1. Сухой лен: образцы должны быть упакованы в мешки и похоронены в сухих льда.
2. Rnastable Tribes: Образцы РНК можно сушить в трубе стабилизации РНК (например, Rnastable®) и отправлены при комнатной температуре.
Обслуживание рабочего перехода

Дизайн эксперимента

Образец доставки

РНК -экстракция

Библиотека строительство

Секвенирование

Анализ данных

Послепродажные услуги
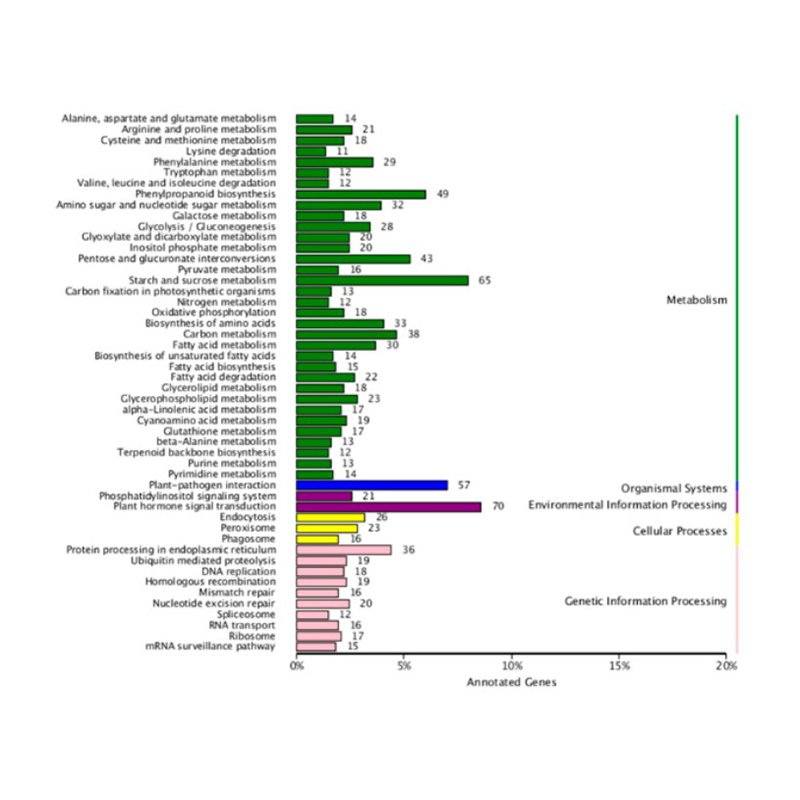
Биоинформатика
Обзор экспрессии РНК
Дифференциально экспрессируемые гены
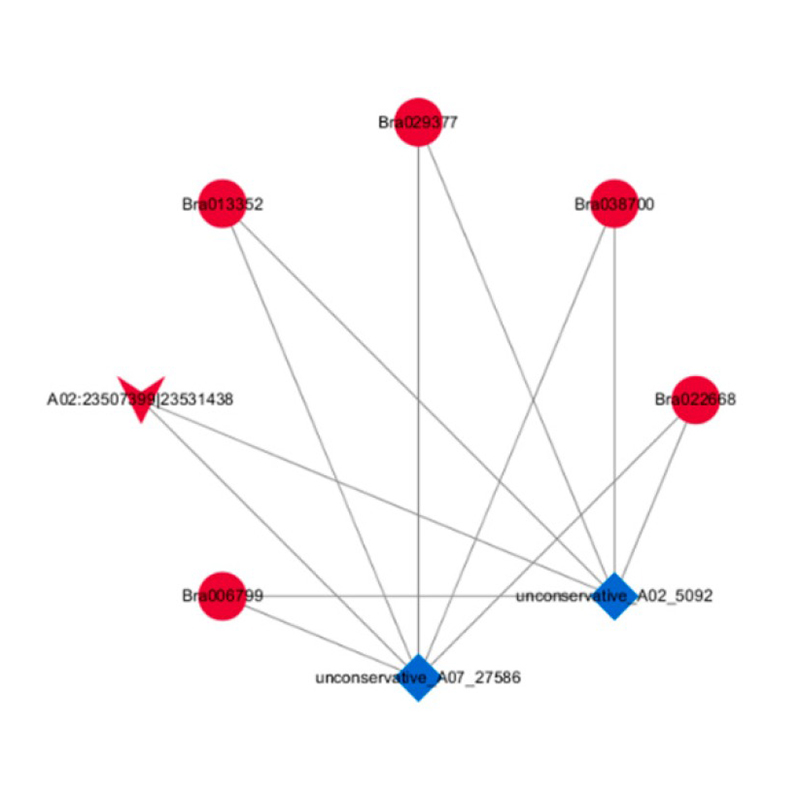
Анализ CERNA
Исследуйте успехи в исследованиях, которые облегчают услуги секвенирования BMKGene, посредством кураторской коллекции публикаций.
Dai, Y. et al. (2022) «Комплексные профили экспрессии мРНК, LNCRNAS и miRNAs при заболевании кашин-газа, идентифицированные с помощью РНК-секвенирования», Molecular Omics, 18 (2), стр. 154–166. doi: 10.1039/d1mo00370d.
Лю, Н. Нан и соавт. (2022) «Полная длина транскриптомов анализа холодной устойчивости API Cerana в горе Чанбай в течение записанного периода», Gene, 830, pp. 146503–146503. doi: 10.1016/j.gene.2022.146503.
Wang, XJ et al. (2022) «Приоритизация конкурирующих эндогенных РНК-регуляционных сетей на основе мульти-амической интеграции в малых клеточных раке легких: молекулярные характеристики и кандидаты на лекарства», «Границы в онкологии», 12, p. 904865. DOI: 10.3389/fonc.2022.904865/bibtex.
Сюй, П. и соавт. (2022) «Интегрированный анализ профилей экспрессии lncrna/circrna-mirna-mRNA выявляет новое понимание потенциальных механизмов в ответ на нематоды корневого узла в арахисе», BMC Genomics, 23 (1), стр. 1–12. doi: 10.1186/S12864-022-08470-3/Рисунки/7.
Ян, З. и соавт. (2022) «Секвенирование РНК всего транскриптома подчеркивает молекулярные механизмы, связанные с поддержанием качества полосатоговеса в брокколи путем облучения красного светодиода», Постаночная биология и технология, 188, с. 111878. DOI: 10.1016/j.posttharvbio.2022.111878.