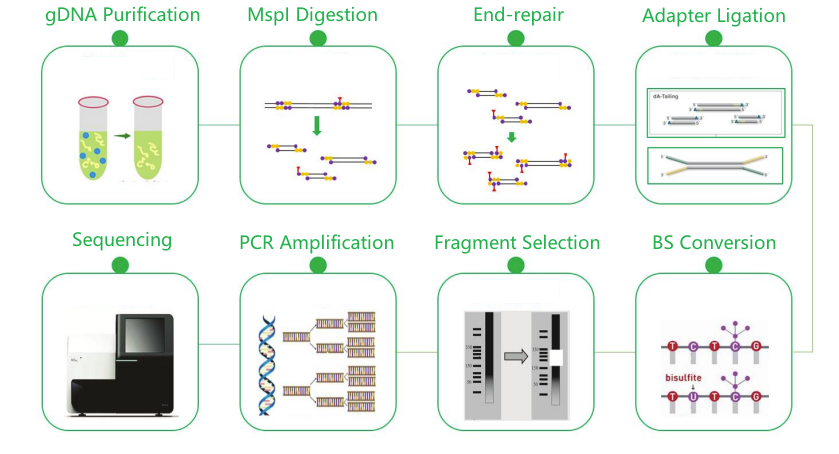
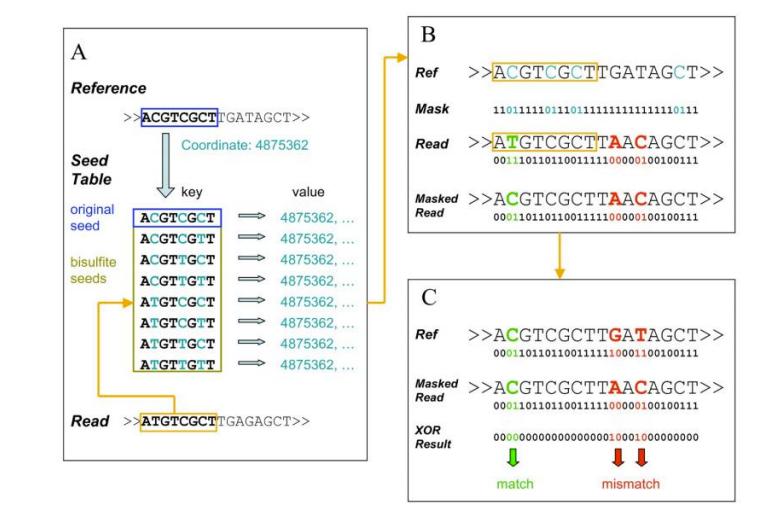
Секвенирование бисульфита с уменьшенным представлением (RRB)
Сервисные функции
● Требуется эталонный геном.
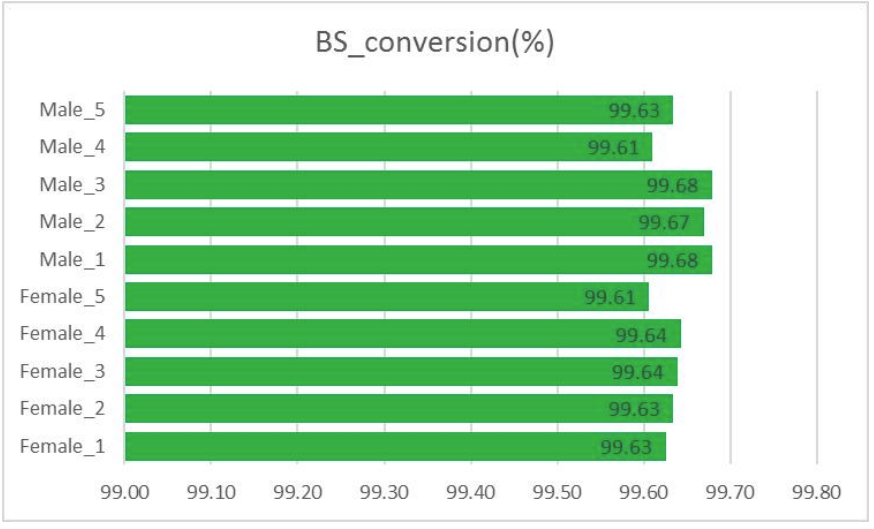
● ДНК Lambda используется для мониторинга эффективности преобразования бисульфита.
● Также контролируется эффективность пищеварения MSPI.
● Двойное пищеварение ферментов для образцов растений.
● Секвенирование на Illumina novaseq.
Сервисные преимущества
●Эффективная и эффективная альтернатива WGBS: Включение анализа быть выполнено при более низких затратах и с более низкими требованиями выборки.
●Полная платформа:Предоставьте отличный сервис от обработки образцов, конструкции библиотеки и секвенирования до анализа биоинформатики.
●Обширный опыт: Благодаря успешному завершению проектов секвенирования RRBS в разнообразных видах, BMKGene приносит более десятилетия опыта, высококвалифицированную группу по анализу, всеобъемлющий контент и отличную поддержку после продажи.
Спецификации обслуживания
| Библиотека | Стратегия секвенирования | Рекомендуемый выход данных | Контроль качества |
| Расщепленная MSPI и бисульфита, обработанная библиотекой | Illumina PE150 | 8 ГБ | Q30 ≥ 85% Конверсия бисульфита> 99% Эффективность резки MSPI> 95% |
Требования к выборке
| Концентрация (нг/мкл) | Общее количество (мкг) |
| |
| Геномная ДНК | ≥ 30 | ≥ 1 | Ограниченная деградация или загрязнение |
Обслуживание рабочего перехода

Образец доставки

Библиотека строительство

Секвенирование

Анализ данных
Послепродажные услуги
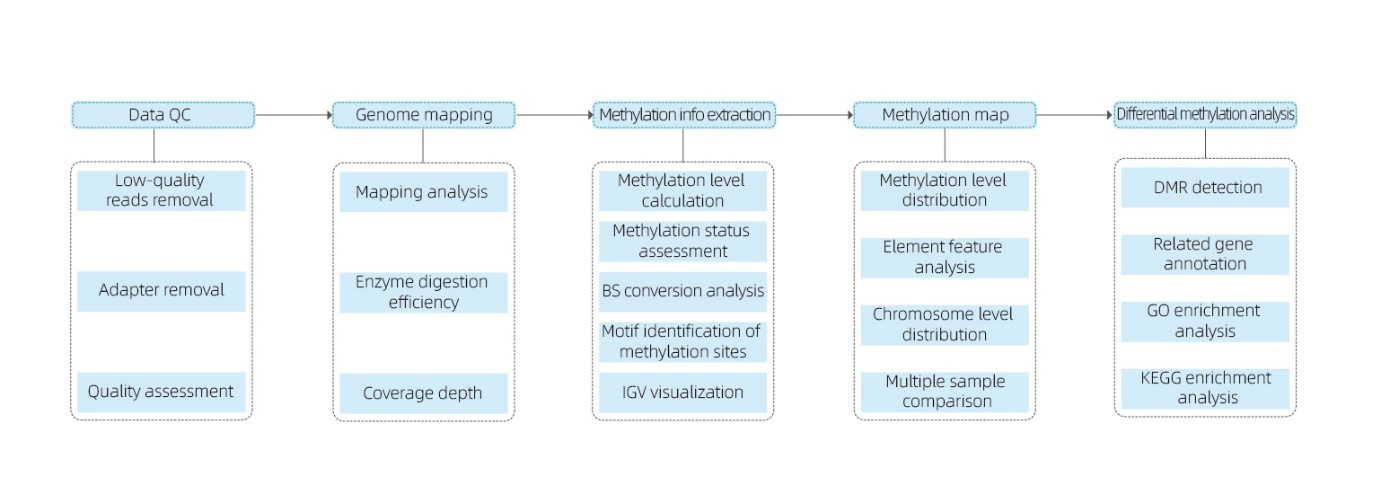
Включает следующий анализ:
● Сырой контроль качества секвенирования;
● Сопоставление на ссылку генома;
● Обнаружение 5 МС метилированных оснований и идентификации мотивов;
● Анализ распределения метилирования и сравнения образцов;
● Анализ дифференциально метилированных областей (DMR);
● Функциональная аннотация генов, связанных с DMRS.
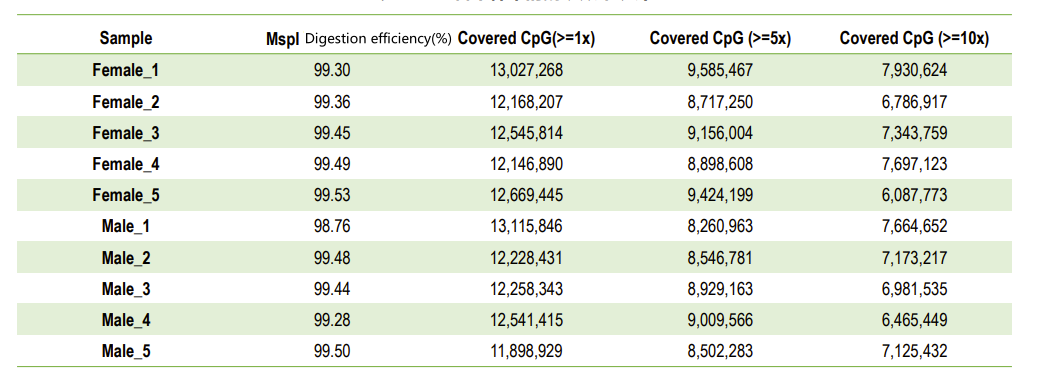
Контроль качества: эффективность пищеварения (в картировании генома)
Контроль качества: преобразование бисульфита (при извлечении метилирования)
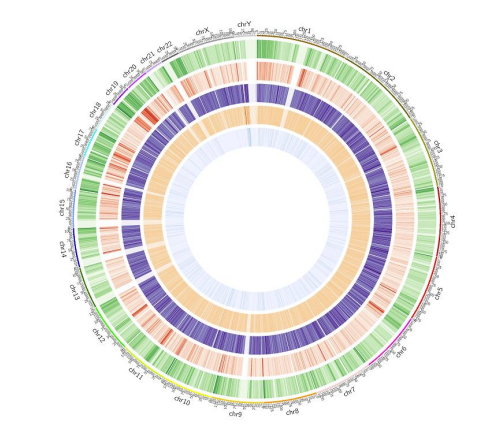
Карта метилирования: 5 МС метилирование по всему геному.
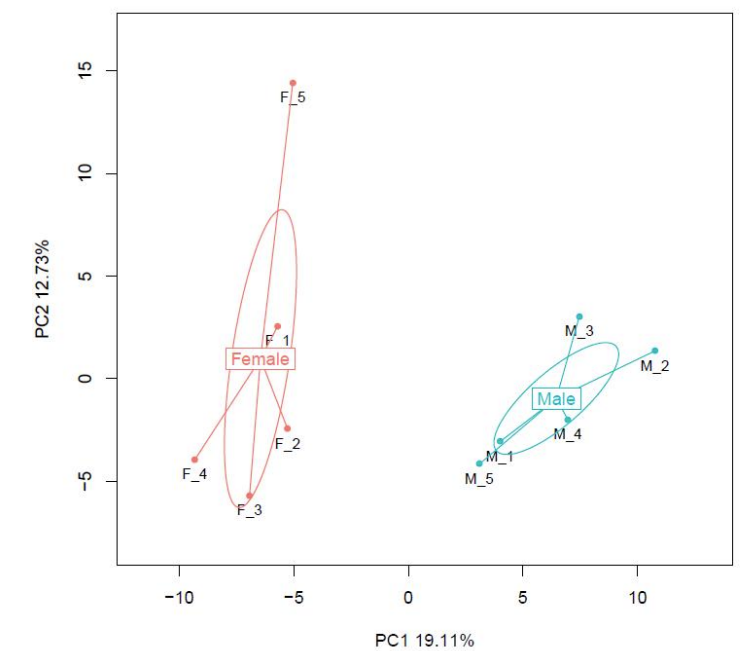
Сравнение образцов: анализ основных компонентов
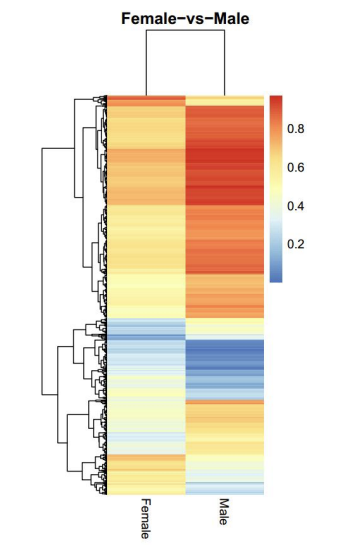
Дифференциально метилированные области (DMRS) Анализ: тепловая карта
Исследуйте достижения в области исследований, облегченные услугами секвенирования бисульфита BMKGene, посредством кураторской коллекции публикаций.
Li, Z. et al. (2022) «Перепрограммирование высокой точки зрения в Leydig-подобные клетки с помощью активации CRISPR и факторов паракрины»,PNAS Nexus, 1 (4). doi: 10.1093/pnasnexus/pgac179.
Tian, H. et al. (2023) «Анализ метилирования ДНК в масштабах генома на составе тела у китайских монозиготных близнецов»,Европейский журнал клинических исследований, 53 (11), с. E14055. doi: 10.1111/eci.14055.
Wu, Y. et al. (2022) «Метилирование ДНК и соотношение талии к хипа: исследование ассоциации в широком эпигеноме на китайских монозиготических близнецах»,Журнал эндокринологического исследования, 45 (12), с. 2365–2376. doi: 10.1007/s40618-022-01878-4.