Секвенирование MetataTrancprickome
Сервисные функции
● Истощение рРНК с последующим подготовкой библиотеки мРНК направленного мРНК.
● Секвенирование на Illumina novaseq.
Сервисные преимущества
●Изучите изменения сложных микробных сообществ:Это происходит на уровне транскрипции и исследует потенциальные новые гены.
●Объяснение взаимодействия микробного сообщества с хозяином или окружающей средой.
●Комплексный биоинформационный анализ: Это дает представление о таксономических и функциональных композициях сообщества, а также о дифференциальном анализе экспрессии генов.
●Обширная аннотация генов:Использование актуальных баз данных функций гена для информативной информации об экспрессии генов микробных сообществ.
●Поддержка после продажи:Наше обязательство выходит за рамки завершения проекта с 3-месячным периодом послепродажного обслуживания. В течение этого времени мы предлагаем последующее наблюдение за проектом, помощь в устранении неполадок и сеансы вопросов и ответов для решения любых запросов, связанных с результатами.
Спецификации обслуживания
| Платформа секвенирования | Стратегия секвенирования | Данные рекомендуются | Контроль качества данных |
| Illumina novaseq | PE150 | 12 ГБ | Q30≥85% |
Требования к выборке
| Концентрация (нг/мкл) | Общее количество (мкг) | Объем (мкл) | OD260/280 | OD260/230 | Рин |
| ≥50 | ≥1,0 | ≥20 | 1,8-2,0 | 1,0-2,5 | ≥6,5 |
Обслуживание рабочего перехода

Образец доставки

Библиотека строительство

Секвенирование

Анализ данных

Послепродажные услуги
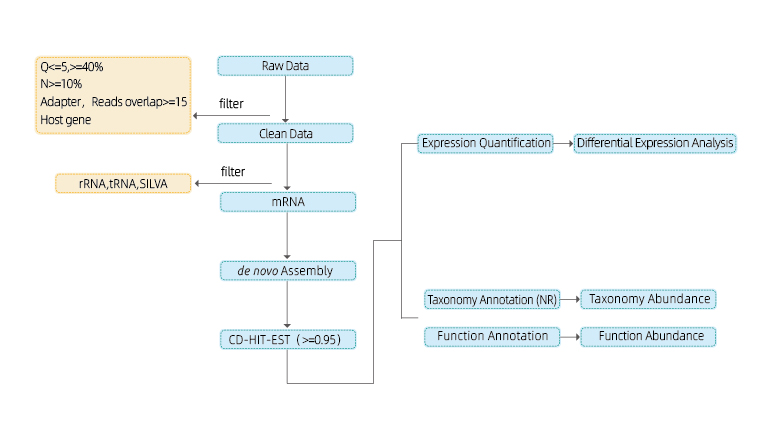
Включает следующий анализ:
● Секвенирование управления качеством данных
● Сборка стенограммы
● Таксономическая аннотация и изобилие
● Функциональная аннотация и изобилие
● Количественная оценка экспрессии и дифференциальный анализ
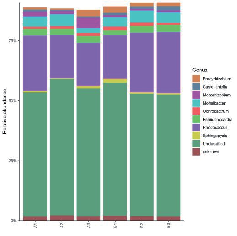
Таксономическое распределение каждой выборки:
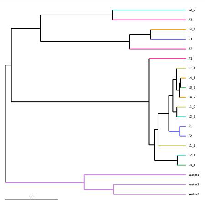
Бета -анализ разнообразия: Upgma
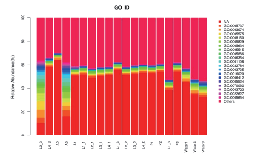
Функциональная аннотация - изобилуйте изобилием
Дифференциальная таксономия численность - левша
Исследуйте достижения, облегченные службами секвенирования Meta Transcriptomics от Bmkgene, посредством кураторской коллекции публикаций.
Lu, Z. et al. (2023) «Устойчивость к кислоте бактерий, использующих лактат бактерий, порядок бактероидов способствует профилактике ацидоза рубца у коз, адаптированных к диете с высоким концентратом»,Питание животных, 14, с. 130–140. doi: 10.1016/j.aninu.2023.05.006.
Песня, Z. et al. (2017) «Раскрытие основной функциональной микробиоты в традиционной твердотельной ферментации путем высокопроизводительных ампликонов и секвенирования метаттранскриптиза»,Границы в микробиологии, 8 (июль). doi: 10.3389/fmicb.2017.01294/full.
Wang, W. et al. (2022) «Новые миковирусы, обнаруженные из обследования фитопатогенного гриба с фитопатогенной альтернарией»,Вирусы, 14 (11), с. 2552. doi: 10.3390/v14112552/s1.
Вей, Дж. И соавт. (2022) «Параллельный анализ метатранскриптома показывает деградацию вторичных метаболитов растений по жукам и их симбионтам кишечника»,Молекулярный Экология, 31 (15), с. 3999–4016. doi: 10.1111/mec.16557.