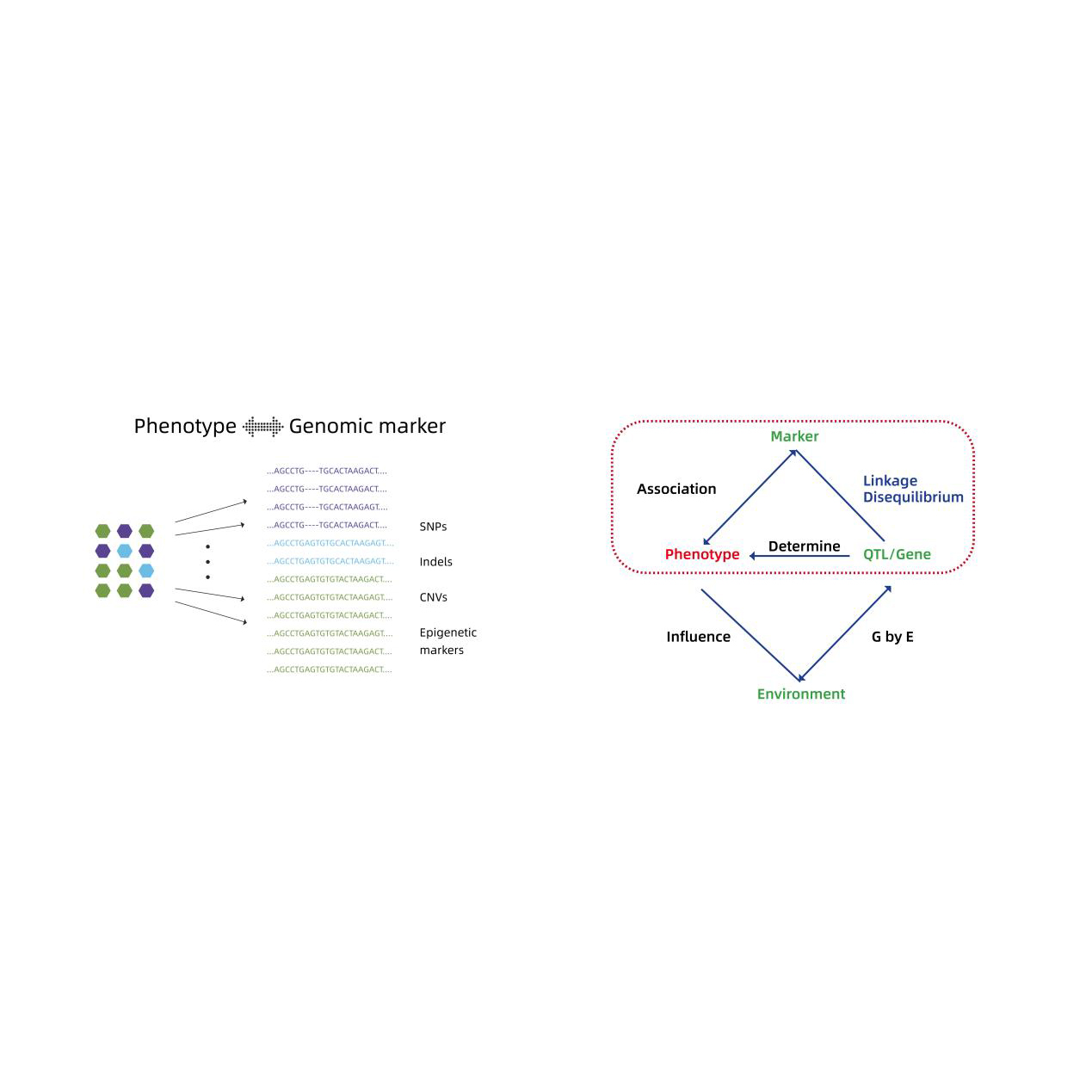
Анализ ассоциации по всему геному
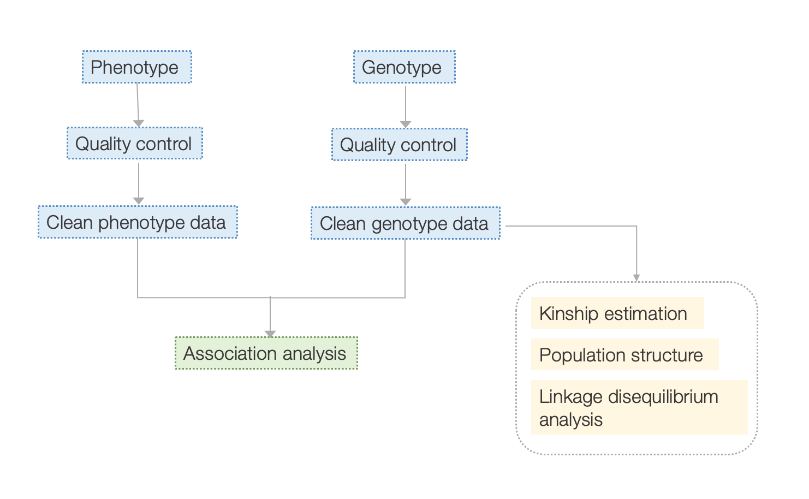
Рабочий процесс

Сервисные преимущества
●Обширные экспертизы и записи публикации: Благодаря накопленному опыту в GWAS BMKGene завершил сотни видовых проектов в популяционных исследованиях GWAS, помощи исследователям опубликовать более 100 статей, а совокупный фактор воздействия достиг 500.
● Комплексный анализ биоинформатики: Рабочий процесс включает анализ ассоциации SNP-признаков, предоставление набора генов-кандидатов и их соответствующую функциональную аннотацию.
●Высококвалифицированная команда биоинформатики и короткий цикл анализа: С большим опытом в анализе передового геномики команда BMKGene проводит всесторонний анализ с быстрым временем обращения.
●Поддержка после продажи:Наше обязательство выходит за рамки завершения проекта с 3-месячным периодом послепродажного обслуживания. В течение этого времени мы предлагаем последующее наблюдение за проектом, помощь в устранении неполадок и сеансы вопросов и ответов для решения любых запросов, связанных с результатами.
Технические характеристики и требования услуг
| Тип секвенирования | Рекомендуемый масштаб населения | Стратегия секвенирования | Нуклеотидные требования |
| Секвенирование всего генома | 200 образцов | 10x | Концентрация: ≥ 1 нг/ мкл Общая сумма ≥ 30 нг Ограниченное или нет деградации или загрязнения |
| Специфический локус амплифицированный фрагмент (SLAF) | Глубина тега: 10x Количество тегов: <400 МБ: рекомендуется WGS <1 ГБ: 100K теги 1 ГБ > 2 ГБ: 300K теги Макс 500 тыс. Теги | Концентрация ≥ 5 нг/мкл Общая сумма ≥ 80 нг Nanodrop od260/280 = 1,6-2,5 Агарозный гель: нет или ограниченного деградации или загрязнения
|
Выбор материала



Различные разновидности, подвиды, Landraces/Genebanks/смешанные семьи/дикие ресурсы
Разные разновидности, подвиды, земли
Семья с половиной сжима/семья/дикие ресурсы
Обслуживание рабочего перехода

Дизайн эксперимента

Образец доставки

РНК -экстракция

Библиотека строительство

Секвенирование

Анализ данных

Послепродажные услуги
Включает следующий анализ:
- Анализ ассоциации по всему геному: LM, LMM, EMMAX, Fastlmm Model
- Функциональная аннотация генов кандидатов
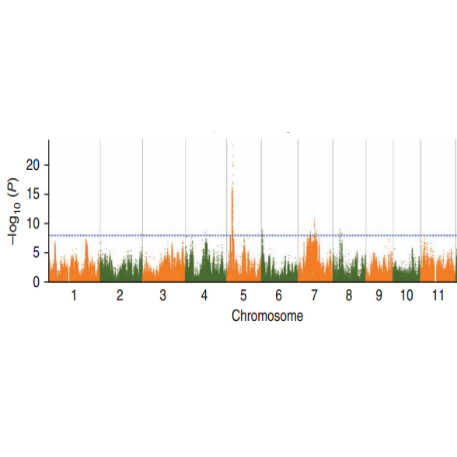
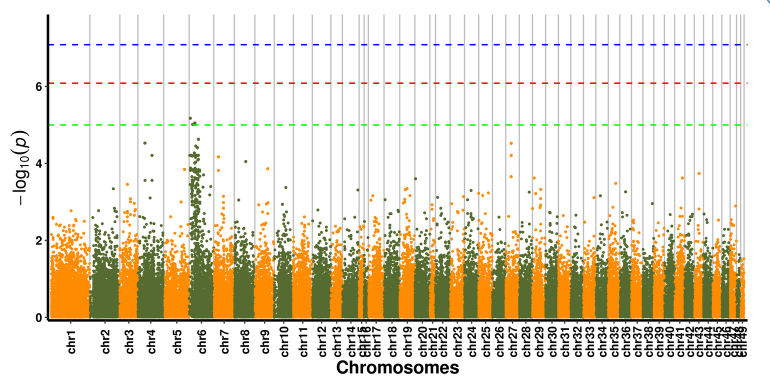
Анализ ассоциации SNP-признаков-участок Манхэттена
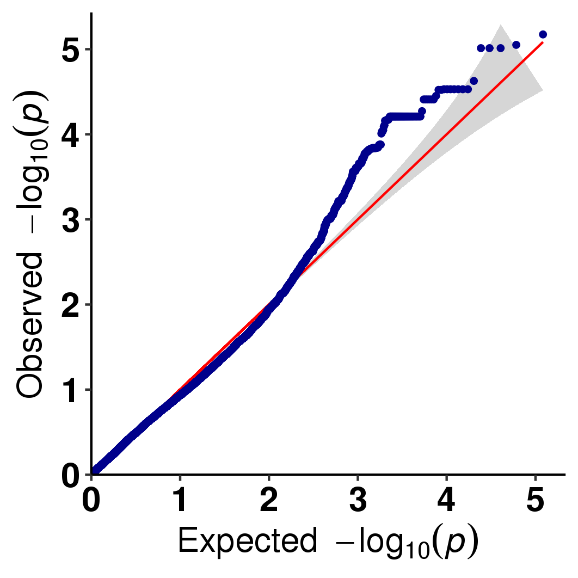
Анализ ассоциации SNP-признаков-график QQ
Исследуйте достижения, облегченные службами DE GWAS от BMKGENE, посредством кураторской коллекции публикаций:
LV, L. et al. (2023) «Понимание генетической основы толерантности к аммиаку в Razor Clam Sinonovacula constricta путем ассоциации по всему геному»,Аквакультура, 569, с. 739351. DOI: 10.1016/j.aquaculture.2023.739351.
Li, X. et al. (2022) «Мультиомический анализ 398 сетевых просочных образцов выявляет геномные области, связанные с одомашниванием, признаками метаболита и противовоспалительным эффектом»,Молекулярное растение, 15 (8), с. 1367–1383. doi: 10.1016/j.molp.2022.07.003.
Li, J. et al. (2022) «Картографирование ассоциации по всему геному безжалостно едва фенотипов в засухе»,Границы в науке о растениях, 13, с. 924892. DOI: 10.3389/fpls.2022.924892/bibtex.
Zhao, X. et al. (2021) 'GMST1, который кодирует сульфотрансфераза, обеспечивает устойчивость к штаммам вируса сои мозаики G2 и G3',Растение, ячейка и окружающая среда, 44 (8), с. 2777–2792. doi: 10.1111/pce.14066.