Полноразмерное секвенирование мРНК – нанопора
Функции
● Захват мРНК поли-А с последующим синтезом кДНК и подготовкой библиотеки.
● Секвенирование полноразмерных транскриптов.
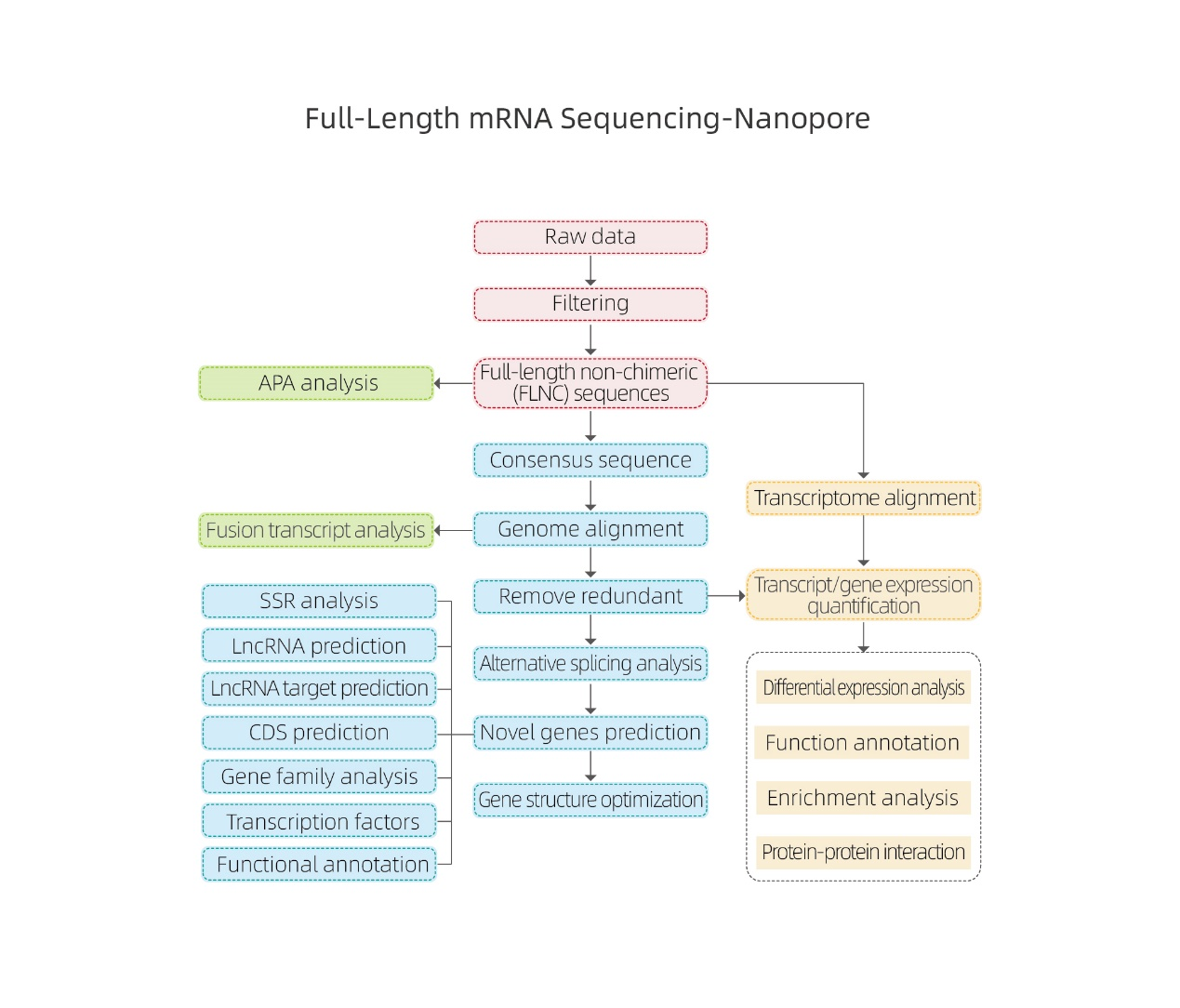
● Биоинформатический анализ, основанный на сопоставлении с эталонным геномом.
● Биоинформатический анализ включает не только экспрессию на уровне генов и изоформ, но также анализ днРНК, слияний генов, полиаденилирования и структуры генов.
Преимущества сервиса
●Количественная оценка экспрессии на уровне изоформы: обеспечивает детальный и точный анализ экспрессии, выявляя изменения, которые могут быть замаскированы при анализе всей экспрессии гена.
●Снижение требований к данным:По сравнению с секвенированием следующего поколения (NGS), секвенирование нанопор требует меньших требований к данным, что позволяет обеспечить эквивалентные уровни насыщения количественной оценки экспрессии генов меньшими данными.
●Более высокая точность количественного определения экспрессии: как на уровне гена, так и на уровне изоформ
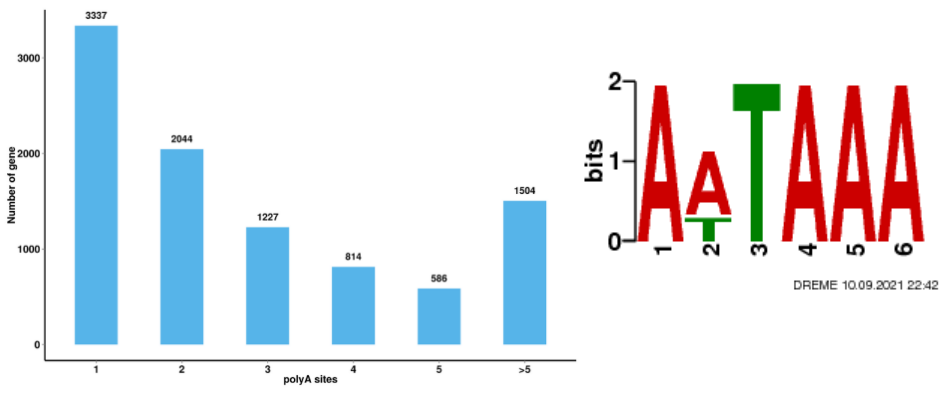
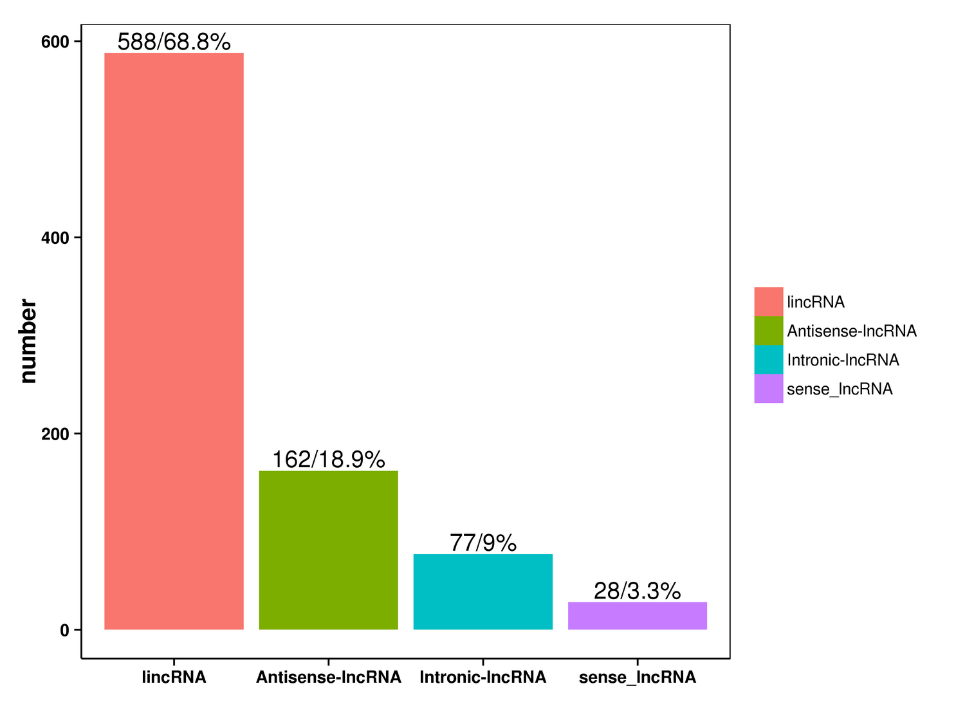
●Идентификация дополнительной транскриптомной информации: альтернативное полиаденилирование, слитые гены и дцнРНК и их гены-мишени.
●Обширная экспертиза: Наша команда привносит богатый опыт в каждый проект, выполнив более 850 проектов полноразмерного транскриптома Nanopore и обработав более 8000 образцов.
●Послепродажная поддержка: наши обязательства выходят за рамки завершения проекта и предусматривают трехмесячный период послепродажного обслуживания. В течение этого времени мы предлагаем дальнейшее сопровождение проекта, помощь в устранении неполадок и сеансы вопросов и ответов для решения любых вопросов, связанных с результатами.
Требования к образцам и доставка
| Библиотека | Стратегия секвенирования | Рекомендуемые данные | Контроль качества |
| Поли А обогащенный | Иллюмина PE150 | 6/12 ГБ | Средний балл качества: Q10 |
Образец требований:
Нуклеотиды:
| Конц. (нг/мкл) | Количество (мкг) | Чистота | Честность |
| ≥ 100 | ≥ 1,0 | ОД260/280=1,7-2,5 ОД260/230=0,5-2,5 Ограниченное или полное отсутствие загрязнения белками или ДНК на геле. | Для растений: RIN≥7,0; Для животных: РИН≥7,5; 5,0≥28S/18S≥1,0; ограниченное или отсутствующее повышение базовой линии |
● Растения:
Корень, стебель или лепесток: 450 мг
Лист или семя: 300 мг
Фрукты: 1,2 г
● Животное:
Сердце или кишечник: 300 мг
Внутренности или мозг: 240 мг
Мышцы: 450 мг
Кости, волосы или кожа: 1 г
● Членистоногие:
Насекомые: 6 г
Ракообразные: 300 мг
● Цельная кровь: 1 тюбик
● Ячейки: 106 клетки
Рекомендуемая доставка образцов
Контейнер: центрифужная пробирка объемом 2 мл (не рекомендуется использовать фольгу).
Маркировка образца: Группа + повтор, например, А1, А2, А3; Б1, Б2, Б3.
Отгрузка:
1. Сухой лед: Пробы необходимо упаковать в мешки и закопать в сухой лед.
2. Пробирки RNAstable: образцы РНК можно сушить в пробирках для стабилизации РНК (например, RNAstable®) и транспортировать при комнатной температуре.
Рабочий процесс обслуживания
Нуклеотиды:

Доставка образца

Строительство библиотеки

Секвенирование

Анализ данных

Послепродажное обслуживание
Рабочий процесс обслуживания
Салфетка:

Дизайн эксперимента

Доставка образца

экстракция РНК

Строительство библиотеки

Секвенирование

Анализ данных

Послепродажное обслуживание
● Обработка необработанных данных.
● Идентификация стенограммы
● Альтернативный сплайсинг
● Количественная оценка экспрессии на уровне генов и изоформ.
● Анализ дифференциальных выражений.
● Аннотация и расширение функций (DEG и DET).
Альтернативный анализ сплайсинга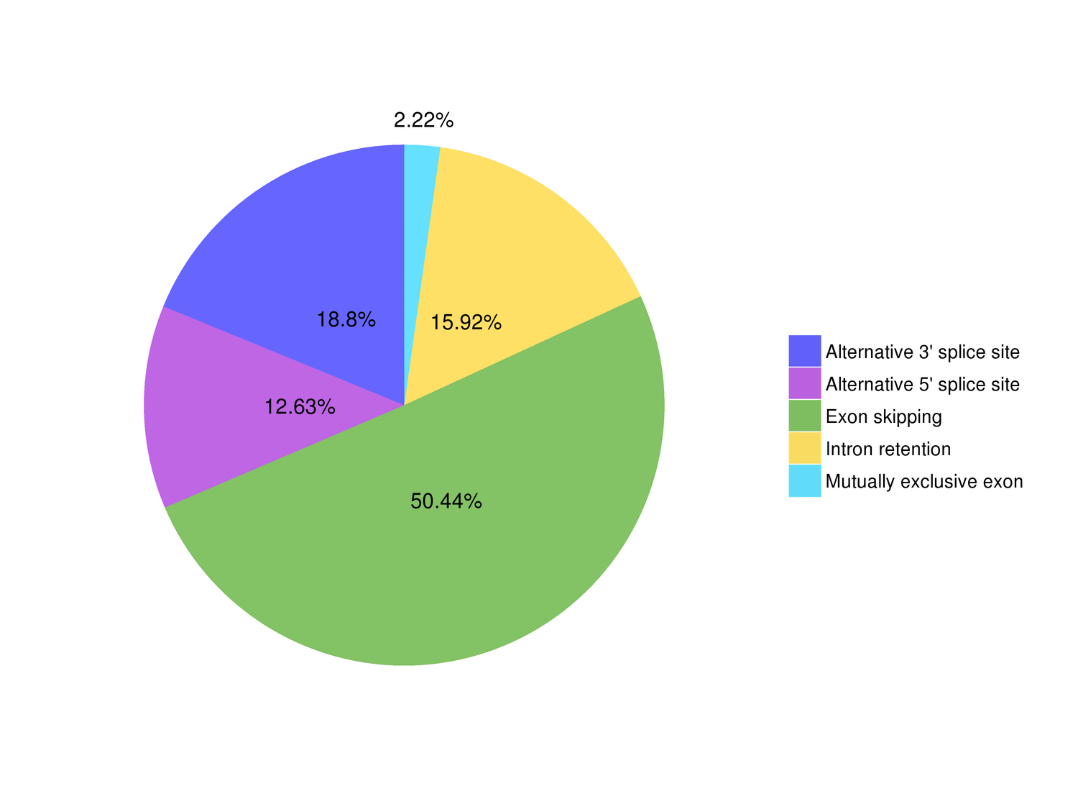 Альтернативный анализ полиаденилирования (APA)
Альтернативный анализ полиаденилирования (APA)
предсказание днРНК
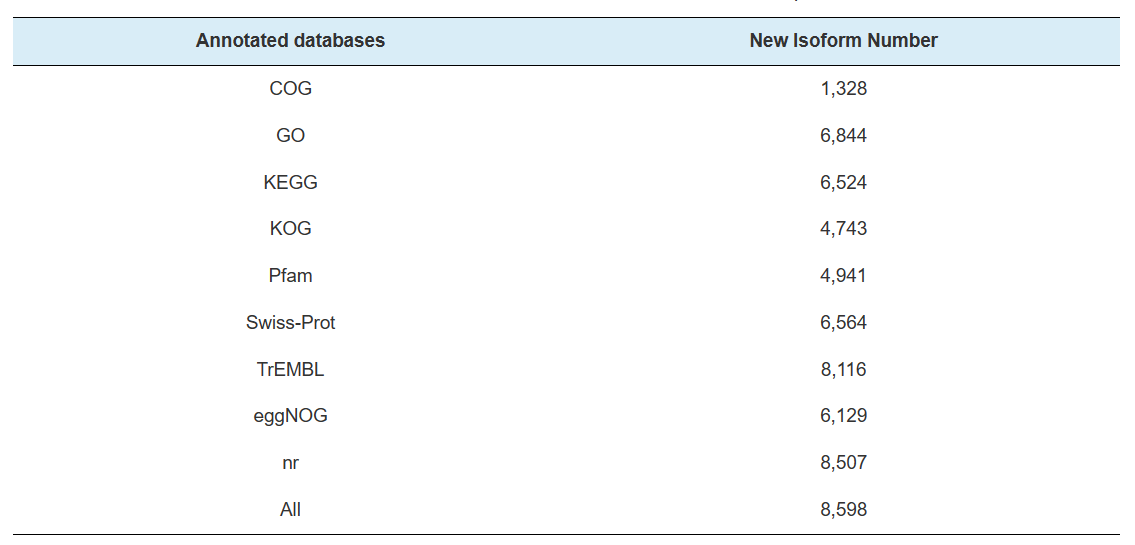
Аннотация новых генов
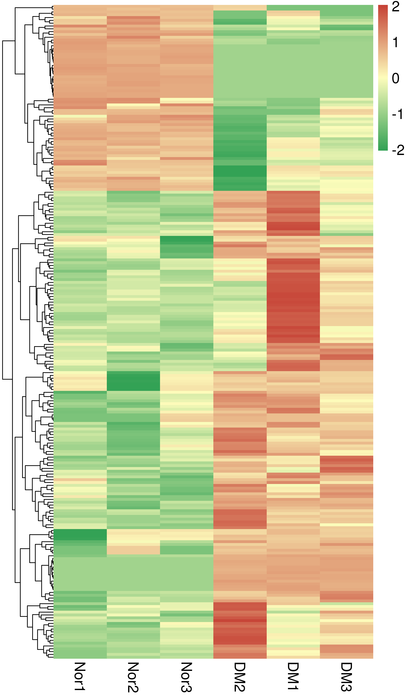
Кластеризация DET
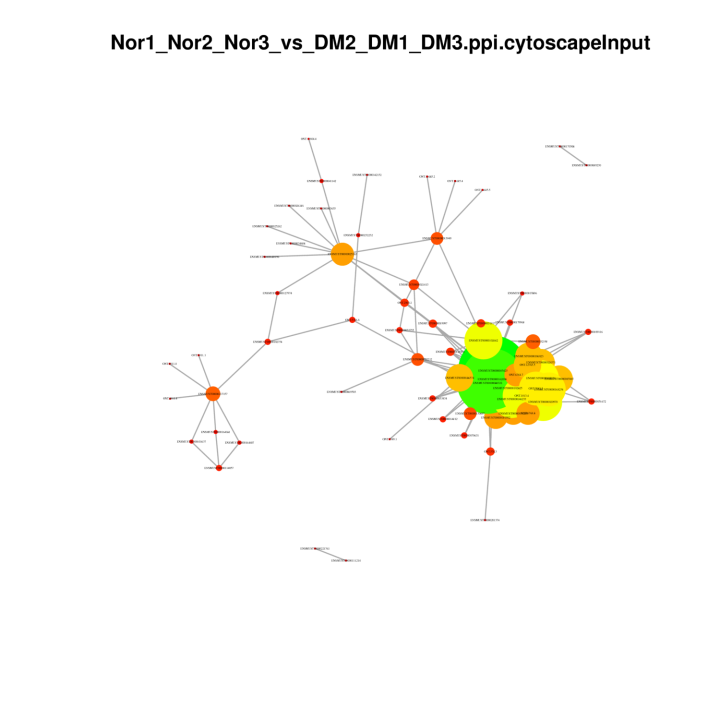
Белко-белковые сети в ДЭГ
Изучите достижения, достигнутые благодаря услугам BMKGene по полноразмерному секвенированию мРНК Nanopore, с помощью тщательно подобранной коллекции публикаций.
Гонг Б. и др. (2023) «Эпигенетическая и транскрипционная активация секреторной киназы FAM20C как онкогена при глиоме», Journal of Genetics and Genomics, 50 (6), стр. 422–433. дои: 10.1016/J.JGG.2023.01.008.
Он, З. и др. (2023) «Полноразмерное секвенирование транскриптома лимфоцитов, реагирующих на IFN-γ, выявило искаженный Th1 иммунный ответ у камбалы (Paralichthys olivaceus)», Fish & Shellfish Immunology, 134, стр. 108636. doi: 10.1016/J.FSI.2023.108636.
Ма, Ю. и др. (2023) «Сравнительный анализ методов секвенирования РНК PacBio и ONT для идентификации яда Nemopilema Nomurai», Genomics, 115(6), стр. 110709. doi: 10.1016/J.YGENO.2023.110709.
Ю, Д. и др. (2023) «Анализ Nano-seq выявляет различную функциональную тенденцию между экзосомами и микровезикулами, полученными из hUMSC», Stem Cell Research and Therapy, 14 (1), стр. 1–13. doi: 10.1186/S13287-023-03491-5/ТАБЛИЦЫ/6.