Пространственный транскриптом BMKMANU S1000
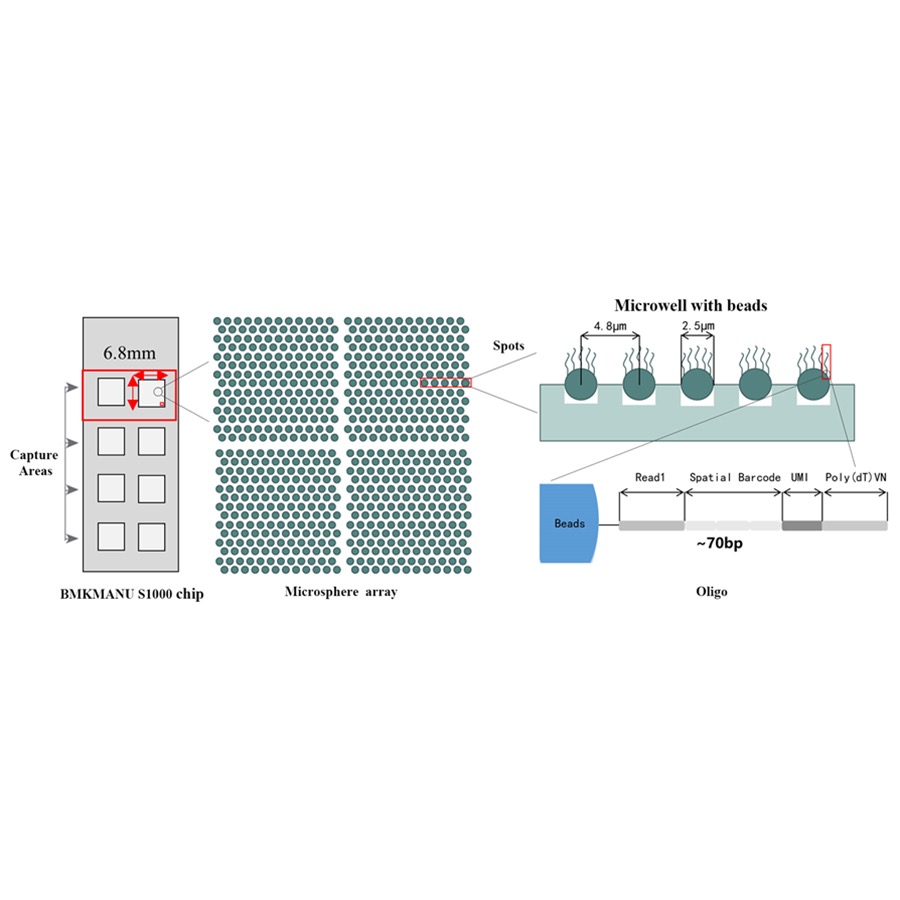
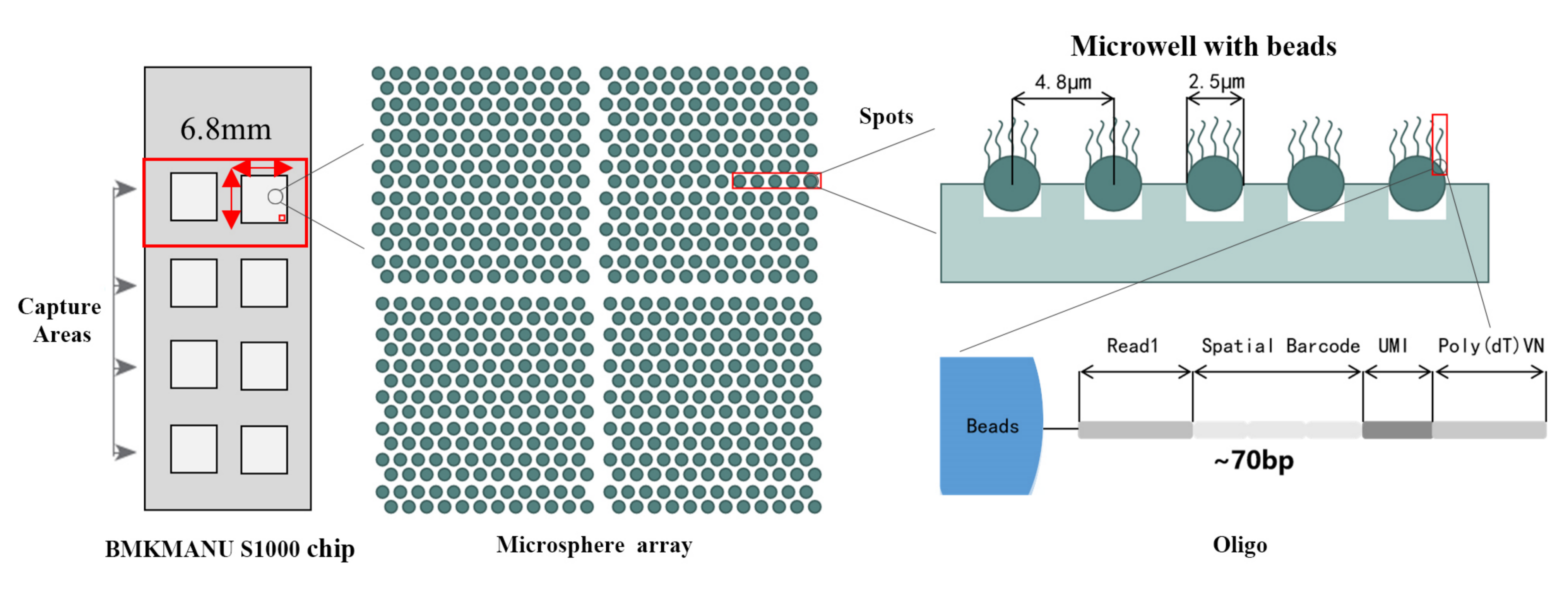
Техническая схема пространственного транскриптома BMKMANU S1000
Функции
● Разрешение: 5 мкм.
● Диаметр пятна: 2,5 мкм.
● Количество рекламных мест: около 2 миллионов.
● 3 возможных формата области захвата: 6,8 мм * 6,8 мм, 11 мм * 11 мм или 15 мм * 20 мм.
● Каждая бусина со штрих-кодом наполнена праймерами, состоящими из 4 секций:
поли(дТ) хвост для прайминга мРНК и синтеза кДНК
Уникальный молекулярный идентификатор (UMI) для коррекции систематической ошибки амплификации.
Пространственный штрих-код
Последовательность связывания праймера секвенирования частичного чтения 1
● H&E и флуоресцентное окрашивание срезов.
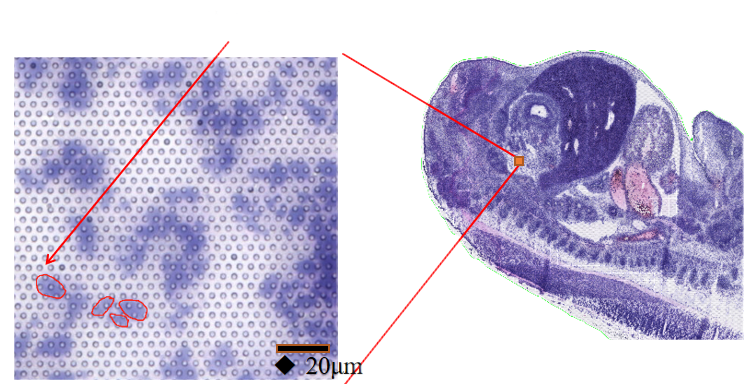
● Возможность использованиятехнология сегментации клеток: интеграция окрашивания H&E, флуоресцентного окрашивания и секвенирования РНК для определения границ каждой клетки и правильного назначения экспрессии генов для каждой клетки.
Преимущества BMKMANU S1000
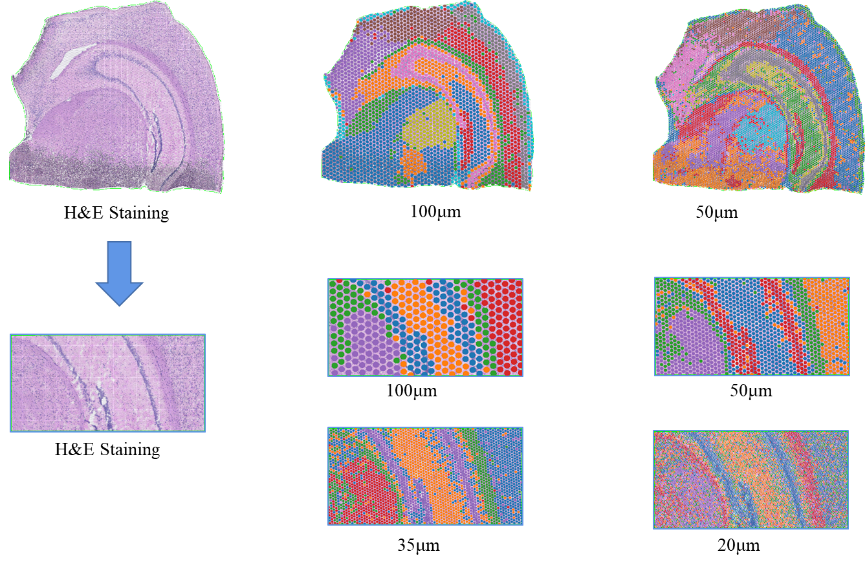
●Субклеточное разрешение: каждая область захвата содержала> 2 миллиона пространственных пятен со штрих-кодом диаметром 2,5 мкм и расстоянием между центрами пятен 5 мкм, что позволяло проводить пространственный транскриптомный анализ с субклеточным разрешением (5 мкм).
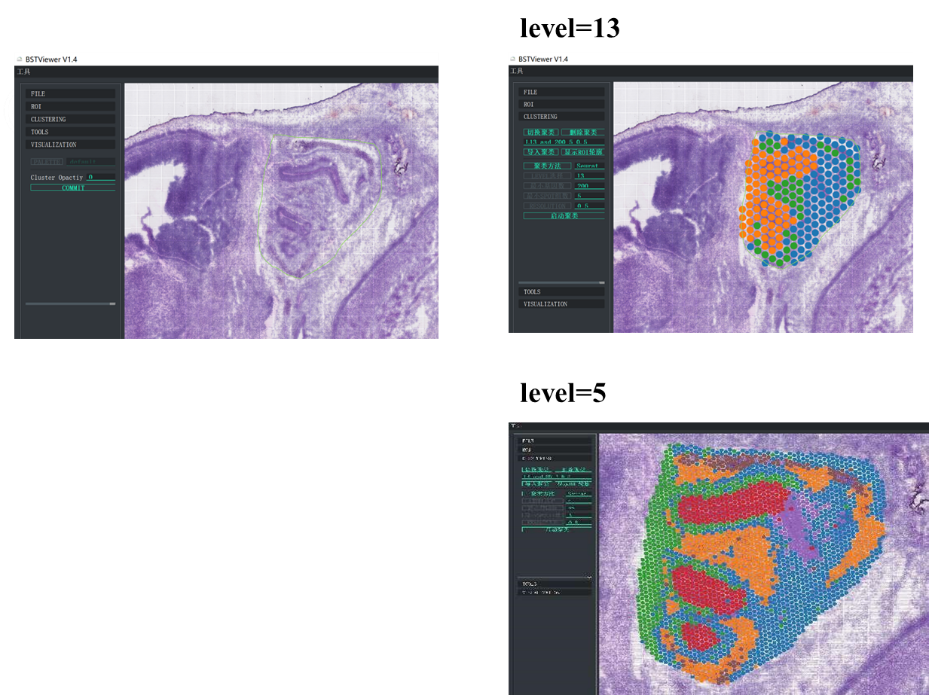
●Многоуровневый анализ разрешения:Гибкий многоуровневый анализ в диапазоне от 100 до 5 мкм для определения различных особенностей тканей с оптимальным разрешением.
●Возможность использования технологии сегментации клеток «три в одном слайде»:Сочетая флуоресцентное окрашивание, окрашивание H&E и секвенирование РНК на одном предметном стекле, наш алгоритм анализа «три в одном» позволяет идентифицировать границы клеток для последующей транскриптомики на основе клеток.
●Совместимость с несколькими платформами секвенирования: Доступны как NGS, так и секвенирование длительного считывания.
●Гибкая конструкция активных зон захвата 1–8: Размер области захвата является гибким, можно использовать 3 формата (6,8 мм * 6,8 мм, 11 мм * 11 мм и 15 мм * 20 мм).
●Комплексное обслуживание: Он объединяет все этапы, основанные на опыте и навыках, включая криосекционирование, окрашивание, оптимизацию тканей, пространственное штрих-кодирование, подготовку библиотеки, секвенирование и биоинформатику.
●Комплексная биоинформатика и удобная визуализация результатов:пакет включает в себя 29 анализов и более 100 высококачественных рисунков в сочетании с использованием программного обеспечения собственной разработки для визуализации и настройки разделения клеток и точечной кластеризации.
●Индивидуальный анализ и визуализация данных: доступен для различных исследовательских запросов
●Высококвалифицированная техническая команда: с опытом работы с более чем 250 типами тканей и более чем 100 видами, включая человека, мышь, млекопитающих, рыб и растений.
●Обновления в реальном времени по всему проекту: с полным контролем хода эксперимента.
●Дополнительный совместный анализ с секвенированием одноклеточной мРНК
Технические характеристики услуги
|
Образец Требования
| Библиотека |
Стратегия секвенирования
| Рекомендуемые данные | Контроль качества |
| Криообразцы, встроенные в ОКТ, 3 блока на образец | Библиотека кДНК S1000 | Illumina PE150 (доступны другие платформы) | 100 тыс. операций чтения PE на 100 мкм ( 60-150 Гб ) | РИН>7 |
Для получения более подробной информации о руководстве по подготовке проб и рабочем процессе обслуживания, пожалуйста, свяжитесь сэксперт БМКГЕНЕ
Рабочий процесс обслуживания
На этапе подготовки проб проводится первоначальная пробная экстракция РНК, чтобы гарантировать возможность получения высококачественной РНК. На этапе оптимизации ткани срезы окрашиваются и визуализируются, а также оптимизируются условия пермеабилизации для высвобождения мРНК из ткани. Оптимизированный протокол затем применяется во время создания библиотеки с последующим секвенированием и анализом данных.
Полный рабочий процесс обслуживания включает обновления в режиме реального времени и подтверждения клиентов для поддержания оперативного цикла обратной связи, гарантируя бесперебойное выполнение проекта.
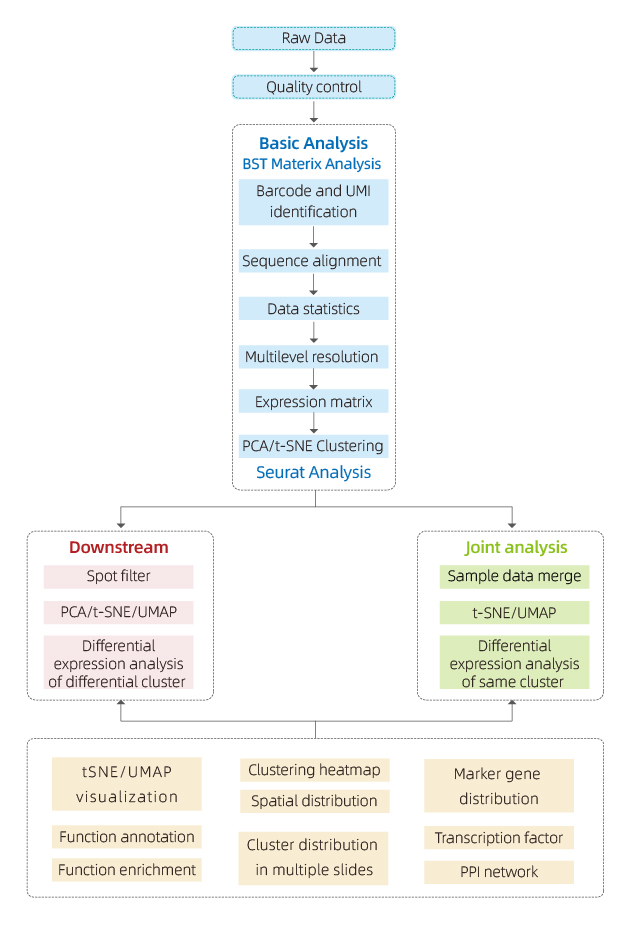
Данные, генерируемые BMKMANU S1000, анализируются с использованием программного обеспечения «BSTMatrix», независимо разработанного BMKGENE, которое генерирует матрицу экспрессии генов. На основе этого создается стандартный отчет, который включает контроль качества данных, анализ внутренней выборки и межгрупповой анализ.
● Контроль качества данных:
Вывод данных и распределение показателей качества
Обнаружение генов на точку
Покрытие тканей
● Внутренний анализ пробы:
Генное богатство
Точечная кластеризация, включая анализ уменьшенных размерностей
Анализ дифференциальной экспрессии между кластерами: идентификация маркерных генов
Функциональная аннотация и обогащение маркерных генов
● Межгрупповой анализ:
Рекомбинация пятен из обоих образцов (например, больного и контрольного) и повторная кластеризация
Идентификация маркерных генов для каждого кластера
Функциональная аннотация и обогащение маркерных генов
Дифференциальное выражение одного и того же кластера между группами
Кроме того, компания BMKGENE разработала «BSTViewer» — удобный инструмент, который позволяет пользователю визуализировать экспрессию генов и определять кластеризацию с различным разрешением.
BMKGene разработала программное обеспечение для удобной визуализации
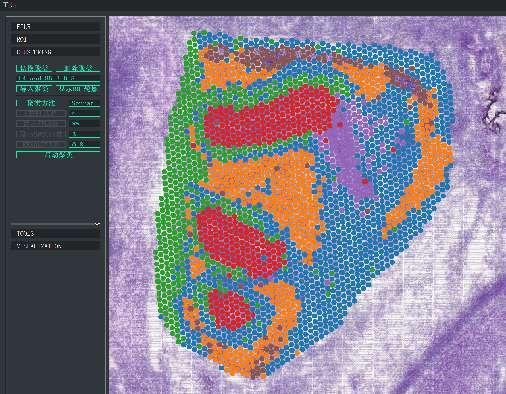
BSTViewer точечная кластеризация с многоуровневым разрешением
BSTCellViewer: автоматическое и ручное разделение ячеек
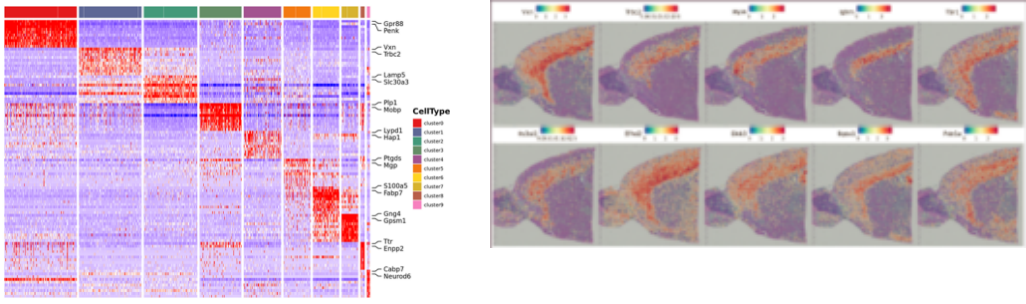
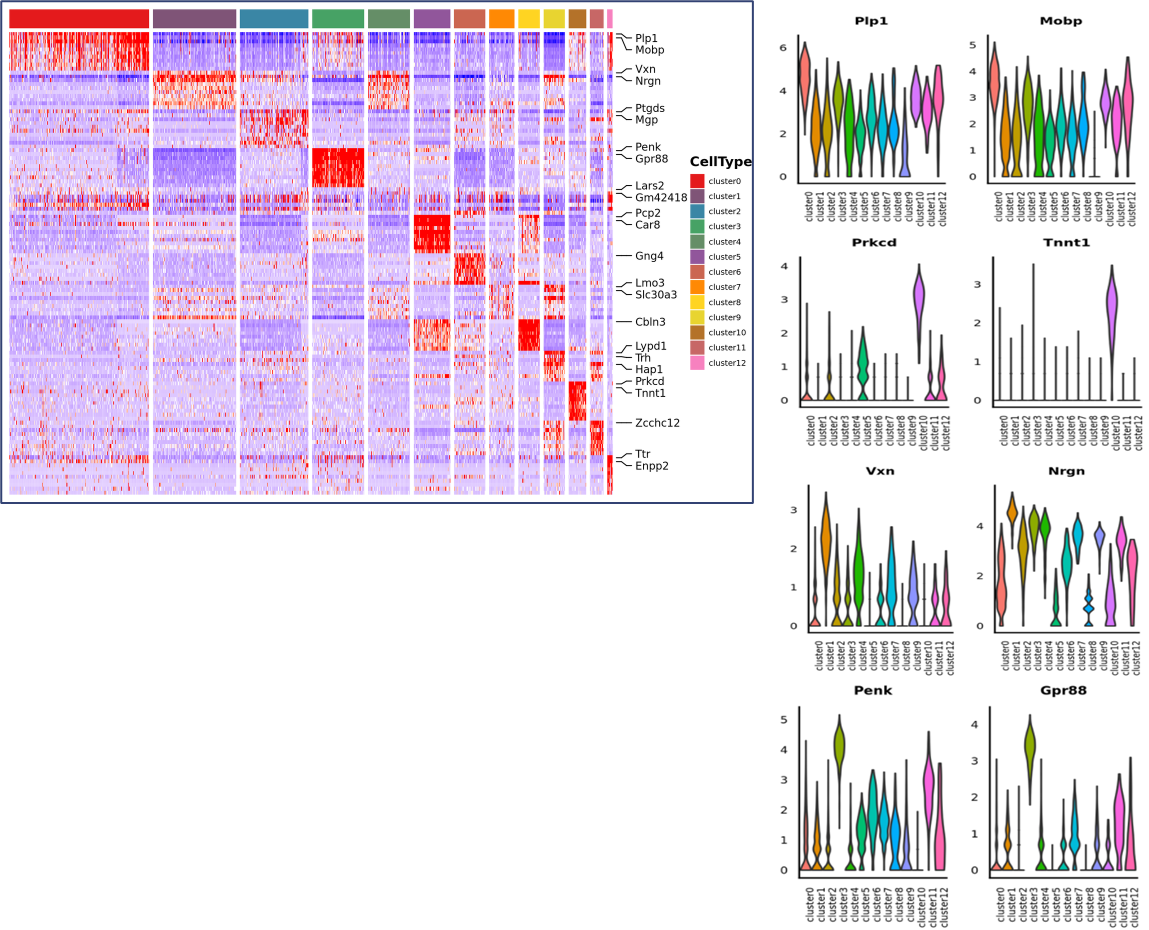
Анализ внутренней пробы
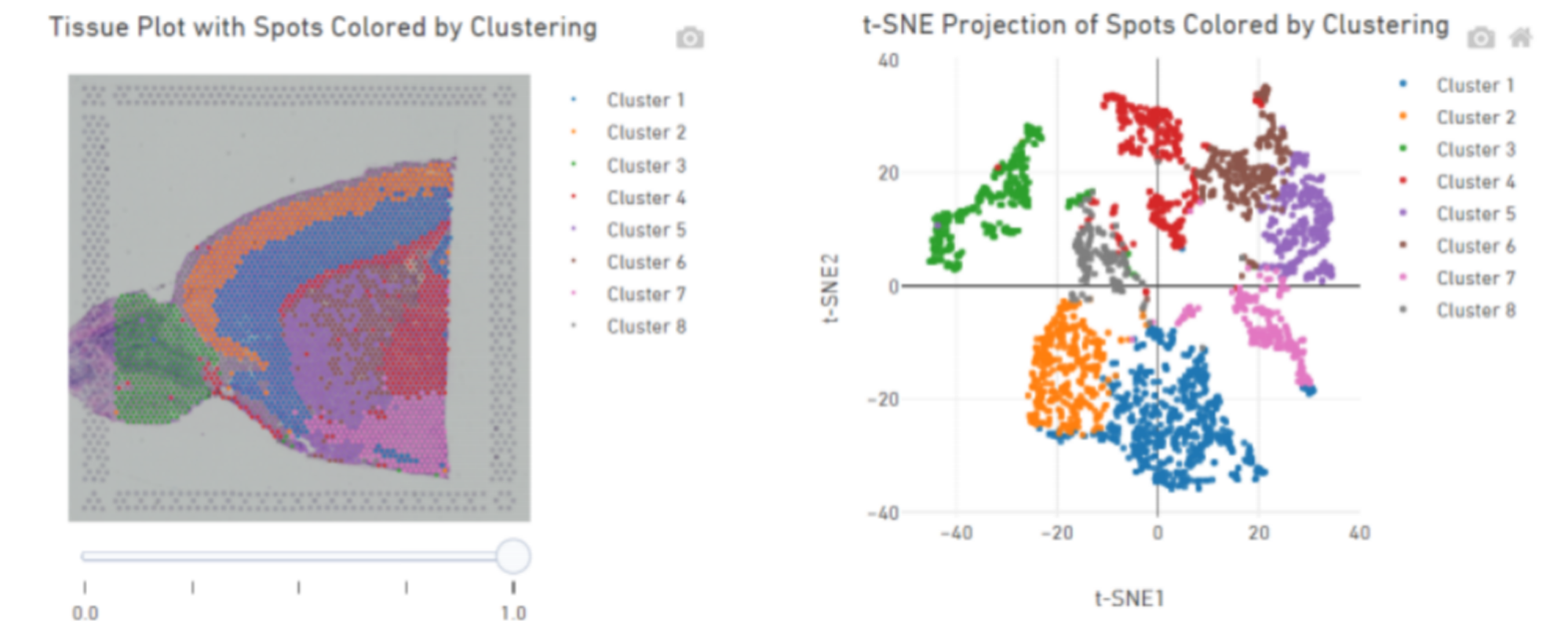
Точечная кластеризация:
Идентификация маркерных генов и пространственное распределение:
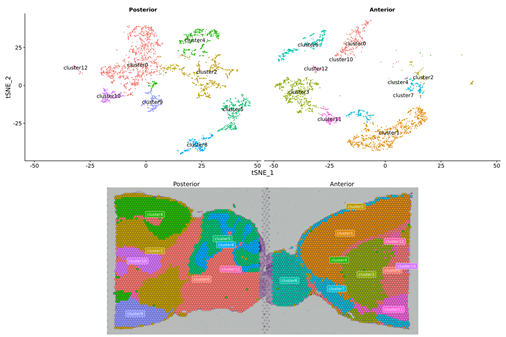
Межгрупповой анализ
Комбинация данных из обеих групп и повторной кластеризации:
Маркерные гены новых кластеров:
Узнайте о достижениях, достигнутых благодаря услугам пространственной транскриптомики BMKGene с помощью технологии BMKManu S1000, в этой избранной публикации:
Сонг, X. и др. (2023) «Пространственная транскриптомика обнаруживает индуцированные светом клетки хлоренхимы, участвующие в стимулировании регенерации побегов каллуса томата»,Труды Национальной академии наук Соединенных Штатов Америки., 120(38), с. е2310163120. дои: 10.1073/pnas.2310163120
Вы, Ю. и др. (2023) «Систематическое сравнение методов пространственной транскриптомики на основе секвенирования»,bioRxiv, с. 2023.12.03.569744. дои: 10.1101/2023.12.03.569744.