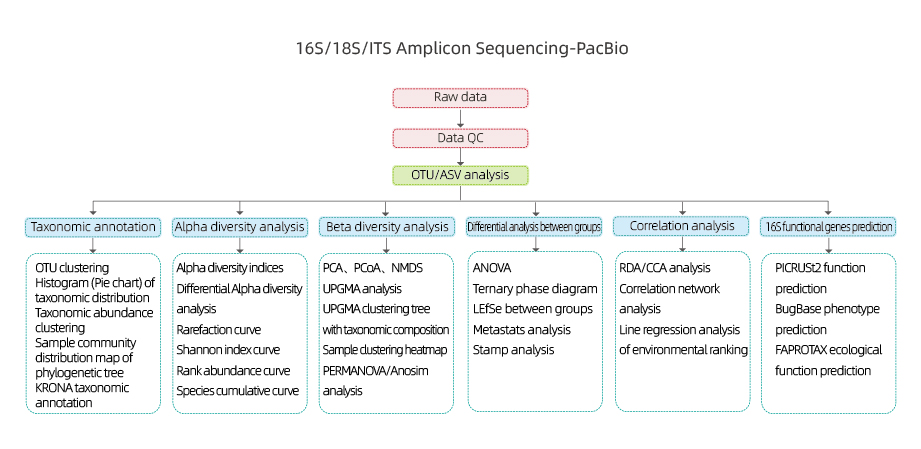
16S/18S/ITS Amplicon Sequencing-Pacbio
Сервисные функции
● Платформа секвенирования: Pacbio Revio
● Режим секвенирования: CCS (HIFI читает)
● Амплификация целевой области с последующим тандемным связыванием ампликонов перед подготовкой библиотеки Hifi Smrt Bell
Сервисные преимущества
●Более высокое таксономическое разрешение: tСеквенирование коротко-ампликонов Хана,обеспечение более высокой скорости классификации OTU на уровне видов.
●Высокий точный базовый вызов: Секвенирование режима PACBIO CCS (HIFI читает).
●Без изоляции: Быстрая идентификация микробного состава в образцах окружающей среды.
●Широко применимо: Разнообразные исследования микробного сообщества.
●Комплексный биоинформационный анализ: Последний пакет QIIME2 (количественное понимание микробной экологии) с разнообразным анализом с точки зрения базы данных, аннотации, OTU/ASV.
●Обширный опыт: С тысячами проектов секвенирования Amplicon, проводимых ежегодно, BMKGene приносит более десятилетия опыта, высококвалифицированную группу по анализу, комплексный контент и отличную поддержку после продажи.
Спецификации обслуживания
| Библиотека | Стратегия секвенирования | Данные рекомендуются |
| Ампликон | Pacbio Revio | 30.10.50 k теги (CCS) |
Требования к выборке
| Концентрация (нг/мкл) | Общее количество (мкг) | Объем (мкл) |
| ≥5 | ≥0,3 | ≥20 |
Рекомендуемая доставка образца
Заморозить образцы в жидком азоте в течение 3-4 часов и хранить в жидком азоте или -80 градусов до длительного резервации. Образец доставки с сухим льдом требуется.
Обслуживание рабочего перехода

Образец доставки

Библиотека строительство

Секвенирование

Анализ данных

Послепродажные услуги
Включает следующий анализ:
● Сырой контроль качества данных
● Кластеризация OTU/de-noise (ASV)
● Аннотация OTU
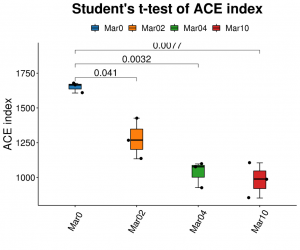
● Анализ альфа -разнообразия: множество индексов, включая Шеннон, Симпсон и ACE.
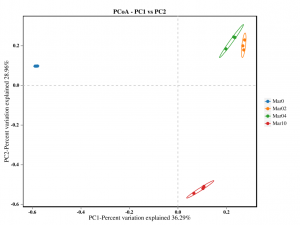
● Анализ бета -разнообразия
● Межгрупповой анализ
● Анализ корреляции: между факторами окружающей среды и составом и разнообразием.
● Прогноз функционального гена 16S
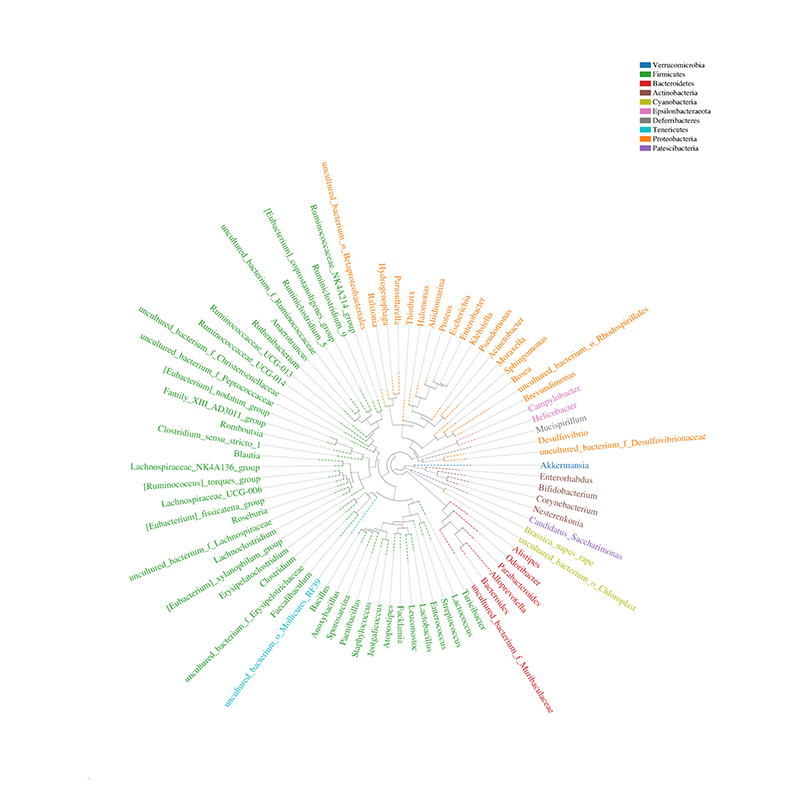
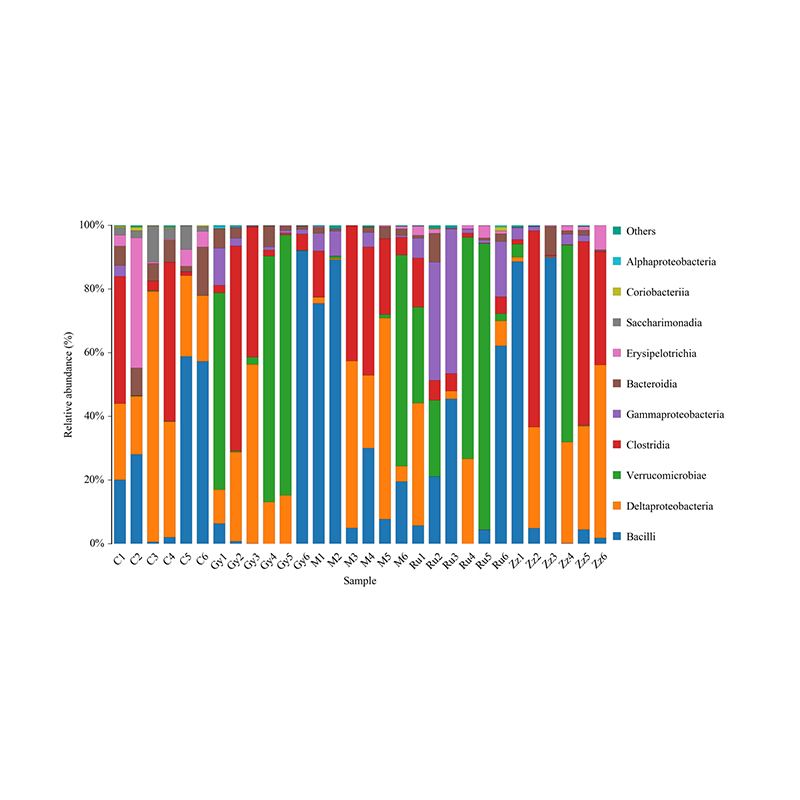
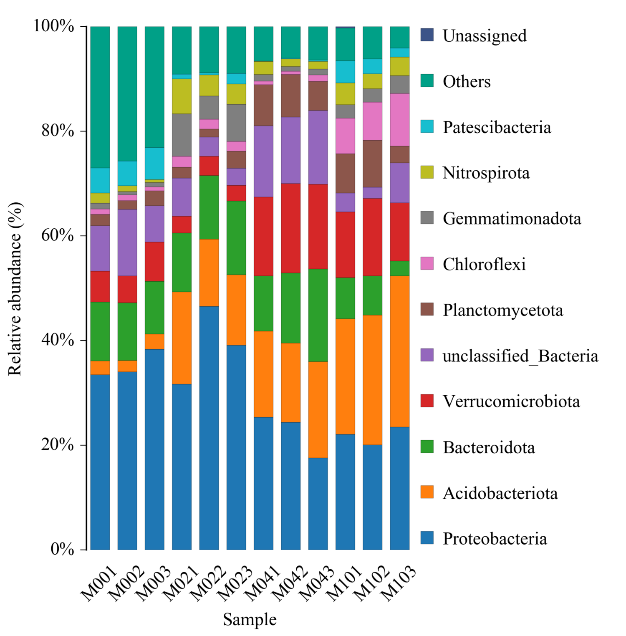
Гистограмма таксономического распределения
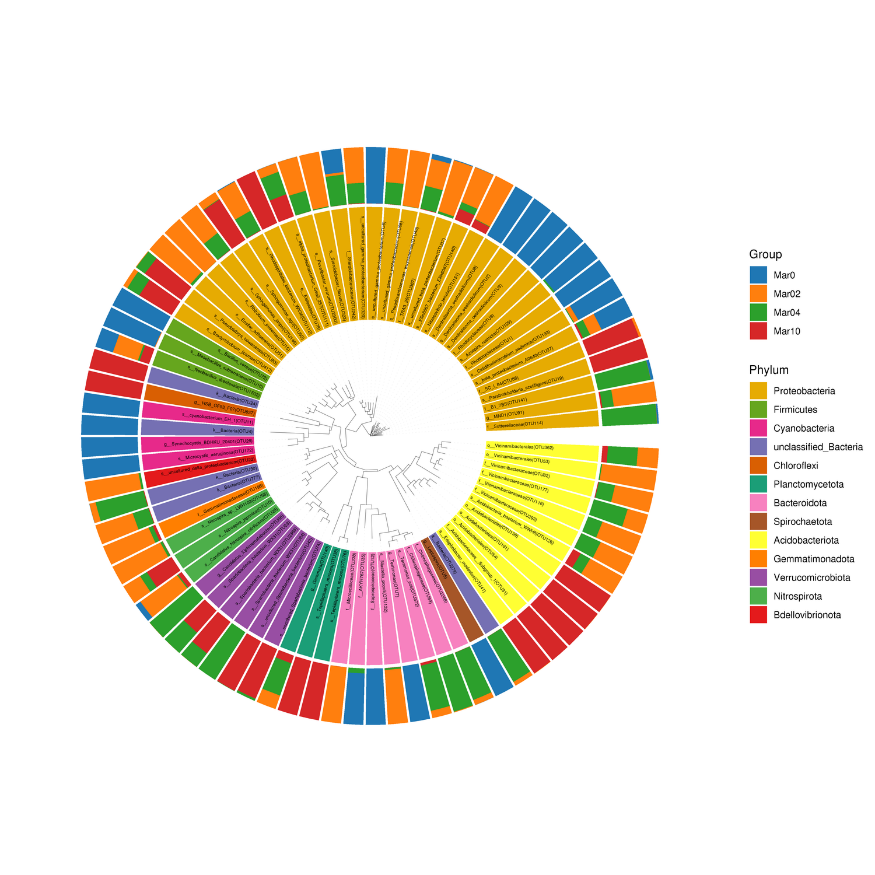
Филогенетическое дерево сообщества
Анализ альфа -разнообразия: туз
Анализ бета -разнообразия: PCOA
Межгрупповой анализ: ANOVA
Исследуйте достижения, облегченные службами секвенирования Amplicon Bmkgene с Pacbio, посредством кураторской коллекции публикаций.
Gao, X. и Wang, H. (2023) «Сравнительный анализ бактериальных профилей рубца и функций во время адаптации к различной фенологии (Regreen vs. Grassy) у альпийских мериновых овец с двумя растущими стадиями на альпийском луге», Ферментация, 9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 (9 1), с. 16. doi: 10.3390/ферментация9010016/s1.
Li, S. et al. (2023) «Захват микробной темной материи в пустынных почвах с использованием метагеномики на основе культуры и анализа высокого разрешения», NPJ BioFilms and Microbiomes 2023 9: 1, 9 (1), стр. 1–14. doi: 10.1038/s41522-023-00439-8.
MU, L. et al. (2022) «Влияние солей жирных кислот на характеристики ферментации, бактериальное разнообразие и аэробную стабильность смешанного силоса, приготовленного с люцерной, рисовой соломенной и пшеничной отруби», Журнал науки о пищевых продуктах и сельском хозяйстве, 102 (4), с. 1475– 1487. doi: 10.1002/jsfa.11482.
Yang, J. et al. (2023) «Взаимодействие между биомаркерами окислительного стресса и микробиотой кишечника в антиоксидантных эффектах экстрактов из Sonchus brachyotus dc. При индуцированном оксазолонам кишечный окислительный стресс у взрослых рыбок данио », Antioxidants 2023, Vol. 12, стр. 192, 12 (1), с. 192. doi: 10.3390/antiox120101192.