10x геномика Visium Spatial Transcriptome
Техническая схема

Функции
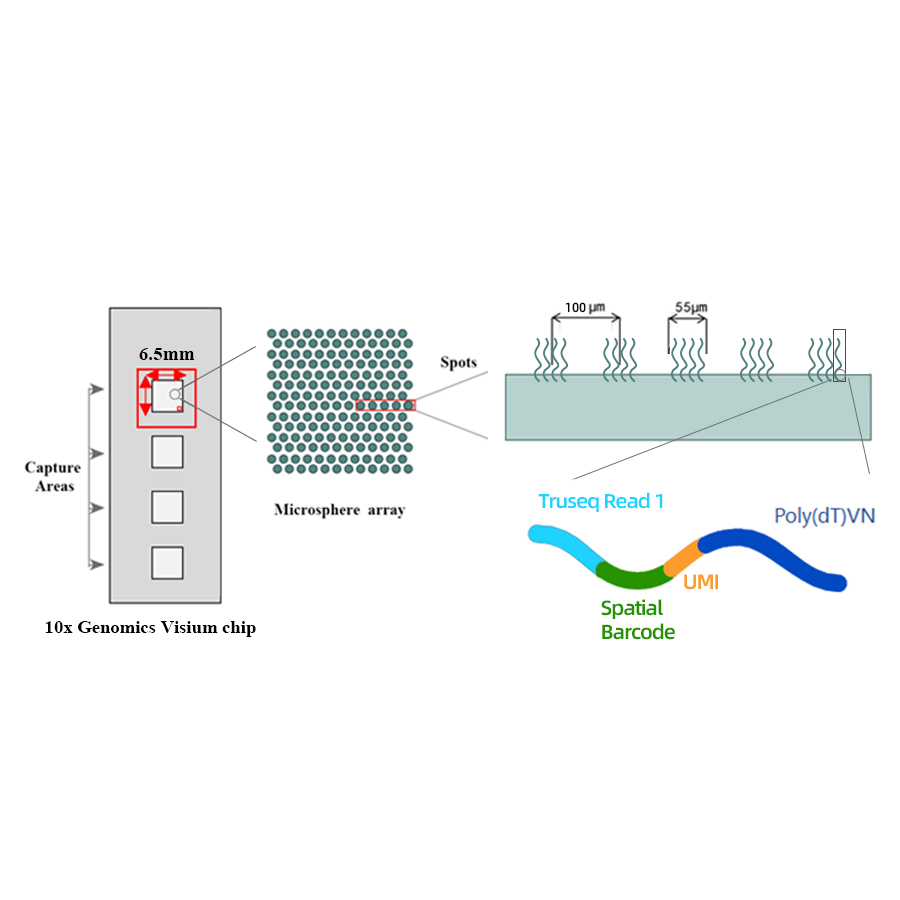
● Разрешение: 100 мкм
● Диаметр пятна: 55 мкм
● Количество пятен: 4992
● Площадь захвата: 6,5 х 6,5 мм
● Каждое пятно штрих -кодирования загружено праймерами, состоящими из 4 секций:
- Поли (DT) хвост для мРНК -праймирования и синтеза кДНК
- Уникальный молекулярный идентификатор (UMI) для исправления смещения усиления
- Пространственный штрих -код
- Последовательность связывания частичного праймера секвенирования считывания 1
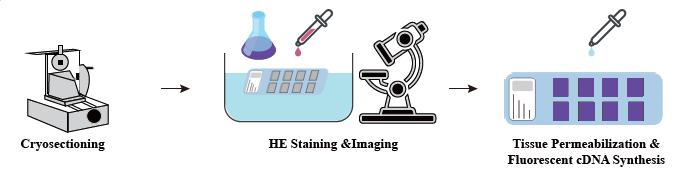
● H & E окрашивание разделами
Преимущества
●ОДИНСКАЯ СЛУЖБА: интегрирует все этапы опыта и навыков, включая крио-сечение, окрашивание, оптимизацию тканей, пространственное штрих-кодирование, подготовку библиотеки, секвенирование и биоинформатику.
● Высокопоставленная техническая команда: с опытом более 250 типов тканей и 100+ видов, включая человека, мыши, млекопитающие, рыбу и растения.
●Обновление в реальном времени на весь проект: с полным контролем экспериментального прогресса.
●Комплексная стандартная биоинформатика:Пакет включает 29 анализов и 100+ высококачественных рисунков.
●Индивидуальный анализ данных и визуализация: Доступно для различных запросов на исследования.
●Дополнительный совместный анализ с одноклеточным секвенированием мРНК
Спецификации
| Требования к выборке | Библиотека | Стратегия секвенирования | Данные рекомендуются | Контроль качества |
| Октубленные образцы крио (Оптимальный диаметр: ок. 6x6x6 мм³) 2 блока на выборку | 10x библиотека кДНК Visium | Illumina PE150 | 50K PE читает за место (60 ГБ) | Рин> 7 |
Для получения более подробной информации о руководстве по подготовке к примеру и рабочим процессам обслуживания, пожалуйста, не стесняйтесь поговорить сЭксперт BMKGENE
Служба рабочего процесса
На фазе препарата образца проводится начальное исследование извлечения объемной РНК для обеспечения получения высококачественной РНК. На стадии оптимизации ткани срезы окрашиваются и визуализируются, а условия проницаемости для высвобождения мРНК из ткани оптимизируются. Оптимизированный протокол затем применяется во время конструкции библиотеки, за которым следует секвенирование и анализ данных.
Полный рабочий процесс обслуживания включает в себя обновления в реальном времени и подтверждения клиентов для поддержания отзывчивого цикла обратной связи, обеспечивая плавное выполнение проекта.

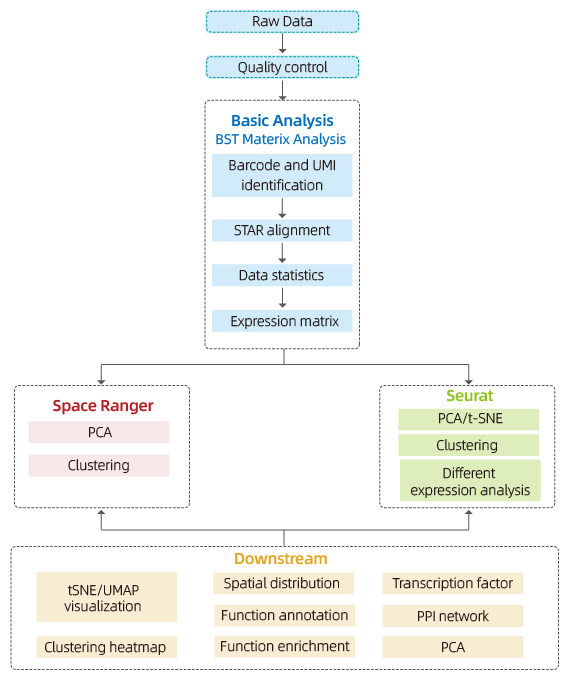
Включает следующий анализ:
Контроль качества данных:
o Вывод данных и распределение баллов качества
o Обнаружение генов за место
o Покрытие тканей
Анализ внутренней выборки:
o Гиновое богатство
o Spot Clustering, включая анализ пониженного размера
o Анализ дифференциальной экспрессии между кластерами: идентификация маркерных генов
o Функциональная аннотация и обогащение маркерных генов
Межгрупповой анализ
o Повторная комбинация пятен из обоих образцов (например, боль.
o Идентификация маркерных генов для каждого кластера
o Функциональная аннотация и обогащение маркерных генов
o Дифференциальная экспрессия одного и того же кластера между группами
Анализ внутренней выборки
Точечная кластеризация

Маркерные гены идентификация и пространственное распределение


Межгрупповой анализ
Комбинация данных как из групп, так и повторного кластера
Маркерные гены новых кластеров
Исследуйте достижения, облегченные Службой пространственной транскриптомики BMKGENE с 10 -кратным VISIUM в этих представленных публикациях:
Chen, D. et al. (2023) 'MTHL1, потенциальный гомолог Drosophila gpcrs адгезии млекопитающих, участвует в противоопухолевых реакциях на инъецированные онкогенные клетки у мух, »,Труды Национальной академии наук Соединенных Штатов Америки, 120 (30), с. E2303462120. doi: /10.1073/pnas.2303462120
Chen, Y. et al. (2023) «Сталь обеспечивает разграничение пространственно-временных транскриптомных данных»,Брифинги в биоинформатике, 24 (2), с. 1–10. doi: 10.1093/bib/bbad068.
Лю, С. и соавт. (2022) «пространственно -временный атлас органогенеза в развитии цветов орхидей»,Исследование нуклеиновых кислот, 50 (17), с. 9724–9737. doi: 10.1093/nar/gkac773.
Wang, J. et al. (2023) «Интеграция пространственной транскриптомики и секвенирования РНК с одним нуклеусом выявляет потенциальные терапевтические стратегии для лейомиомы матки»,Международный журнал биологических наук, 19 (8), с. 2515–2530. doi: 10.7150/ijbs.83510.