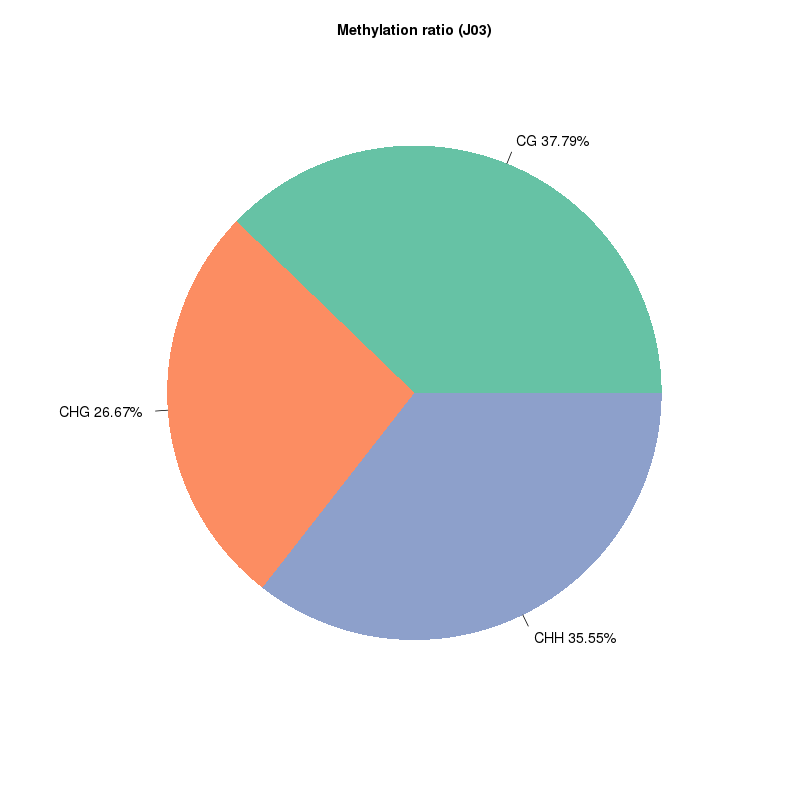Sequenciamento de bissulfito do genoma completo (WGBS)
Recursos de serviço
● Requer um genoma de referência.
● Lambda DNA é adicionado para monitorar a eficiência de conversão de bissulfito.
● Sequenciamento no Illumina NovaSeq.
Vantagens do serviço
●Padrão Ouro para Pesquisa de Metilação de DNA: Esta tecnologia madura de processamento de conversão de metilação tem alta precisão e boa reprodutibilidade.
●Ampla cobertura e resolução de base única:detecção de locais de metilação em todo o genoma.
●Plataforma Completa:fornecer excelente serviço completo, desde processamento de amostras, construção de bibliotecas, sequenciamento até análise de bioinformática.
●Ampla experiência: com projetos de sequenciamento WGBS concluídos com sucesso em uma ampla gama de espécies, a BMKGENE traz mais de uma década de experiência, uma equipe de análise altamente qualificada, conteúdo abrangente e excelente suporte pós-venda.
●Possibilidade de união com análise transcriptômica: permitindo a análise integrada de WGBS com outros dados ômicos, como RNA-seq.
Especificações de amostra
| Biblioteca | Estratégia de sequenciamento | Saída de dados recomendada | Controle de qualidade |
| Tratado com bissulfito | Illumina PE150 | Profundidade 30x | Q30 ≥ 85% Conversão de bissulfito > 99% |
Requisitos de amostra
| Concentração (ng/µL) | Quantidade total (µg) | Requisitos adicionais | |
| DNA genômico | ≥ 5 | ≥ 400ng | Degradação ou contaminação limitada |
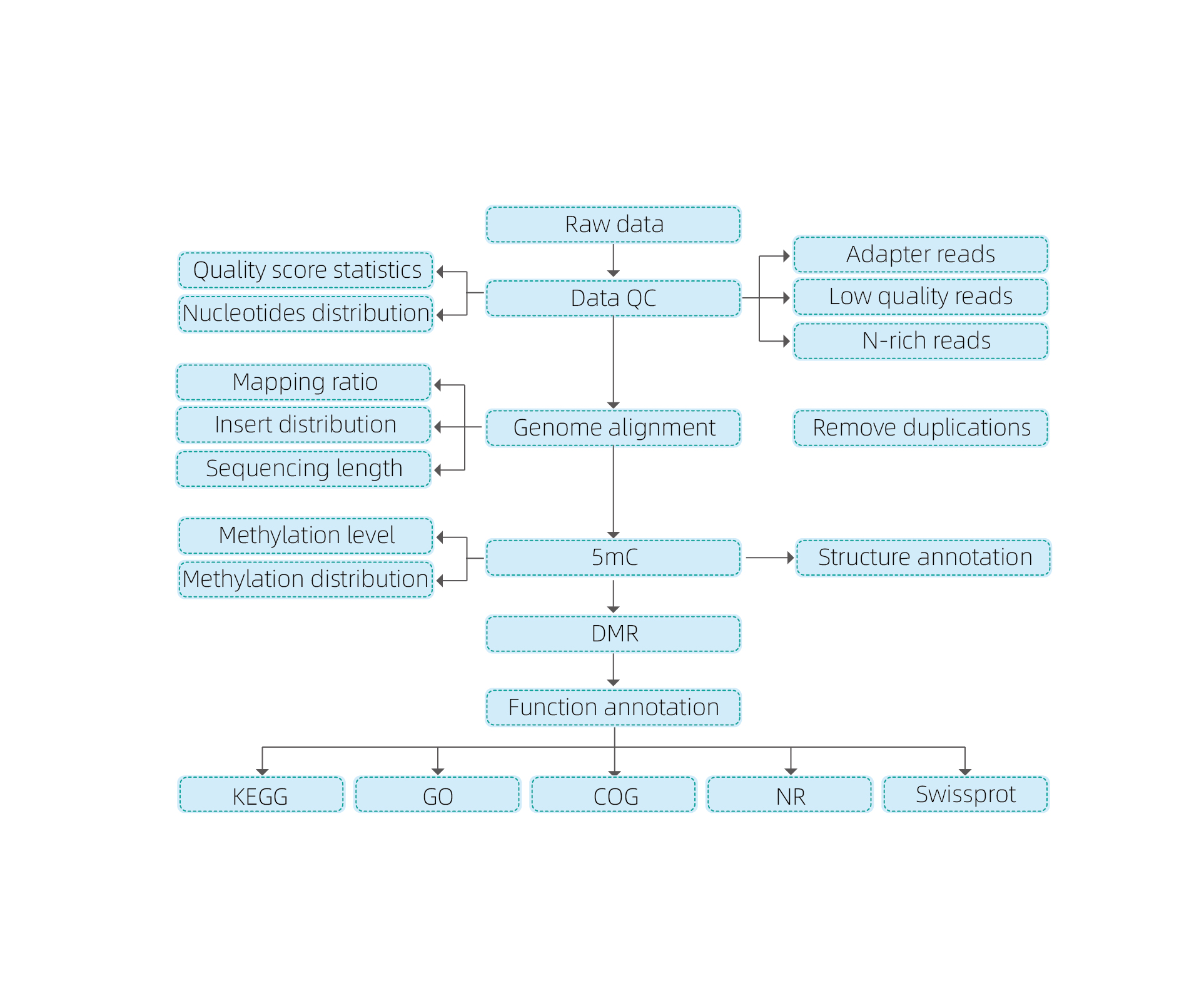
Fluxo de trabalho de serviço

Entrega de amostra

Extração de DNA

Construção de biblioteca

Sequenciamento

Análise de dados

Entrega de dados
Inclui a seguinte análise:
● Controle de qualidade do sequenciamento bruto;
● Mapeamento para genoma de referência;
● Detecção de bases metiladas 5mC;
● Análise de distribuição e anotação de metilação;
● Análise de Regiões Diferencialmente Metiladas (DMRs);
● Anotação funcional de genes associados a DMRs.
Detecção de metilação 5mC: tipos de locais metilados
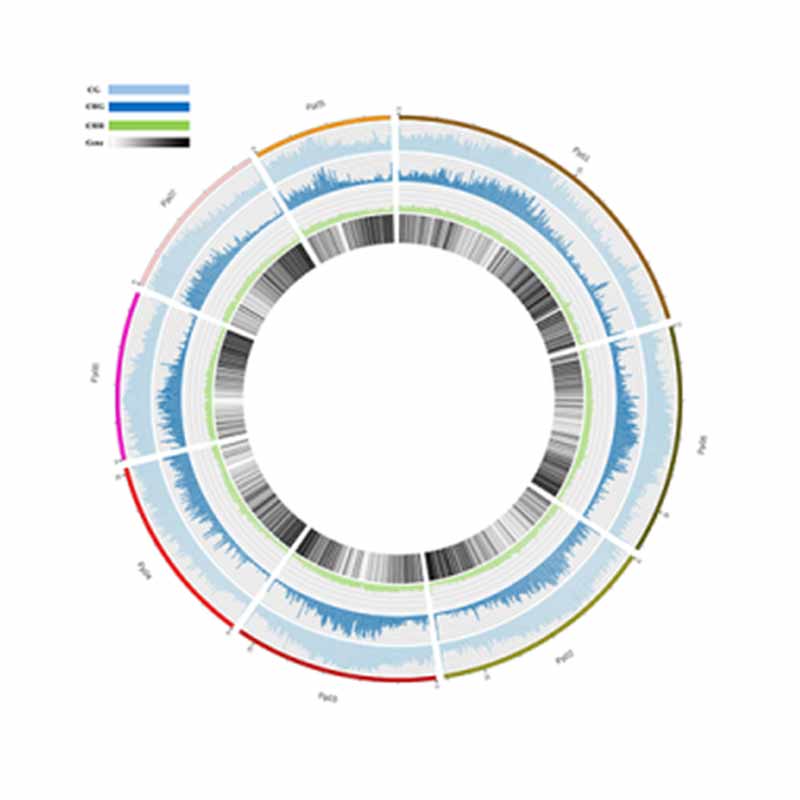
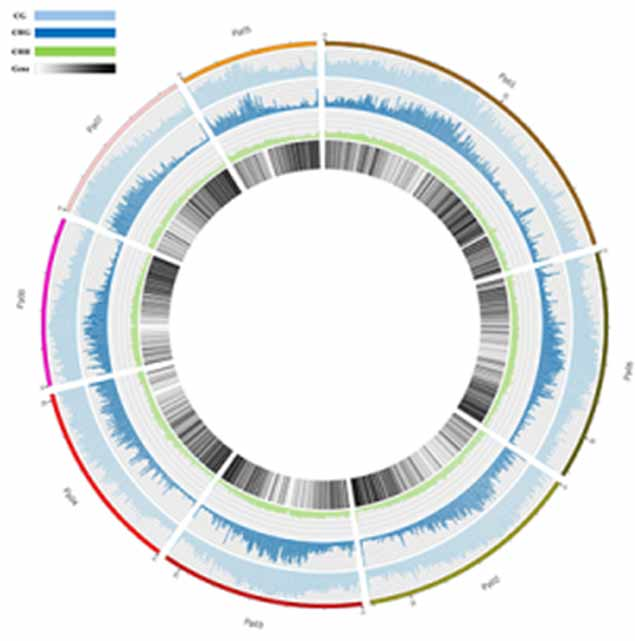
Mapa de metilação. Distribuição genômica de metilação de 5mC
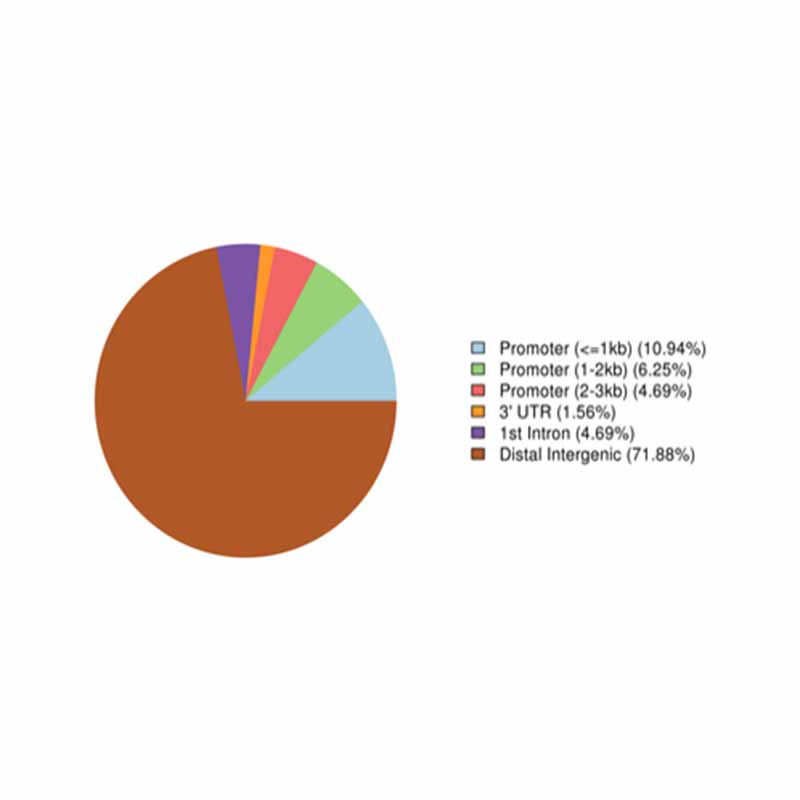
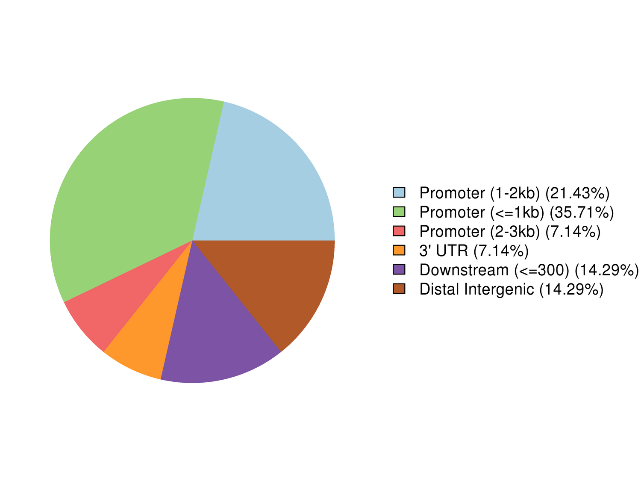
Anotação de regiões altamente metiladas
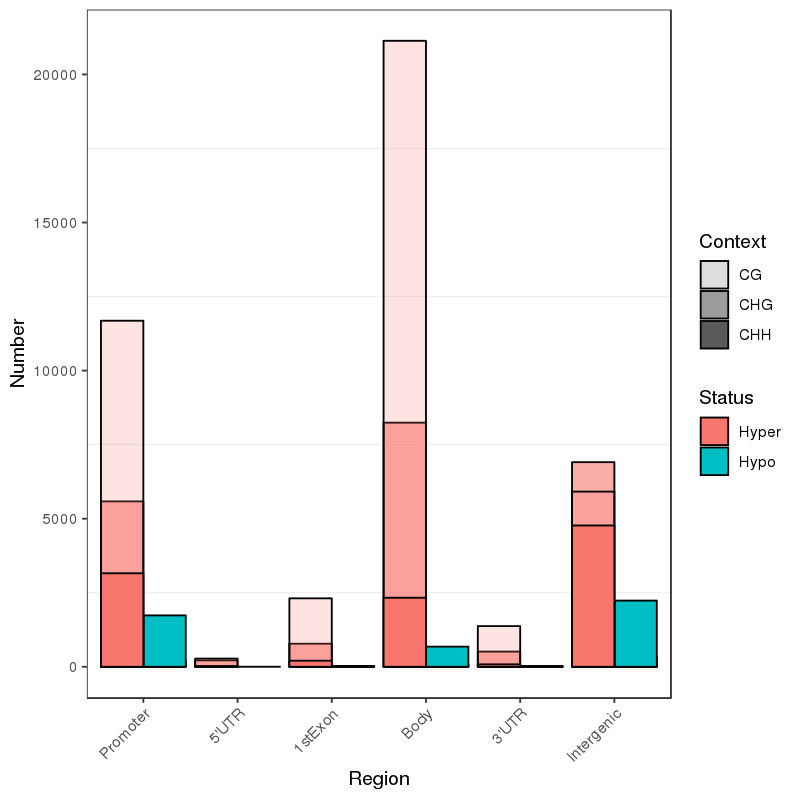
Regiões diferencialmente metiladas: genes associados
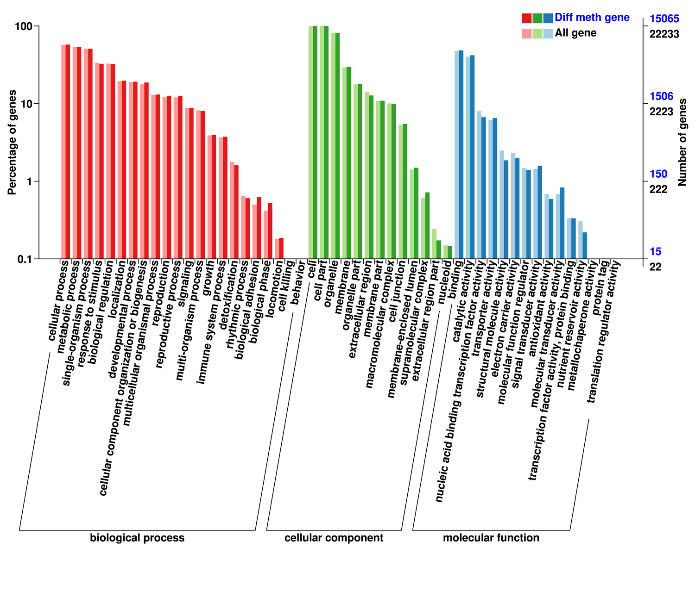
Regiões diferencialmente metiladas: anotação de genes associados (Gene Ontology)
Explore os avanços da pesquisa facilitados pelos serviços de sequenciamento de bissulfito do genoma completo da BMKGene por meio de uma coleção selecionada de publicações.
Fan, Y. et al. (2020) 'Análise de perfis de metilação do DNA durante o desenvolvimento do músculo esquelético de ovelhas usando sequenciamento de bissulfito do genoma completo',Genômica BMC, 21(1), pp. doi: 10.1186/S12864-020-6751-5.
Zhao, X. et al. (2022) 'Novas perturbações da metilação do ácido desoxirribonucléico em trabalhadores expostos ao cloreto de vinil',Toxicologia e Saúde Industrial, 38(7), pp. doi: 10.1177/07482337221098600
Zuo, J. et al. (2020) 'Relações entre metilação do genoma, níveis de RNAs não codificantes, mRNAs e metabólitos no amadurecimento do tomate',O Diário da Planta, 103(3), pp. doi: 10.1111/TPJ.14778.