Sequenciamento de RNA de núcleos únicos
Esquema técnico
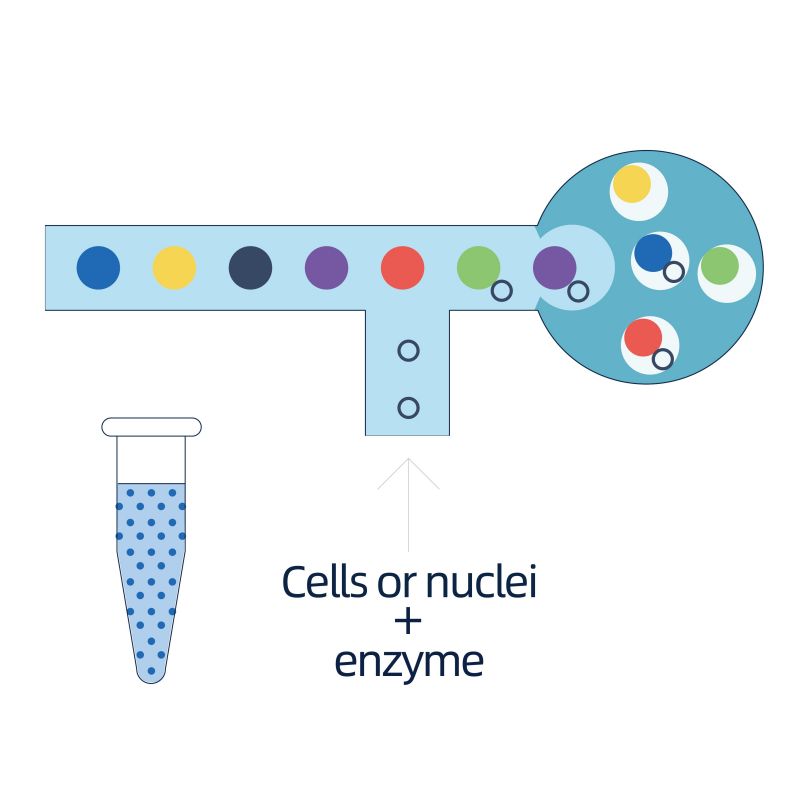
O isolamento dos núcleos é alcançado por 10 × Genomics Chromium ™, que consiste no sistema de microfluídicos de oito canais com cruzamentos duplos. Nesse sistema, um gel de gel com códigos de barras e primer, enzimas e um único núcleo são encapsulados na queda de óleo do tamanho de nanoliter, gerando contas de gel em emulsão (gem). Uma vez formado a gem, a lise celular e a liberação de códigos de barras são realizados em cada gem. O mRNA é transcrito reverso em moléculas de cDNA com códigos de barras 10 × e UMI, que estão mais sujeitos à construção de bibliotecas de sequenciamento padrão.

Características
● Preparação da suspensão de nuclei de tecidos congelados
● Formação de Gel Bead-Emulsão (GEM) seguida de síntese de cDNA
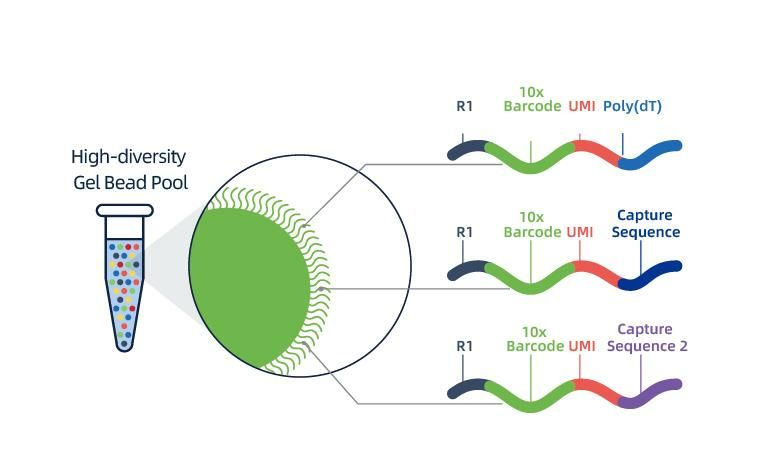
● Cada cordão em uma jóia é carregado com iniciadores compostos por 4 seções:
Tail de poli (dt) para priming de mRNA e síntese de cDNA,
Identificador molecular exclusivo (UMI) para corrigir o viés de amplificação
10x código de barras
Sequência de ligação de leitura parcial 1 Primer de seqüenciamento
Vantagens
O sequenciamento de RNA único-nucleus circunha as limitações do sequenciamento de RNA de célula única, permitindo:
● O uso de amostras congeladas e não apenas limitada a amostras novas
● Baixo estresse das células congeladas quando comparado ao tratamento enzimático de células frescas, refletidas nos dados do transcriptoma na forma de menos genes induzidos por estresse
● Não há necessidade de remoção prévia de glóbulos vermelhos
● diâmetro celular ilimitado
● Grande variedade de amostras que são elegíveis para análise, incluindo tipos de tecido complexos e frágeis que são propensos a aglomerados ou destruição de células durante a dissociação do tecido
Amostras que não podem ser analisadas pelo seqüenciamento de RNA de célula única e são elegíveis para o seqüenciamento de RNA de núcleos únicos:
| Célula / tecido | Razão |
| Tecido congelado sem preenchimento | Incapaz de obter organizações novas ou de longo prazo |
| Célula muscular, megakariócitos, gordura… | O diâmetro da célula é muito grande para entrar no instrumento |
| Fígado… | Muito frágil para quebrar, incapaz de distinguir células únicas |
| Célula de neurônios, cérebro… | Mais sensível, fácil de estressar, mudará os resultados de seqüenciamento |
| Pâncreas, tireóide… | Rico em enzimas endógenas, afetando a produção de suspensão de célula única |
Nucleo único vs célula única
| Núcleo único | Célula única |
| Diâmetro celular ilimitado | Diâmetro celular: 10-40 μm |
| O material pode ser congelado | O material deve ser tecido fresco |
| Baixo estresse de células congeladas | O tratamento enzimático pode causar reação de estresse celular |
| Nenhum glóbulo vermelho precisa ser removido | Os glóbulos vermelhos precisam ser removidos |
| A nuclear expressa bioinformação | Toda a célula expressa bioinformação |
Especificações
| Requisitos de amostra | Biblioteca | Estratégia de sequenciamento | Dados recomendados | Controle de qualidade |
| Tecido animal ≥ 200 mg Tecido vegetal ≥ 400 mg | Biblioteca de cDNA da genômica 10x | Illumina PE150 | 100k PE lê por célula (100-200 GB) | 700-1200 núcleos/μl e integridade de núcleos observados sob microscópio |
Para mais detalhes sobre a orientação de preparação de amostras e fluxo de trabalho de serviço, sinta -se à vontade para conversar com umEspecialista em BMKGENE
Fluxo de trabalho de serviço

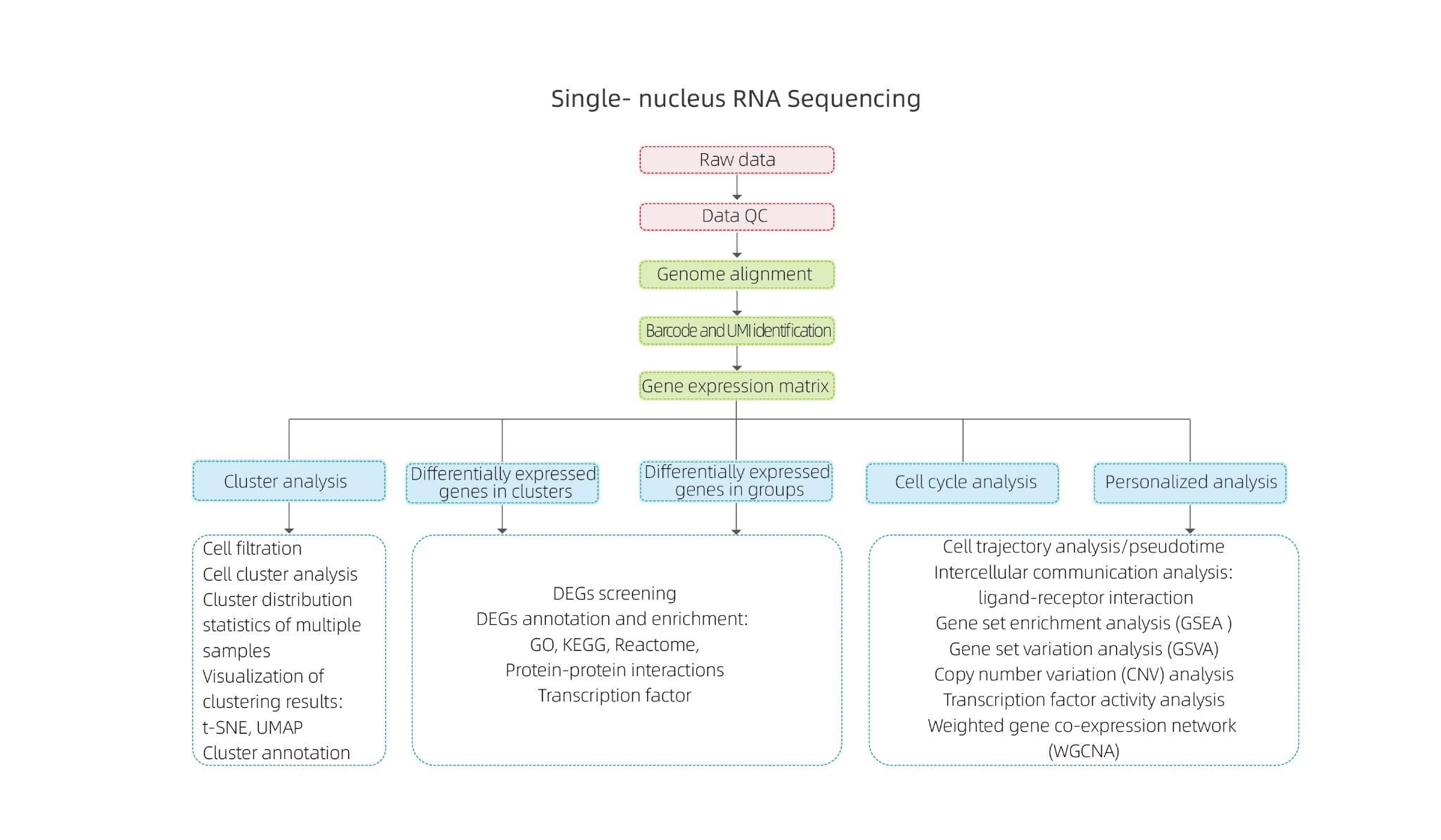
Inclui a seguinte análise:
● Controle de qualidade: número de células, detecção de genes, identificação precisa das células, moléculas de RNA e quantificação de expressão
● Análise de amostra interna:
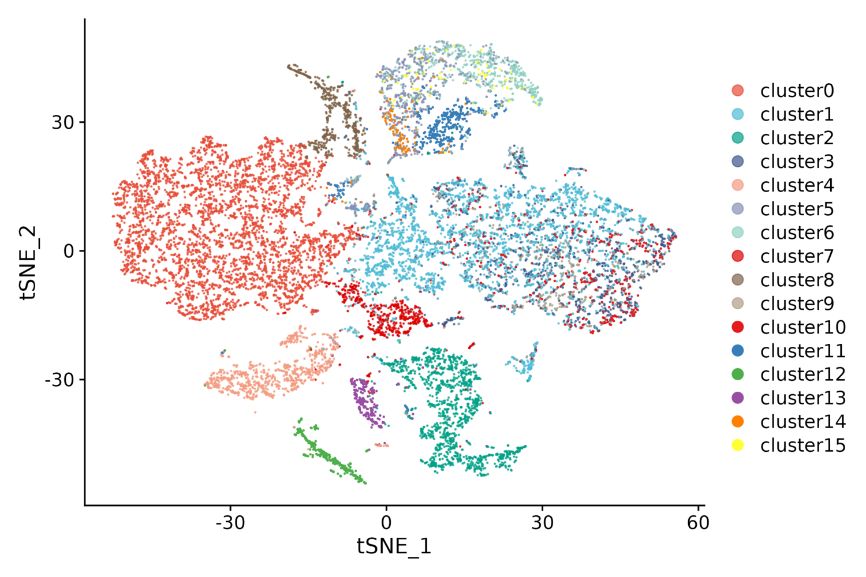
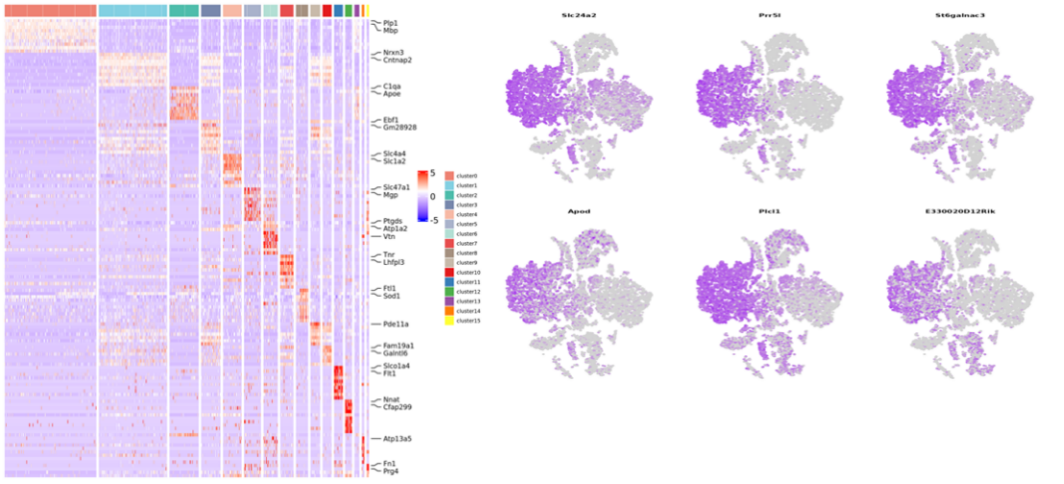
Agrupamento de células e anotação de cluster
Análise de expressão diferencial: identificação de degs em clusters
Anotação funcional e enriquecimento de degs de cluster
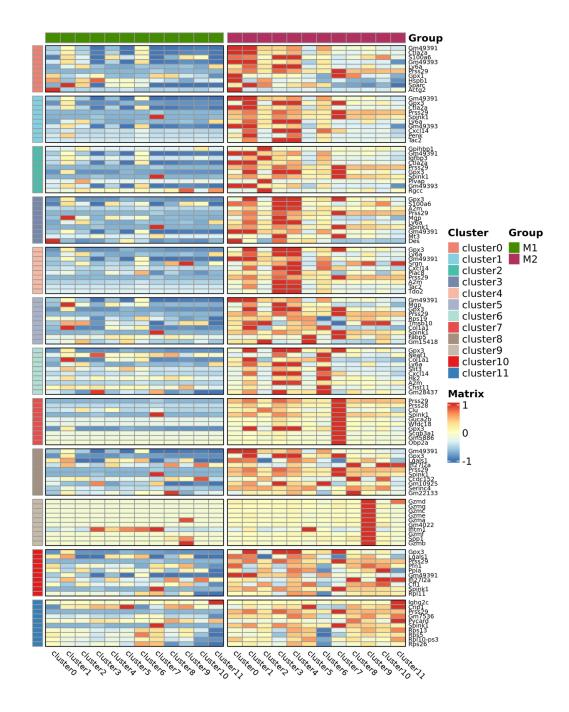
● Análise entre grupos:
Combinação de dados
Análise de expressão diferencial: identificação de DEGs em grupos
Anotação funcional e enriquecimento de Grupo Degs
● Análise avançada:
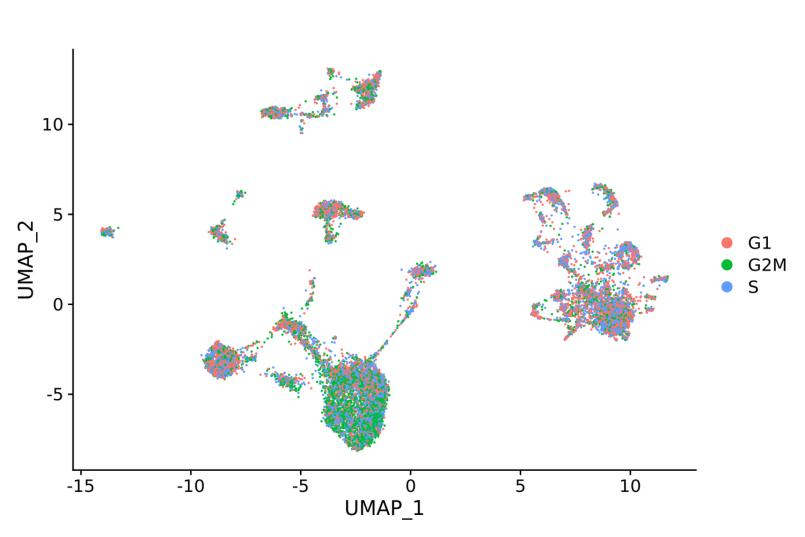
Análise do ciclo celular
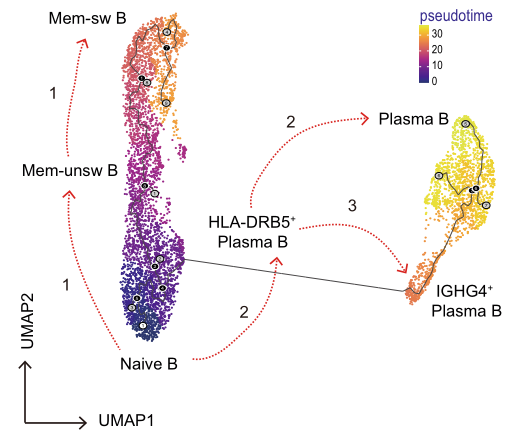
Análise de pseudotime
Análise de comunicação celular (CellphonedB)
Análise de enriquecimento do conjunto de genes (GSEA)
Análise de amostra interna
Cluster de células:
Análise de expressão diferencial: degs de cluster
Análise entre grupos
Análise de expressão diferencial: Grupo Degs
Análise Avançada:
Análise de pseudotime:
Análise do ciclo celular:
Explore os avanços facilitados pelos Serviços de Sequenciamento de RNA de Nucleus único da BMKGENE por 10x Chromium nessas publicações em destaque:
Wang, L. et al. (2021) 'Análise transcriptômica de célula única revela o cenário imunológico do pulmão na exacerbação da asma resistente a esteróides',Anais da Academia Nacional de Ciências dos Estados Unidos da América, 118 (2), p. E2005590118. doi: 10.1073/pnas.2005590118
Zheng, H. et al. (2022) 'Uma rede regulatória global para expressão gênica desregulada e sinalização metabólica anormal em células imunes no microambiente da doença de Graves e na tireoidite de Hashimoto',Fronteiras em imunologia, 13, p. 879824. DOI: 10.3389/FIMMU.2022.879824/BIBTEX.
Tian, H. et al. (2023) 'O transcriptoma de célula única descobre a heterogeneidade e as respostas imunes dos leucócitos após a vacinação com Edwardsiella Tarda inativada em linguado (Paralichthys olivaceus)',Aquicultura, 566, p. 739238. Doi: 10.1016/j.aquaculture.2023.739238.
Yu, Y. et al. (2023) 'A terapia fotodinâmica melhora o resultado de inibidores do ponto de verificação imune por meio da remodelação da imunidade antitumoral em pacientes com câncer gástrico',Câncer gástrico, 26 (5), pp. 798–813. doi: 10.1007/s10120-023-01409-x/métricas.