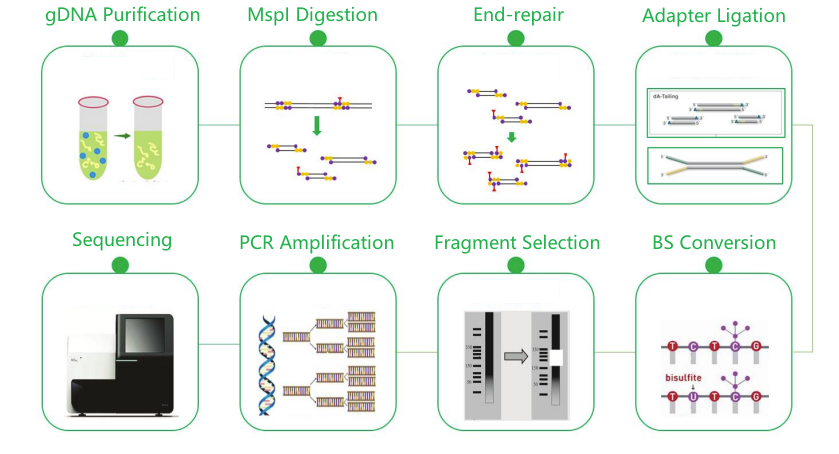
Sequenciamento de Bissulfito de Representação Reduzida (RRBS)
Recursos de serviço
● Requer um genoma de referência.
● Lambda DNA é usado para monitorar a eficiência de conversão de bissulfito.
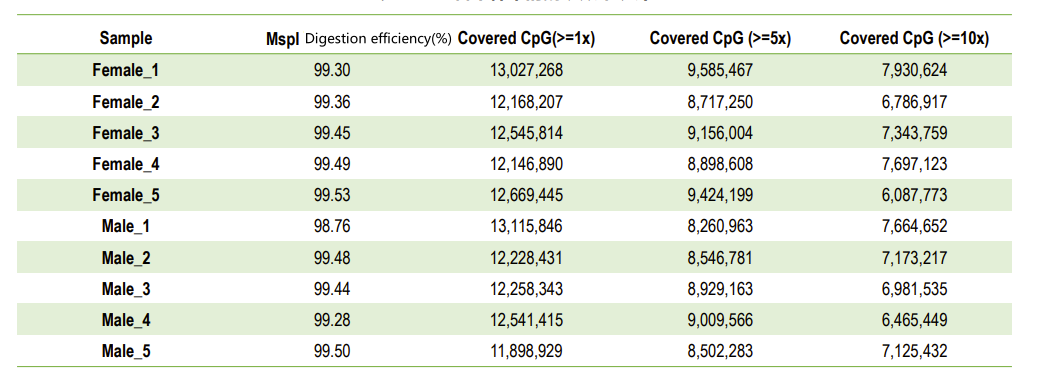
● A eficiência da digestão MspI também é monitorada.
● Digestão enzimática dupla para amostras de plantas.
● Sequenciamento no Illumina NovaSeq.
Vantagens do serviço
●Alternativa econômica e eficiente ao WGBS: possibilitando que a análise seja realizada com menor custo e com menor necessidade de amostra.
●Plataforma Completa:fornecer excelente serviço completo, desde processamento de amostras, construção de biblioteca e sequenciamento até análise de bioinformática.
●Ampla experiência: com projetos de sequenciamento de RRBS concluídos com sucesso em uma ampla gama de espécies, a BMKGENE traz mais de uma década de experiência, uma equipe de análise altamente qualificada, conteúdo abrangente e excelente suporte pós-venda.
Especificações de serviço
| Biblioteca | Estratégia de sequenciamento | Saída de dados recomendada | Controle de qualidade |
| Biblioteca digerida com MspI e tratada com bissulfito | Illumina PE150 | 8 GB | Q30 ≥ 85% Conversão de bissulfito > 99% Eficiência de corte MspI > 95% |
Requisitos de amostra
| Concentração (ng/µL) | Quantidade total (µg) |
| |
| DNA genômico | ≥ 30 | ≥ 1 | Degradação ou contaminação limitada |
Fluxo de trabalho de serviço

Entrega de amostra

Construção de biblioteca

Sequenciamento

Análise de dados
Serviços pós-venda
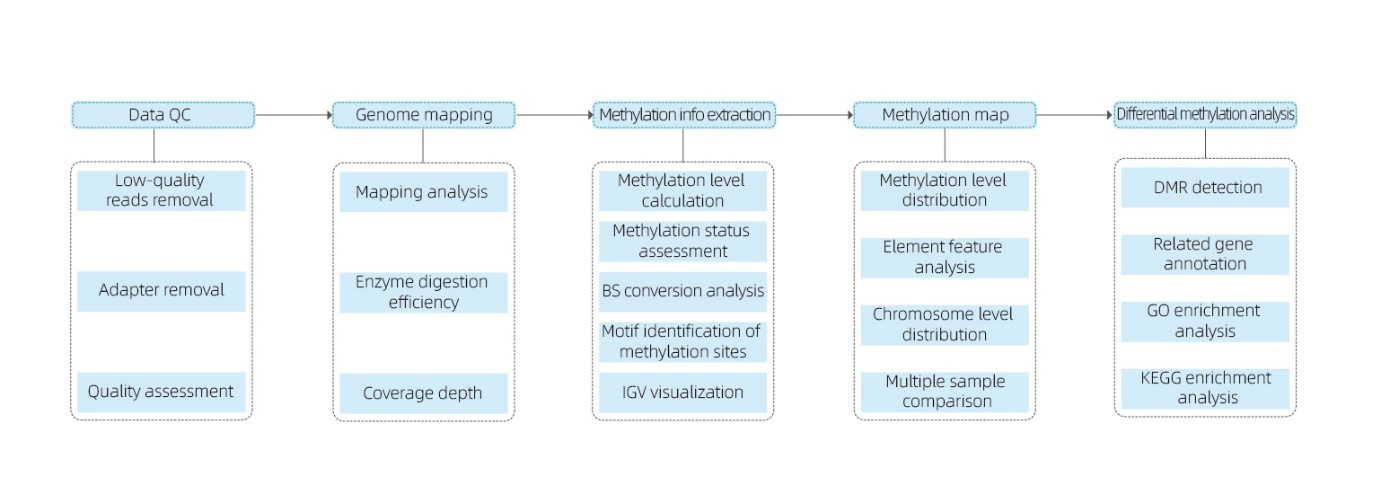
Inclui a seguinte análise:
● Controle de qualidade do sequenciamento bruto;
● Mapeamento para genoma de referência;
● Detecção de bases metiladas 5mC e identificação de motivos;
● Análise de distribuição de metilação e comparação de amostras;
● Análise de Regiões Diferencialmente Metiladas (DMRs);
● Anotação funcional de genes associados a DMRs.
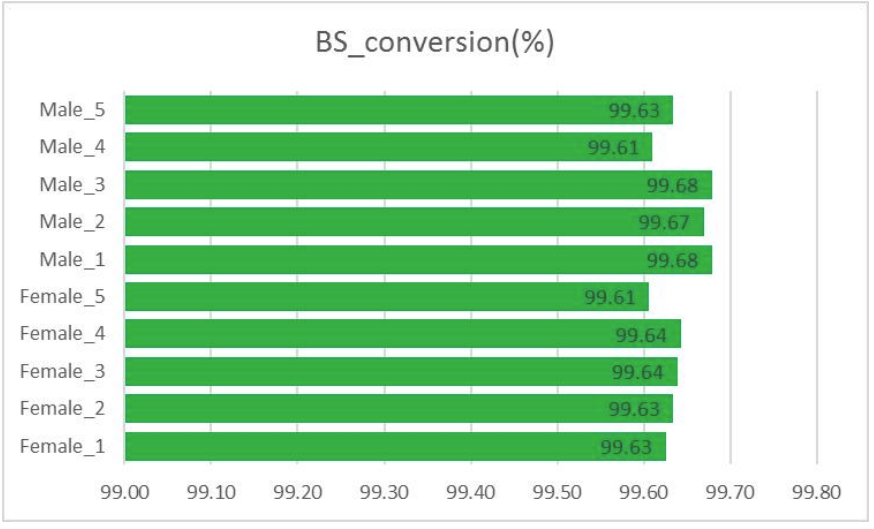
Controle de qualidade: eficiência da digestão (no mapeamento do genoma)
Controle de qualidade: conversão de bissulfito (na extração de informações de metilação)
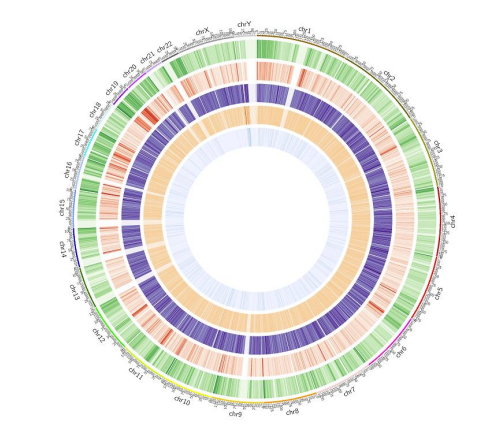
Mapa de metilação: distribuição genômica de metilação de 5mC
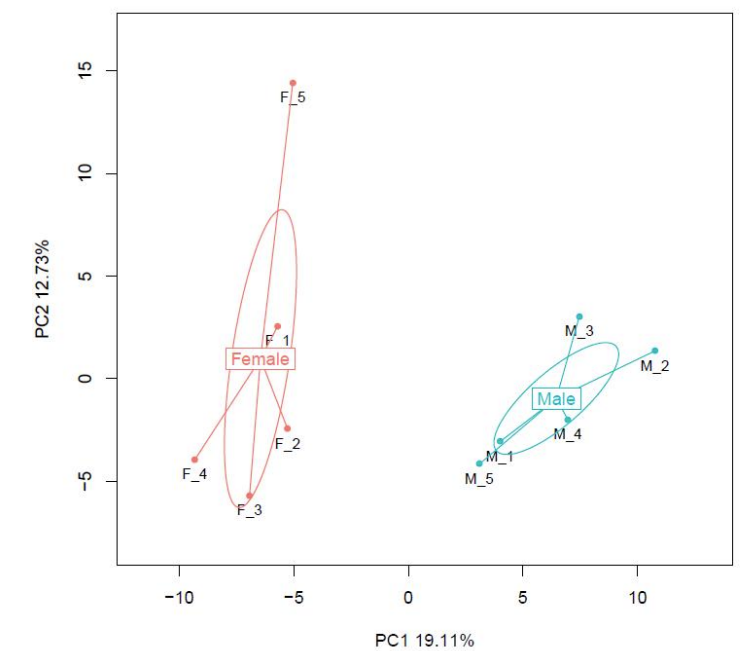
Comparação de amostras: Análise de Componentes Principais
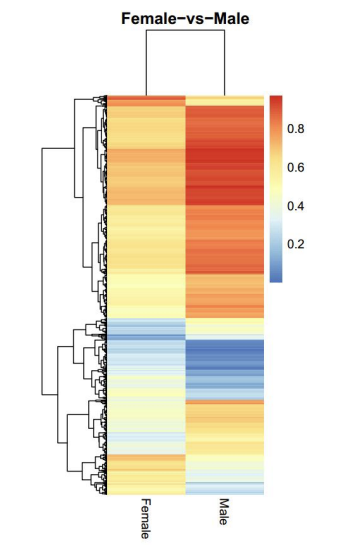
Análise de regiões diferencialmente metiladas (DMRs): mapa de calor
Explore os avanços da pesquisa facilitados pelos serviços de sequenciamento de bissulfito do genoma completo da BMKGene por meio de uma coleção selecionada de publicações.
Li, Z. et al. (2022) 'Reprogramação de alta fidelidade em células semelhantes a Leydig por ativação CRISPR e fatores parácrinos',Nexus do PNAS, 1(4). doi: 10.1093/PNASNEXUS/PGAC179.
Tian, H. et al. (2023) 'Análise de metilação do DNA em todo o genoma da composição corporal em gêmeos monozigóticos chineses',Jornal Europeu de Investigação Clínica, 53(11), pág. e14055. doi: 10.1111/ECI.14055.
Wu, Y. et al. (2022) 'Metilação do DNA e relação cintura-quadril: um estudo de associação de todo o epigenoma em gêmeos monozigóticos chineses',Revista de Investigação Endocrinológica, 45(12), pp. doi: 10.1007/S40618-022-01878-4.