Sequenciamento de RNA procariótico
Características
● O processamento da amostra de RNA envolveu a depleção de rRNA seguida pela preparação direcional da biblioteca de RNA.
● Análise bioinformática com base no alinhamento a um genoma de referência
● Análise inclui expressão gênica e DEGs, mas também a estrutura da transcrição e a análise de sRNA
Vantagens de serviço
●Controle de qualidade rigoroso: Implementamos pontos de controle principal em todos os estágios, desde a preparação da amostra e da biblioteca até o sequenciamento e a bioinformática. Esse monitoramento meticuloso garante a entrega de resultados consistentemente de alta qualidade.
●Dados de sequenciamento específicos da fita: devido à preparação da biblioteca de RNA ser direcional, permitindo a identificação de transcritos anti-senso.
●Análise completa adaptada a transcriptomos procarióticos: O pipeline bioinformático inclui não apenas a análise da expressão gênica, mas também a análise da estrutura da transcrição, incluindo a identificação de operons, UTRs e promotores. Ele também inclui a análise dos SRNAs, a saber, anotação e previsão de estrutura e alvos secundários.
●Suporte pós-venda: Nosso compromisso se estende além da conclusão do projeto com um período de serviço pós-venda de três meses. Durante esse período, oferecemos acompanhamento do projeto, assistência de solução de problemas e sessões de perguntas e respostas para abordar quaisquer consultas relacionadas aos resultados.
Requisitos de amostra e entrega
| Biblioteca | Estratégia de sequenciamento | Dados recomendados | Controle de qualidade |
| biblioteca direcional esgotada do rRNA | Illumina PE150 | 1-2 GB | Q30≥85% |
Requisitos de amostra:
| Conc. (Ng/μl) | Quantidade (μg) | Pureza | Integridade |
| ≥ 50 | ≥ 1 | OD260/280 = 1,8-2.0 OD260/230 = 1,0-2.5 Contaminação limitada ou sem proteína ou DNA mostrado em gel. | Rin≥6,5 |
Entrega de amostra recomendada
Contêiner: 2 ml de tubo de centrífuga (folha de estanho não é recomendada)
Rotulagem de amostra: grupo+replicar por exemplo A1, A2, A3; B1, B2, B3.
Remessa:
1. Gelo seco: as amostras precisam ser embaladas em sacos e enterradas em gelo seco.
2. Tubos RNASTABLE: As amostras de RNA podem ser secas no tubo de estabilização de RNA (por exemplo, rnastable®) e enviadas em temperatura ambiente.
Fluxo de trabalho de serviço

Entrega de amostra

Construção da biblioteca

Sequenciamento

Análise de dados

Serviços pós-venda
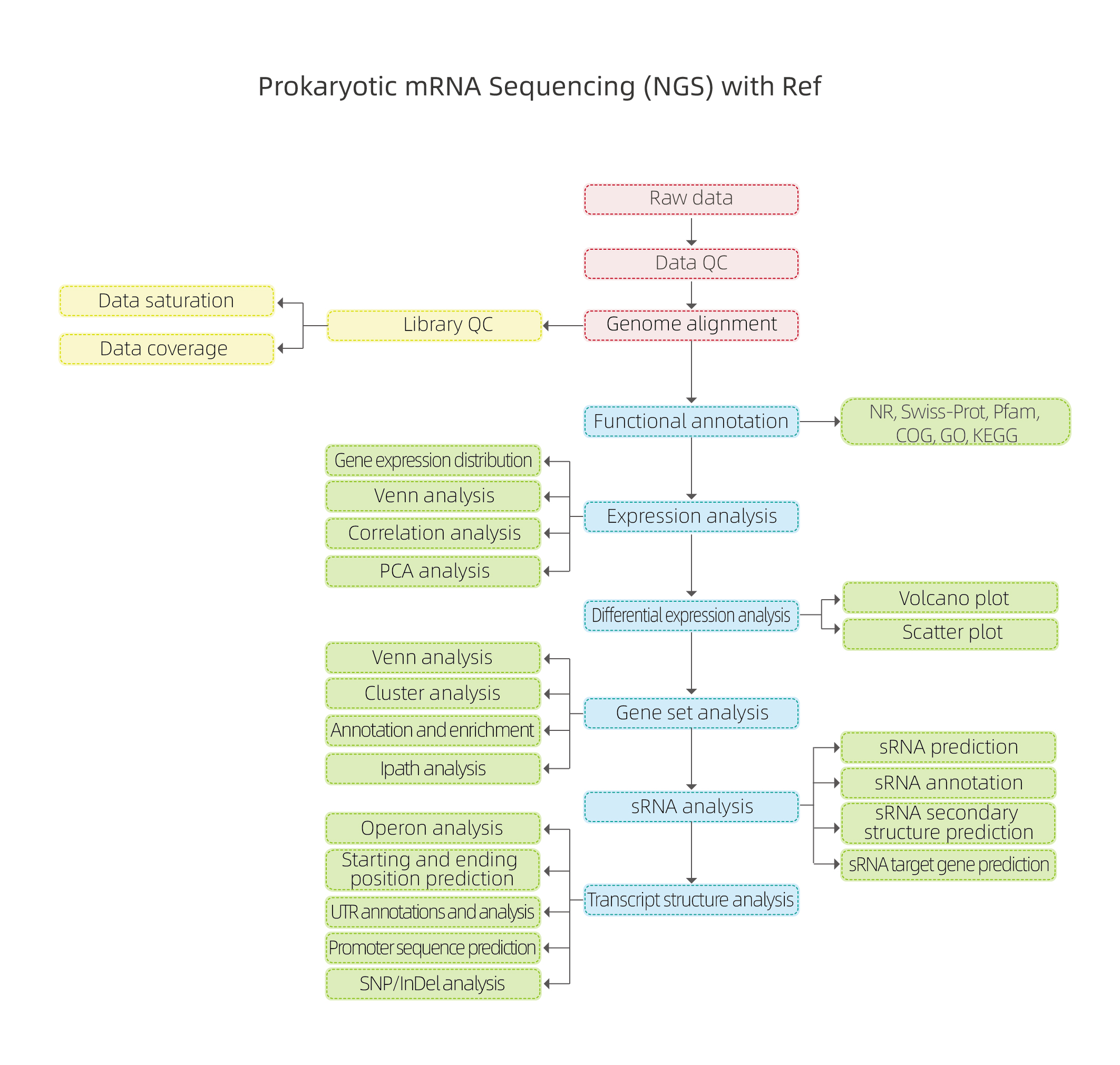
Fluxo de trabalho de análise bioinformática
Inclui a seguinte análise:
● Controle de qualidade de dados brutos
● Alinhamento ao genoma de referência
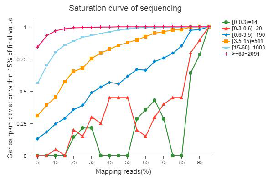
● Avaliação da qualidade da biblioteca: fragmentação de RNA, aleatoriedade, tamanho de inserção e saturação de seqüenciamento
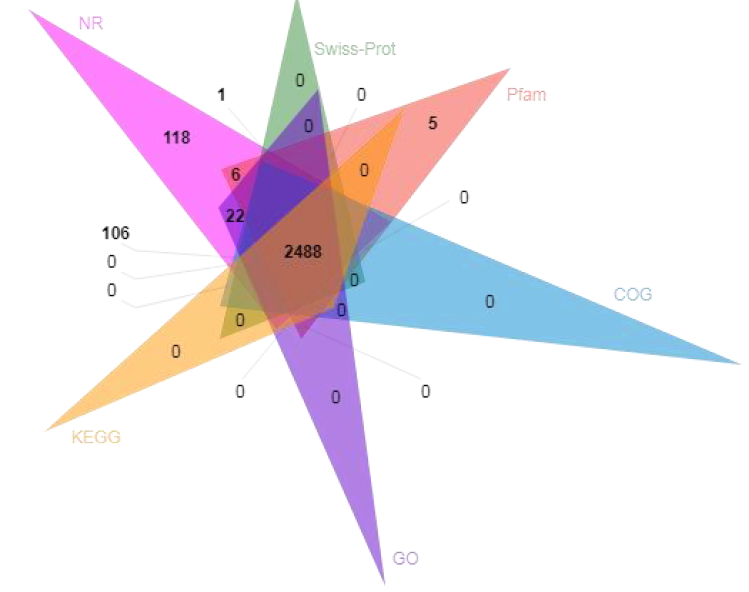
● Anotação funcional de genes de codificação previstos
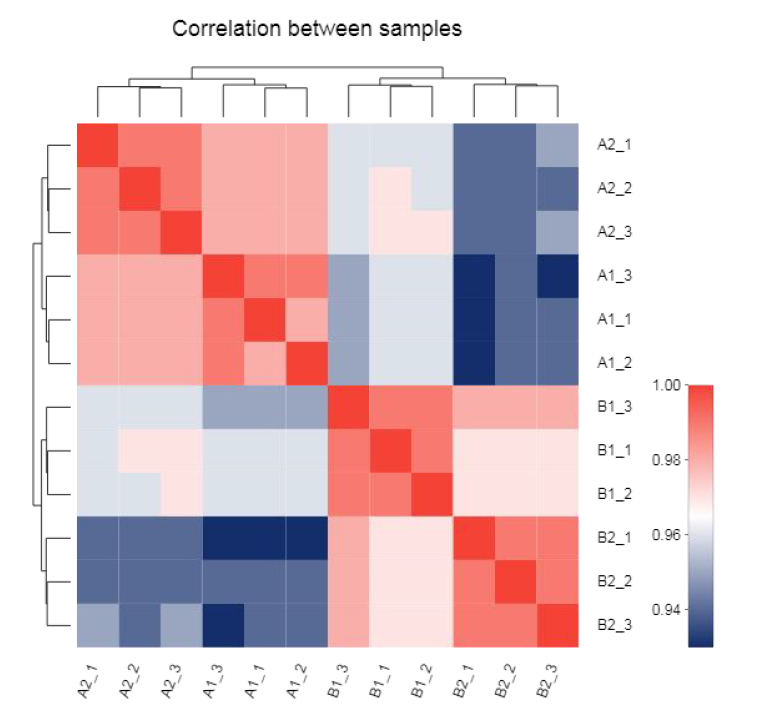
● Análise de expressão: correlação e análise de componentes principais (PCA)
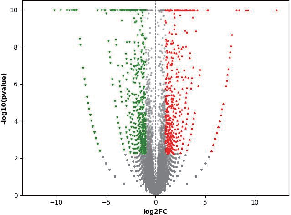
● Expressão do gene diferencial (DEGs)
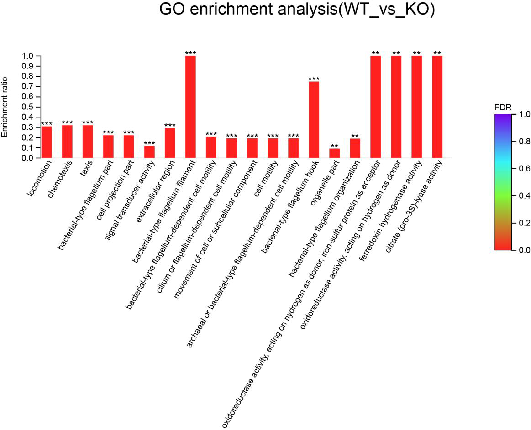
● Anotação funcional e enriquecimento de DEGs
● Análise de sRNA: previsão, anotação, alvo e previsão de estrutura secundária
● Análise da estrutura da transcrição: operons, posições iniciais e finais, região não traduzida (UTS), promotor e análise SNP/INDEL
Saturação de sequenciamento
Anotação funcional de genes de codificação
Correlação entre amostras
Análise de genes expressos diferenciais (DEGs)
Análise de enriquecimento funcional
anotação srna
Explore os avanços facilitados pelos serviços de sequenciamento de mRNA de Nanopore de Bmkgene nesta publicação em destaque.
Guan, Cp et al. (2018) 'Alterações globais de transcriptoma de Staphylococcus epidermidis formador de biofilme respondendo a alcalóides totais de Alopecuroides Sophorea',Jornal polonês de microbiologia, 67 (2), p. 223. DOI: 10.21307/PJM-2018-024.