-

Sequenciamento Metagenômico -NGS
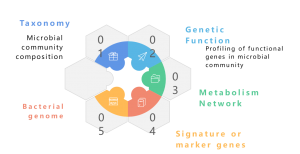
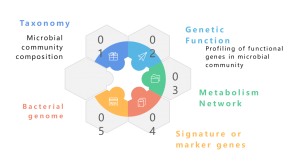
Um metagenoma é uma coleção do material genético total de uma comunidade mista de organismos, como metagenomas ambientais e humanos. Ele contém genomas de microrganismos cultiváveis e não cultiváveis. O sequenciamento metagenômico shotgun com NGS permite o estudo dessas intrincadas paisagens genômicas incorporadas em amostras ambientais, fornecendo mais do que perfis taxonômicos, fornecendo também insights granulares sobre a diversidade de espécies, dinâmica de abundância e estruturas populacionais complexas. Além dos estudos taxonômicos, a metagenômica shotgun também oferece uma perspectiva genômica funcional, permitindo a exploração de genes codificados e seus supostos papéis em processos ecológicos. Finalmente, o estabelecimento de redes de correlação entre elementos genéticos e factores ambientais contribui para uma compreensão holística da intricada interacção entre as comunidades microbianas e o seu contexto ecológico. Em conclusão, o sequenciamento metagenómico constitui um instrumento fundamental para desvendar os meandros genómicos de diversas comunidades microbianas, iluminando as relações multifacetadas entre genética e ecologia dentro destes ecossistemas complexos.
Plataformas: Illumina NovaSeq e DNBSEQ-T7
-

Sequenciamento Metagenômico-TGS
Um metagenoma é uma coleção de material genético de uma comunidade mista de organismos, como metagenomas ambientais e humanos. Ele contém genomas de microrganismos cultiváveis e não cultiváveis. O sequenciamento metagenômico permite o estudo dessas intrincadas paisagens genômicas incorporadas em amostras ecológicas, fornecendo mais do que perfis taxonômicos. Também oferece uma perspectiva genômica funcional, explorando os genes codificados e seus supostos papéis nos processos ambientais. Embora as abordagens tradicionais com sequenciamento Illumina tenham sido amplamente utilizadas em estudos metagenômicos, o advento do sequenciamento de leitura longa Nanopore e PacBio mudou o campo. As tecnologias Nanopore e PacBio aprimoram as análises bioinformáticas downstream, principalmente a montagem do metagenoma, garantindo montagens mais contínuas. Os relatórios indicam que a metagenômica baseada em Nanopore e PacBio gerou com sucesso genomas bacterianos completos e fechados a partir de microbiomas complexos (Moss, EL, et al., Nature Biotech, 2020). A integração das leituras do Nanopore com as leituras da Illumina fornece uma abordagem estratégica para correção de erros, mitigando a baixa precisão inerente do Nanopore. Esta combinação sinérgica aproveita os pontos fortes de cada plataforma de sequenciamento, oferecendo uma solução robusta para superar possíveis limitações e avançando na precisão e confiabilidade das análises metagenômicas.
Plataforma: Nanopore PromethION 48, Illumia e PacBio Revio
-

Sequenciamento de Amplicon 16S/18S/ITS-PacBio
Os genes 16S e 18S rRNA, juntamente com a região Internal Transcribed Spacer (ITS), servem como marcadores de impressão digital molecular essenciais devido à sua combinação de regiões altamente conservadas e hipervariáveis, tornando-os ferramentas inestimáveis para caracterizar organismos procarióticos e eucarióticos. A amplificação e o sequenciamento dessas regiões oferecem uma abordagem livre de isolamento para investigar a composição microbiana e a diversidade em vários ecossistemas. Embora o sequenciamento da Illumina normalmente tenha como alvo regiões hipervariáveis curtas, como V3-V4 de 16S e ITS1, foi demonstrado que a anotação taxonômica superior é alcançável pelo sequenciamento de todo o comprimento de 16S, 18S e ITS. Esta abordagem abrangente resulta em percentagens mais elevadas de sequências classificadas com precisão, alcançando um nível de resolução que se estende à identificação de espécies. A plataforma de sequenciamento em tempo real de molécula única (SMRT) da PacBio se destaca por fornecer leituras longas (HiFi) altamente precisas que cobrem os amplicons completos, rivalizando com a precisão do sequenciamento da Illumina. Esta capacidade permite aos investigadores obter uma vantagem incomparável – uma visão panorâmica da paisagem genética. A cobertura estendida eleva significativamente a resolução na anotação de espécies, particularmente em comunidades bacterianas ou fúngicas, permitindo uma compreensão mais profunda dos meandros das populações microbianas.
-

Sequenciamento de Amplicon 16S/18S/ITS-NGS
O sequenciamento de amplicon com tecnologia Illumina, visando especificamente os marcadores genéticos 16S, 18S e ITS, é um método poderoso para desvendar a filogenia, a taxonomia e a abundância de espécies nas comunidades microbianas. Esta abordagem envolve o sequenciamento das regiões hipervariáveis dos marcadores genéticos de manutenção. Originalmente introduzida como uma impressão digital molecular porWoeses et alem 1977, esta técnica revolucionou o perfil do microbioma, permitindo análises livres de isolamento. Através do sequenciamento de 16S (bactérias), 18S (fungos) e espaçador interno transcrito (ITS, fungos), os pesquisadores podem identificar não apenas espécies abundantes, mas também espécies raras e não identificadas. Amplamente adotado como uma ferramenta fundamental, o sequenciamento de amplicon tornou-se fundamental para discernir composições microbianas diferenciais em diversos ambientes, incluindo a boca humana, intestinos, fezes e muito mais.
-

Re-sequenciação completa do genoma bacteriano e fúngico
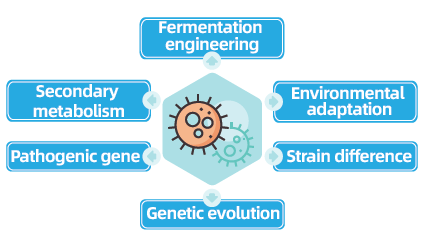
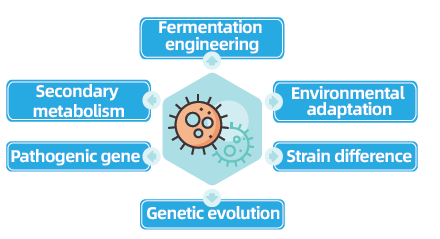
Os projetos de re-sequenciação do genoma completo de bactérias e fungos são fundamentais para o avanço da genômica microbiana, permitindo a conclusão e comparação de genomas microbianos. Isto facilita a engenharia de fermentação, a otimização de processos industriais e a exploração de vias de metabolismo secundário. Além disso, a re-sequenciação de fungos e bactérias é crucial para compreender a adaptação ambiental, optimizar estirpes e revelar a dinâmica da evolução genética, com amplas implicações na medicina, agricultura e ciências ambientais.
-

Sequenciamento de RNA procariótico
O sequenciamento de RNA permite o perfil abrangente de todos os transcritos de RNA dentro das células sob condições específicas. Esta tecnologia de ponta serve como uma ferramenta potente, revelando intrincados perfis de expressão genética, estruturas genéticas e mecanismos moleculares associados a diversos processos biológicos. Amplamente adotado em pesquisas fundamentais, diagnósticos clínicos e desenvolvimento de medicamentos, o sequenciamento de RNA oferece insights sobre as complexidades da dinâmica celular e da regulação genética. Nosso processamento de amostras de RNA procariótico é adaptado para transcriptomas procarióticos, envolvendo depleção de rRNA e preparação de biblioteca direcional.
Plataforma: Illumina NovaSeq
-

Sequenciamento de Metatranscriptoma
Aproveitando a tecnologia de sequenciamento da Illumina, o serviço de sequenciamento de metatranscriptoma da BMKGENE revela a expressão genética dinâmica de uma gama diversificada de micróbios, abrangendo eucariotos, procariontes e vírus, em ambientes naturais como solo, água, mar, fezes e intestino. Nosso serviço abrangente capacita os pesquisadores a se aprofundarem nos perfis completos de expressão gênica de comunidades microbianas complexas. Além da análise taxonômica, nosso serviço de sequenciamento de metatranscriptoma facilita a exploração do enriquecimento funcional, esclarecendo genes expressos diferencialmente e suas funções. Descubra uma riqueza de insights biológicos enquanto navega pelas paisagens complexas de expressão genética, diversidade taxonômica e dinâmica funcional dentro desses diversos nichos ambientais.
-

Conjunto de genoma fúngico de novo
A BMKGENE oferece soluções versáteis para genomas de fungos, atendendo a diversas necessidades de pesquisa e à integridade desejada do genoma. A utilização apenas do sequenciamento Illumina de leitura curta permite a geração de um rascunho do genoma. Sequenciamento de leituras curtas e longas usando Nanopore ou Pacbio são combinados para um genoma fúngico mais refinado com contigs mais longos. Além disso, a integração do sequenciamento Hi-C aumenta ainda mais as capacidades, permitindo a obtenção de um genoma completo em nível cromossômico.
-

Montagem do genoma bacteriano de novo
Oferecemos um serviço completo de montagem de genoma bacteriano, garantindo 0 lacunas. Isso é possível integrando tecnologias de sequenciamento de leitura longa, como Nanopore e PacBio para montagem e sequenciamento de leitura curta com Illumina para validação de montagem e correção de erros de leituras ONT. Nosso serviço fornece o fluxo de trabalho completo de bioinformática desde montagem, anotação funcional e análise bioinformática avançada, atendendo objetivos específicos de pesquisa. Este serviço permite o desenvolvimento de genomas de referência precisos para diversos estudos genéticos e genômicos. Além disso, constitui a base para aplicações como otimização de cepas, engenharia genética e desenvolvimento de tecnologia microbiana, garantindo dados genômicos confiáveis e sem lacunas, cruciais para o avanço dos conhecimentos científicos e da inovação biotecnológica.