Sequenciamento de Exoma Completo Humano
Recursos de serviço
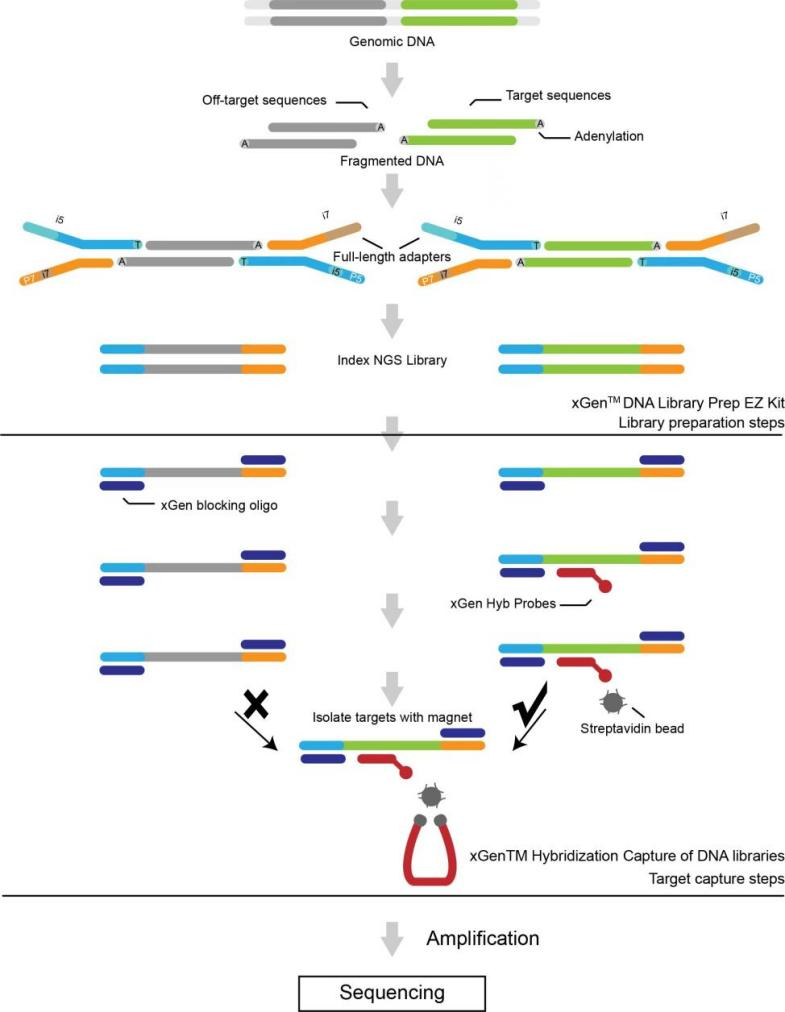
● Dois painéis de exoma disponíveis com base no enriquecimento de alvo com sondas: Sure Select Human All Exon v6 (Agilent) e xGen Exome Hybridization Panel v2 (IDT).
● Sequenciamento no Illumina NovaSeq.
● Pipeline de bioinformática direcionado para análise de doenças ou análise de tumores.
Vantagens do serviço
●Tem como alvo a região de codificação de proteínas: Ao capturar e sequenciar regiões codificadoras de proteínas, o hWES é utilizado para revelar variantes relacionadas à estrutura da proteína.
●Custo Efetivo:hWES produz aproximadamente 85% das mutações associadas a doenças humanas de 1% do genoma humano.
●Alta precisão: Com alta profundidade de sequenciamento, o hWES facilita a detecção de variantes comuns e variantes raras com frequências inferiores a 1%.
●Rigoroso controle de qualidade: Implementamos cinco pontos de controle principais em todas as etapas, desde a preparação de amostras e bibliotecas até sequenciamento e bioinformática. Esse monitoramento meticuloso garante a entrega de resultados consistentemente de alta qualidade.
●Análise Abrangente de Bioinformática: nosso pipeline vai além da identificação de variações no genoma de referência, pois incorpora análises avançadas projetadas para abordar especificamente questões de pesquisa relacionadas a aspectos genéticos de doenças ou análise de tumores.
●Suporte pós-venda:Nosso compromisso vai além da conclusão do projeto, com um período de serviço pós-venda de 3 meses. Durante esse período, oferecemos acompanhamento do projeto, assistência na solução de problemas e sessões de perguntas e respostas para esclarecer quaisquer dúvidas relacionadas aos resultados.
Especificações de amostra
| Estratégia de Captura de Exons | Estratégia de sequenciamento | Saída de dados recomendada |
| Sure Select Humano Todos Exon v6 (Agilent) ou painel de hibridização xGen Exome v2 (IDT)
| Illumina NovaSeq PE150 | 5 -10 GB Para distúrbios mendelianos/doenças raras: > 50x Para amostras de tumor: ≥ 100x |
Requisitos de amostra
| Tipo de amostra
| Quantia(Qubit®)
| Concentração | Volume
| Pureza (NanoDrop™) |
|
DNA genômico
| ≥ 50ng | ≥ 6 ng/μL | ≥ 15 μL | DO260/280=1,8-2,0 sem degradação, sem contaminação
|
Bioinformática

A análise bioinformática de amostras da doença hWES inclui:
● CQ de dados de sequenciamento
● Alinhamento do Genoma de Referência
● Identificação de SNPs e InDels
● Anotação Funcional de SNPs e InDels

A análise bioinformática de amostras de tumores inclui:
● CQ de dados de sequenciamento
● Alinhamento do Genoma de Referência
● Identificação de SNPs, InDels e variações somáticas
● Identificação de variantes germinativas
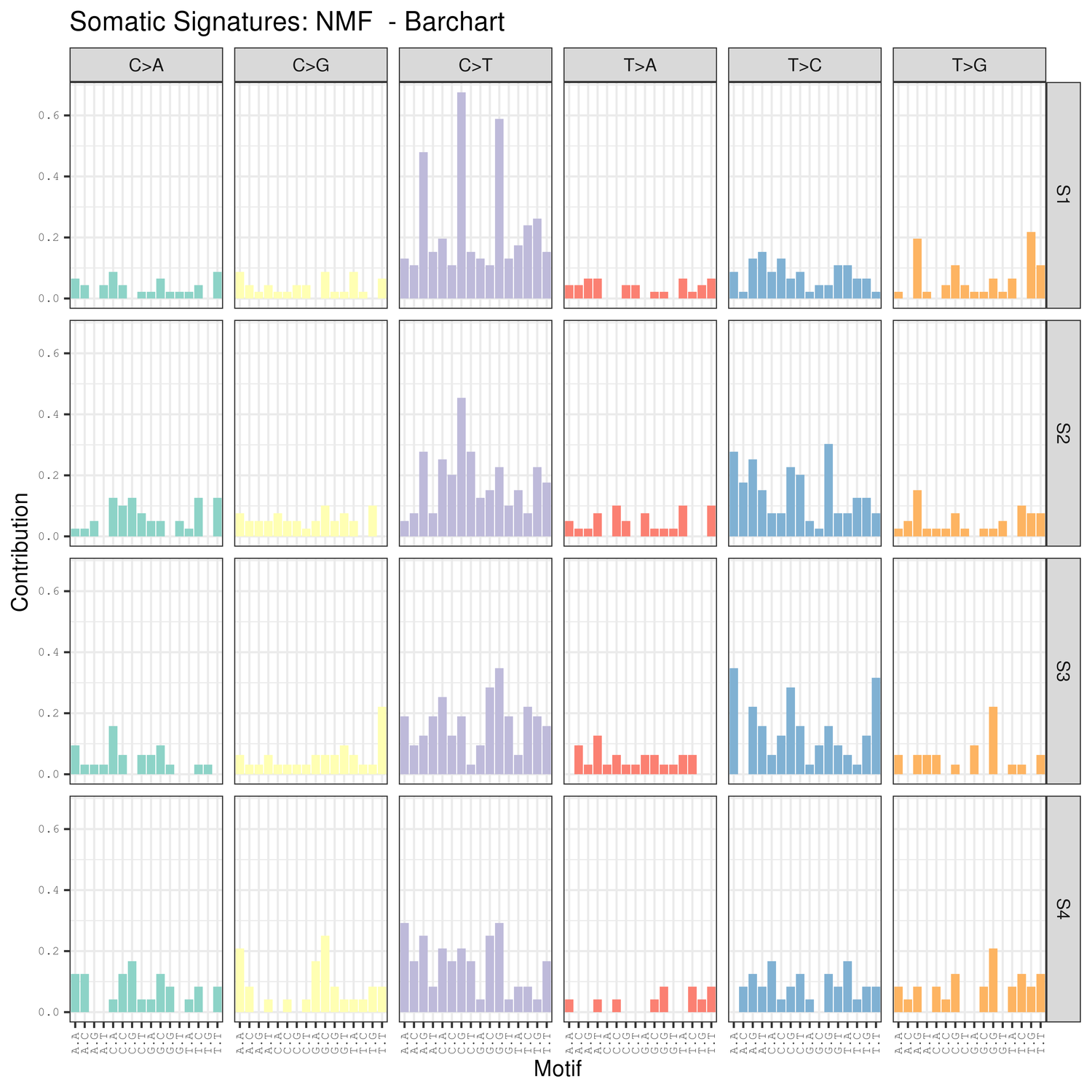
● Análise de assinaturas de mutação
● Identificação de genes impulsionadores com base em mutações de ganho de função
● Anotação de mutação no nível de suscetibilidade a medicamentos
● Análise de heterogeneidade – cálculo de pureza e ploidia
Fluxo de trabalho de serviço

Entrega de amostra

Extração de DNA

Construção de biblioteca

Sequenciamento

Análise de dados

Entrega de dados

Serviços pós-venda
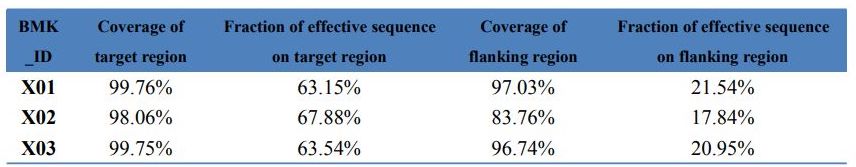
CQ de dados – Estatísticas de captura de Exome
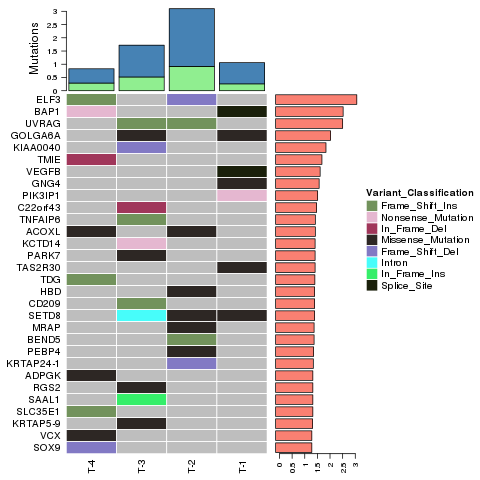
Identificação de variante – InDels
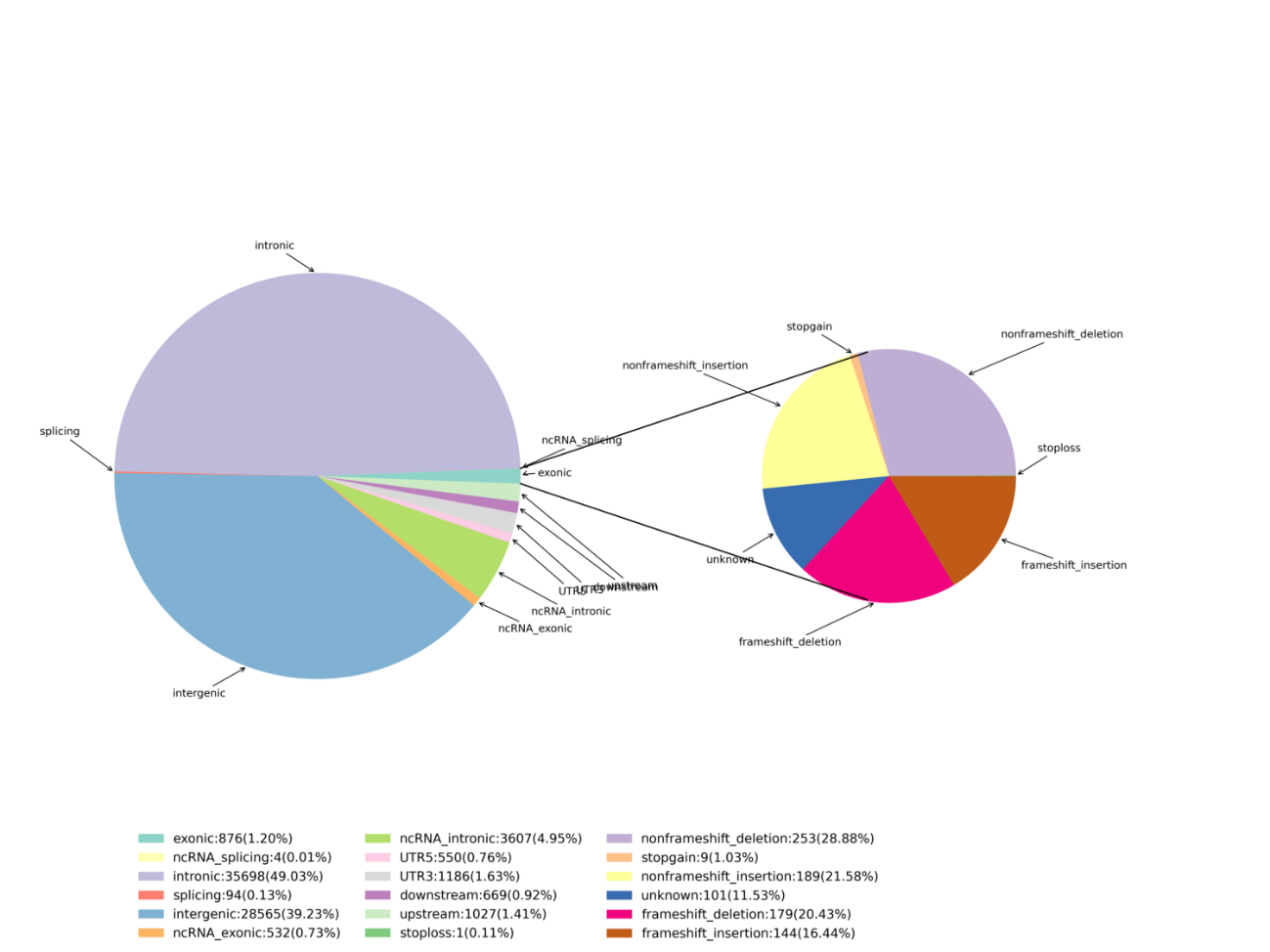
Análise avançada: identificação e distribuição de SNPs/InDels deletérios – Gráfico Circos
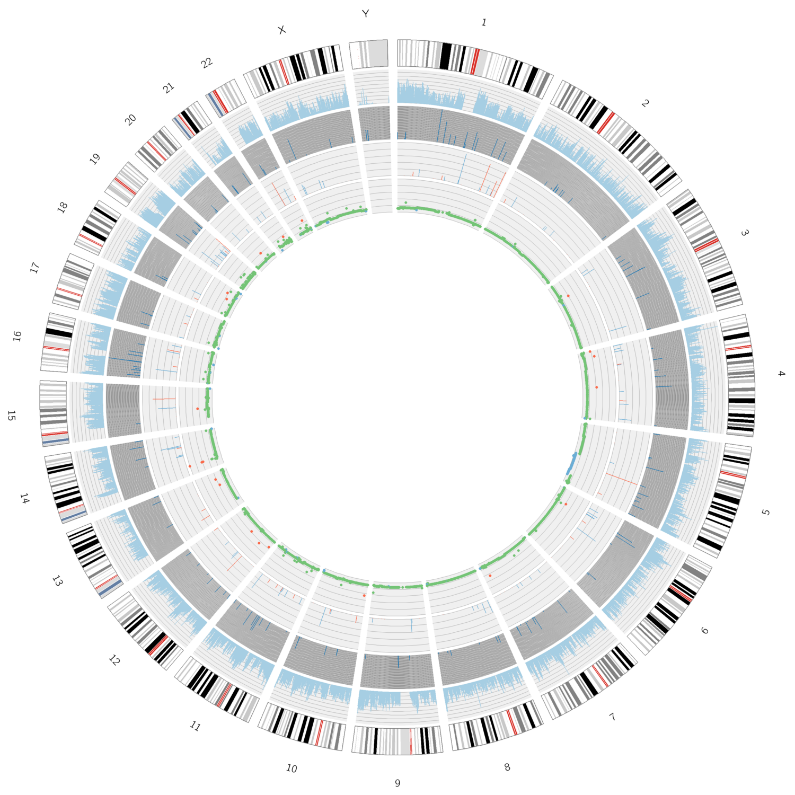
Análise tumoral: identificação e distribuição de mutações somáticas – Gráfico Circos
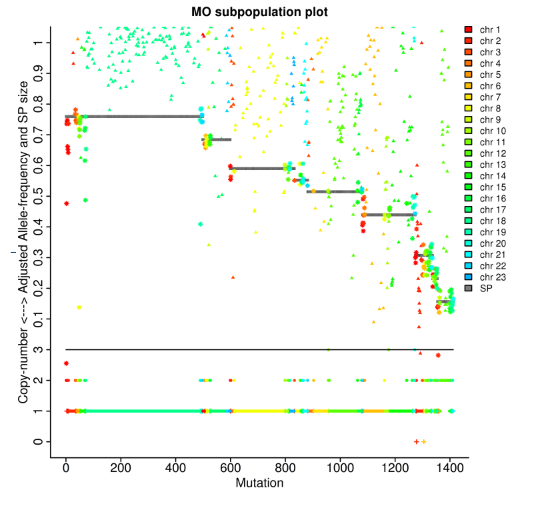
Análise tumoral: linhagens clonais