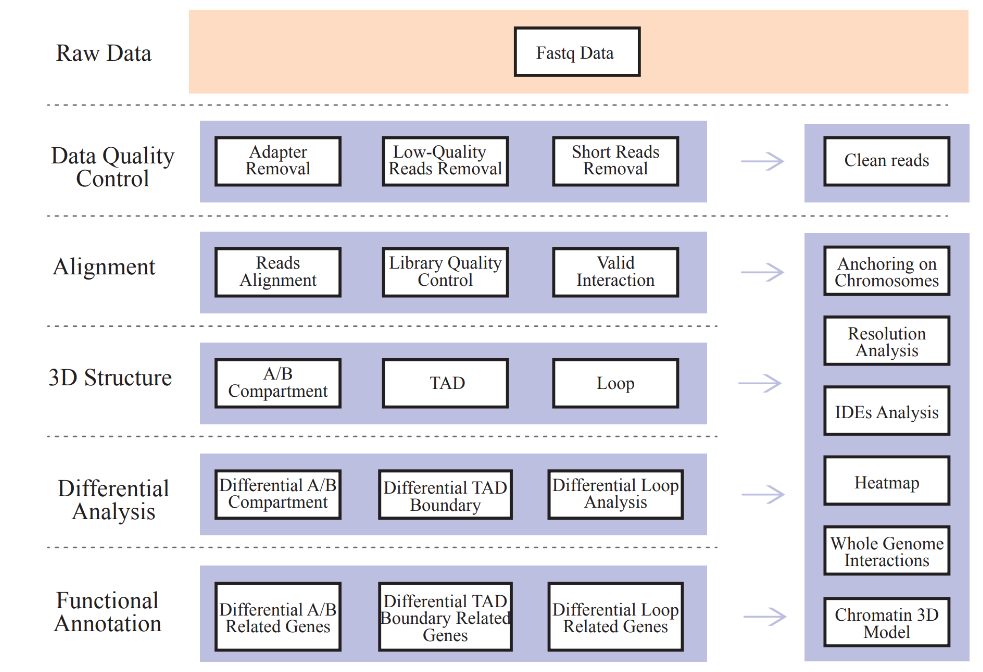
Interação de cromatina baseada em Hi-C
Recursos de serviço
● Sequenciamento no Illumina NovaSeq com PE150.
● O serviço requer amostras de tecido, em vez de ácidos nucleicos extraídos, para reticulação com formaldeído e conservação das interações DNA-proteína.
● O experimento Hi-C envolve restrição e reparo final das pontas pegajosas com biotina, seguido pela circularização das pontas rombas resultantes, preservando as interações. O DNA é então extraído com esferas de estreptavidina e purificado para posterior preparação da biblioteca.
Vantagens do serviço
●Design ideal de enzimas de restrição: para garantir uma alta eficiência de Hi-C em diferentes espécies com até 93% de pares de interação válidos.
●Ampla experiência e registros de publicação:A BMKGene tem vasta experiência com mais de 2.000 projetos de sequenciamento Hi-C de 800 espécies diferentes e diversas patentes. Mais de 100 casos publicados com fator de impacto acumulativo superior a 900.
●Equipe de Bioinformática altamente qualificada:com patentes internas e direitos autorais de software para experimentos Hi-C e análise de dados e um software de visualização de dados desenvolvido pela própria empresa.
●Suporte pós-venda:Nosso compromisso vai além da conclusão do projeto, com um período de serviço pós-venda de 3 meses. Durante esse período, oferecemos acompanhamento do projeto, assistência na solução de problemas e sessões de perguntas e respostas para esclarecer quaisquer dúvidas relacionadas aos resultados.
●Anotação abrangente: utilizamos múltiplos bancos de dados para anotar funcionalmente os genes com variações identificadas e realizar a análise de enriquecimento correspondente, fornecendo insights sobre múltiplos projetos de pesquisa.
Especificações de serviço
| Biblioteca | Estratégia de sequenciamento | Saída de dados recomendada | Resolução de sinal Hi-C |
| Biblioteca Hi-C | Illumina PE150 | Loop de cromatina: 150x TAD: 50x | Loop de cromatina: 10Kb TAD: 40Kb |
Requisitos de serviço
| Tipo de amostra | Quantidade necessária |
| Tecido animal | ≥2g |
| Sangue Total | ≥2mL |
| Fungos | ≥1g |
| Planta - tecido jovem | 1g/alíquota, 2-4 alíquotas recomendadas |
| Células cultivadas | ≥1x107 |
Inclui a seguinte análise:
● CQ de dados brutos;
● Mapeamento e QC da biblioteca Hi-C: pares de interação válidos e Interaction Decay Exponents (IDEs);
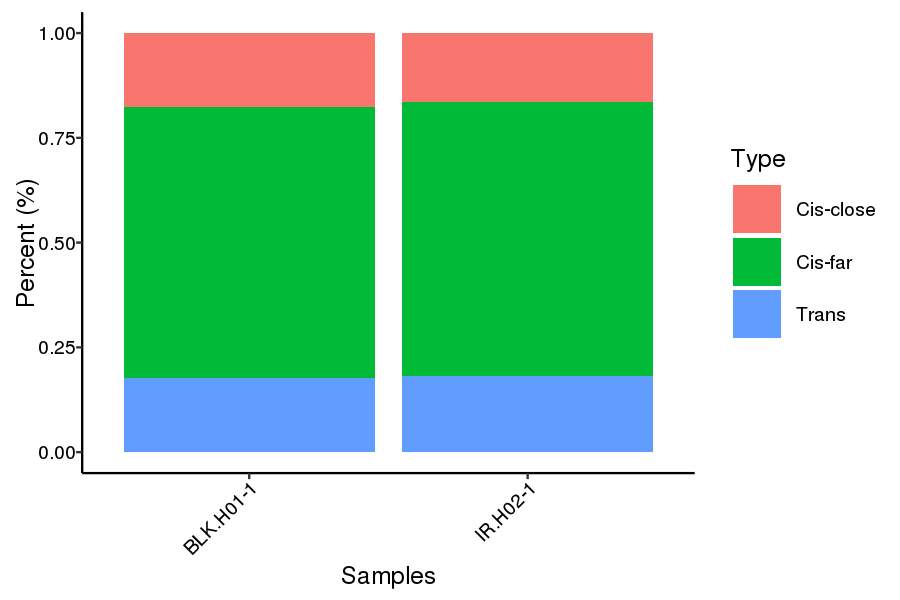
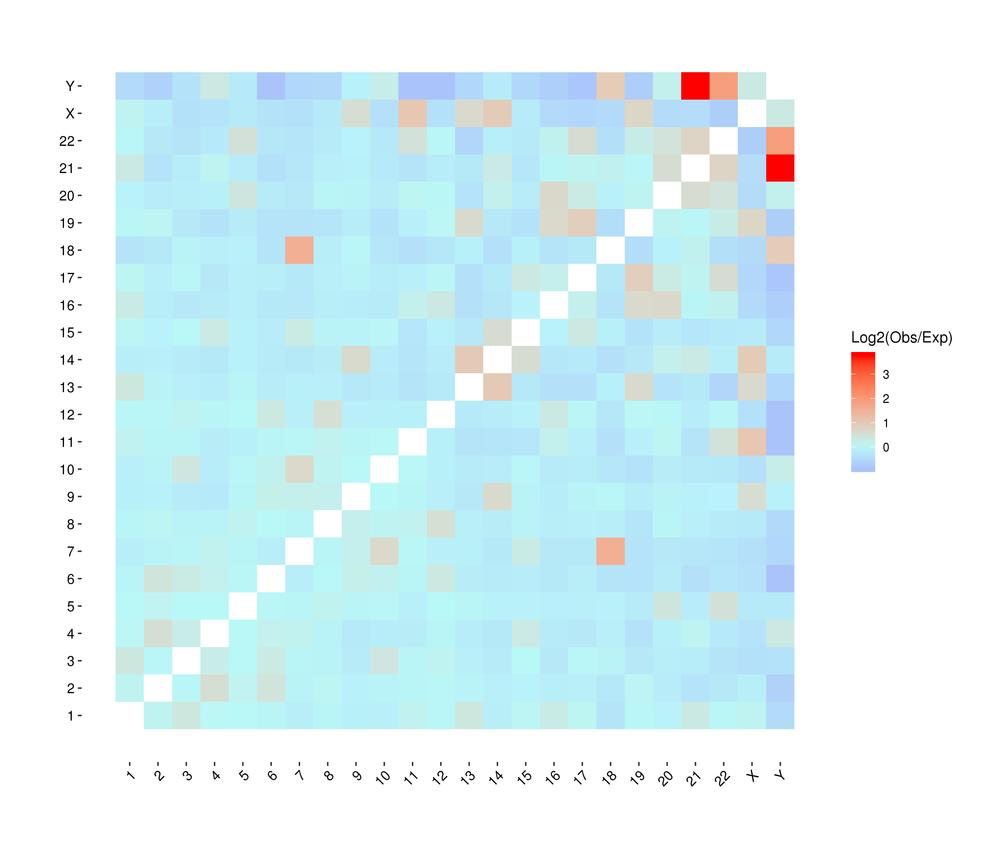
● Perfil de interação genômica ampla: análise cis/trans e mapa de interação Hi-C;
● Análise da distribuição dos compartimentos A/B;
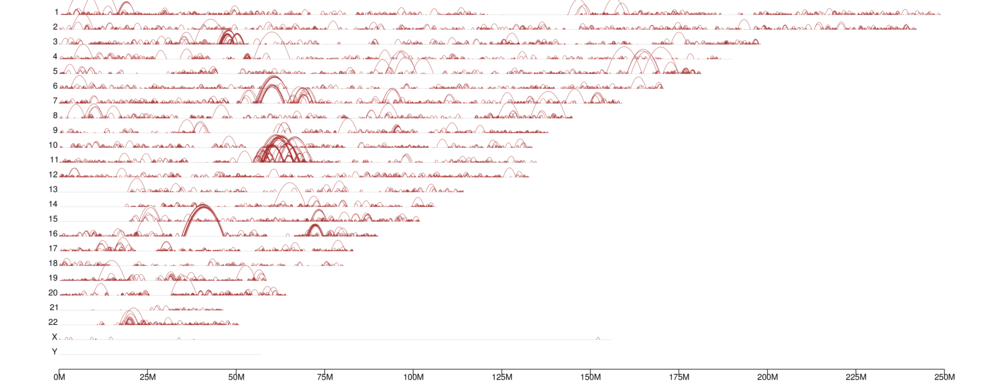
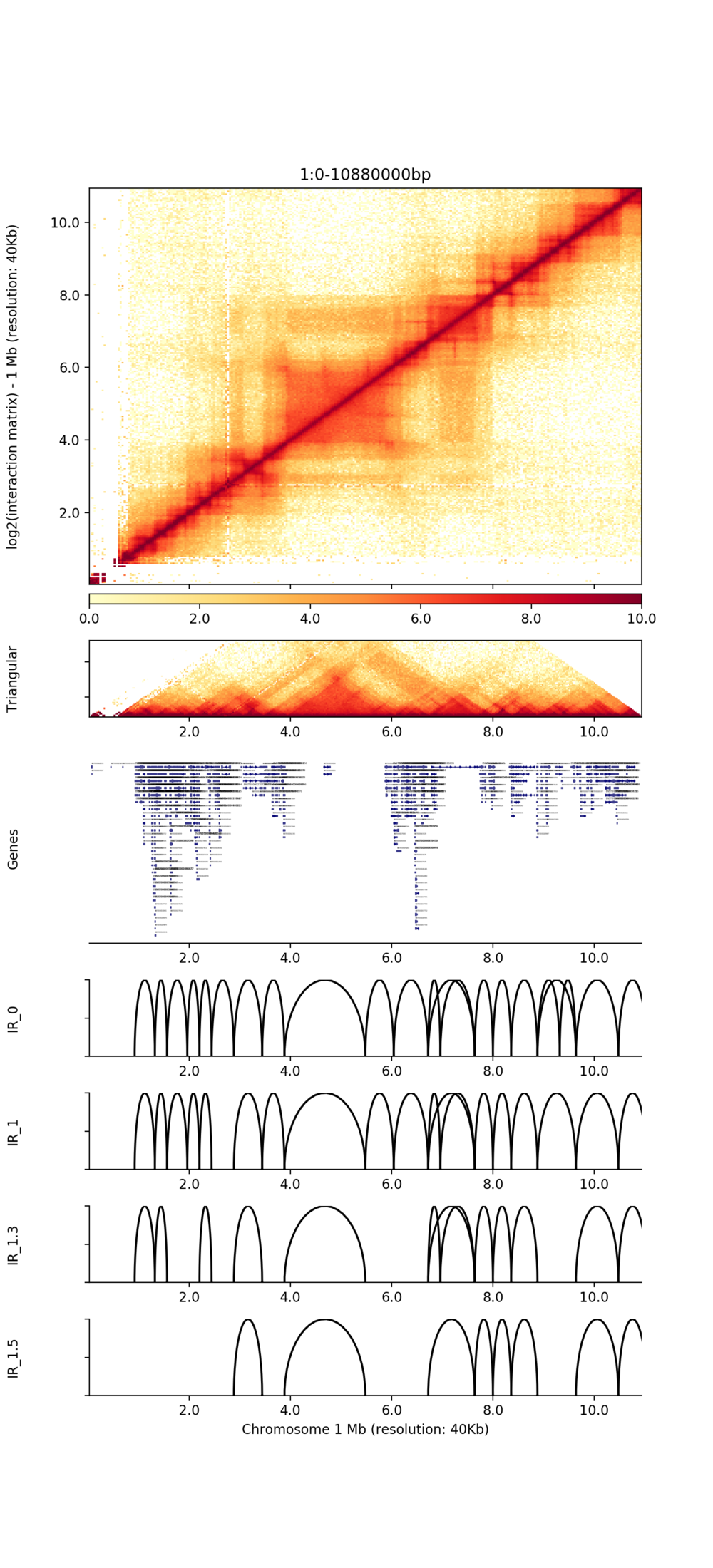
● Identificação de TADs e alças de cromatina;
● Análise diferencial de elementos da estrutura da cromatina 3D entre amostras e correspondente anotação funcional de genes associados.
Distribuição de proporção cis e trans
Mapa térmico de interações cromossômicas entre amostras
Distribuição de compartimentos A/B em todo o genoma
Distribuição de alças de cromatina em todo o genoma
Visualização de TADs
Explore os avanços da pesquisa facilitados pelos serviços de sequenciamento Hi-C da BMKGene por meio de uma coleção selecionada de publicações.
Meng, T. et al. (2021) 'Uma análise comparativa integrada multiômica identifica CA2 como um novo alvo para cordoma',Neuro-Oncologia, 23(10), pp. doi: 10.1093/NEUONC/NOAB156.
Xu, L. et al. (2021) 'A desorganização 3D e o rearranjo do genoma fornecem insights sobre a patogênese da NAFLD por meio de sequenciamento integrado de Hi-C, Nanopore e RNA',Acta Pharmaceutica Sinica B, 11(10), pp. doi: 10.1016/J.APSB.2021.03.022.