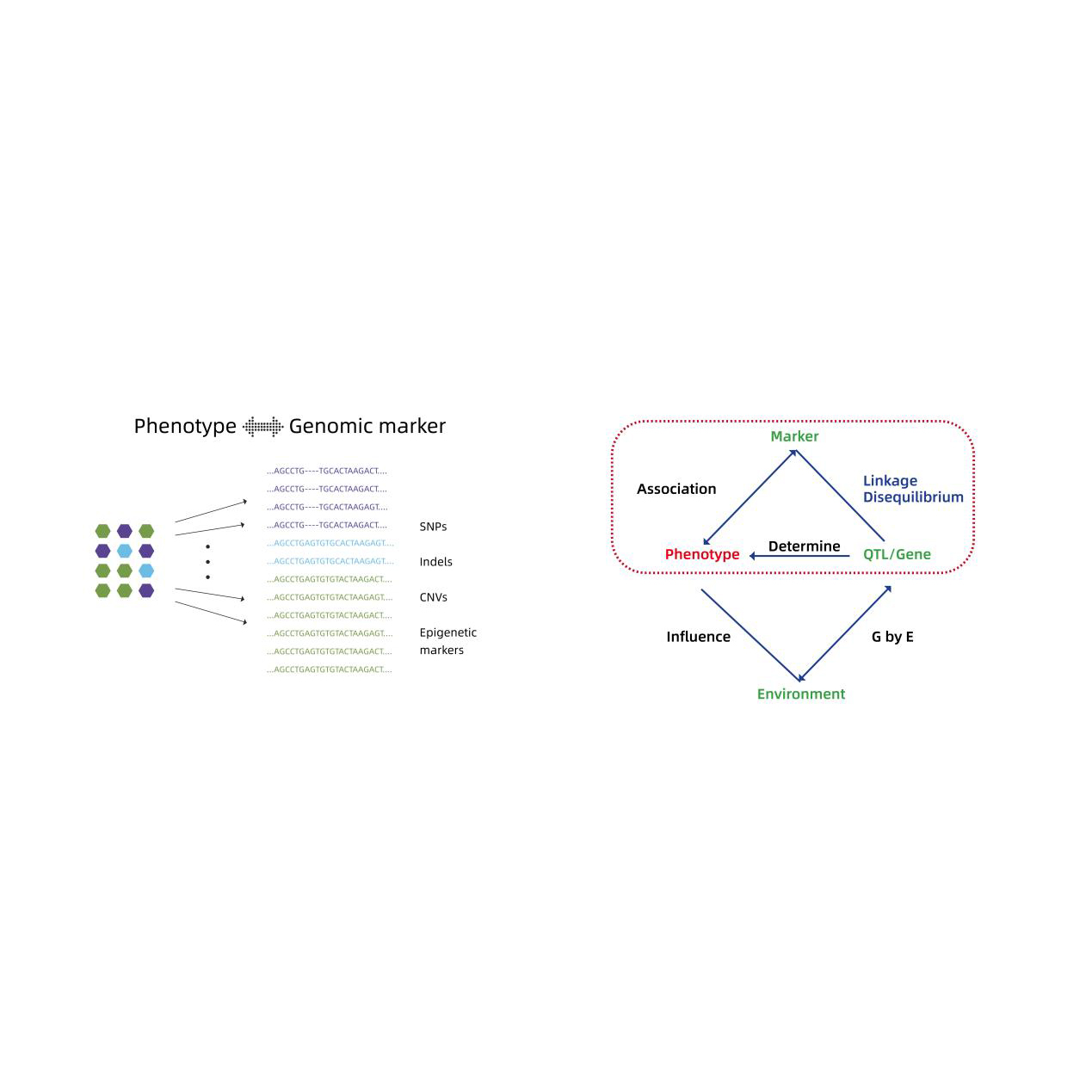
Análise de associação em todo o genoma
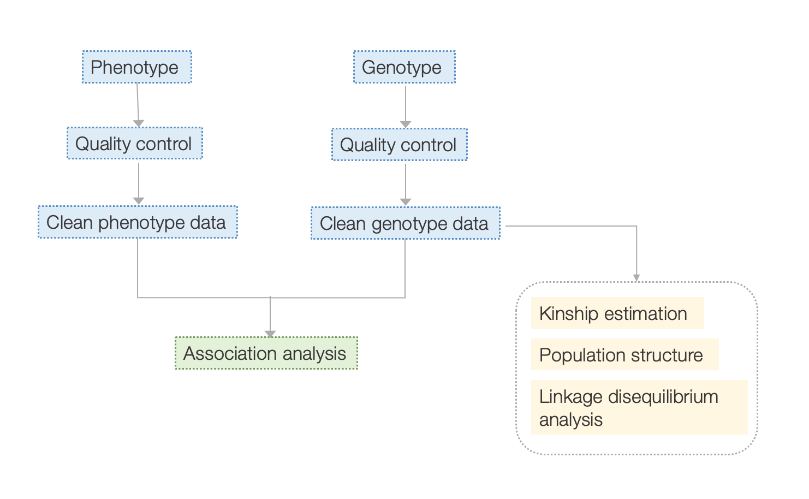
Fluxo de trabalho

Vantagens de serviço
●Extensos conhecimentos e registros de publicação: Com experiência acumulada no GWAS, o BMKGENE concluiu centenas de projetos de espécies na pesquisa da População GWAS, ajudou os pesquisadores a publicar mais de 100 artigos e o fator de impacto cumulativo atingiu 500.
● Análise abrangente de bioinformática: O fluxo de trabalho inclui análise da Associação de T-Armas de SNP, fornecendo um conjunto de genes candidatos e sua anotação funcional correspondente.
●Equipe de bioinformática altamente qualificada e ciclo de análise curta: Com ótima experiência em análise de genômica avançada, a equipe da BMKGENE oferece análises abrangentes com um tempo rápido de resposta.
●Suporte pós-venda:Nosso compromisso se estende além da conclusão do projeto com um período de serviço pós-venda de três meses. Durante esse período, oferecemos acompanhamento do projeto, assistência de solução de problemas e sessões de perguntas e respostas para abordar quaisquer consultas relacionadas aos resultados.
Especificações e requisitos de serviço
| Tipo de sequenciamento | Escala populacional recomendada | Estratégia de sequenciamento | Requisitos de nucleotídeo |
| Sequenciamento de genoma inteiro | 200 amostras | 10x | Concentração: ≥ 1 ng/ µl Valor total 5Ng Limitada ou sem degradação ou contaminação |
| Fragmento amplificado de Locus específico (SLAF) | Profundidade da tag: 10x Número de tags: <400 MB: WGS é recomendado <1 GB: tags 100k 1 GB > 2 GB: tags 300k Tags máximas de 500k | Concentração ≥ 5 ng/µl Quantidade total ≥ 80 ng Nanodrop OD260/280 = 1,6-2.5 Gel de agarose: sem degradação ou contaminação limitada
|
Seleção de material



Diferentes variedades, subespécies, Landraces/Genebanks/Families Mixed/Wild Resources
Diferentes variedades, subespécies, LandRaces
Half-Sib Family/Family Family/Wild Resources
Fluxo de trabalho de serviço

Projeto de experimento

Entrega de amostra

Extração de RNA

Construção da biblioteca

Sequenciamento

Análise de dados

Serviços pós-venda
Inclui a seguinte análise:
- Análise de associação em todo o genoma: LM, LMM, Emmax, Fastlmm Modelo
- Anotação funcional de genes candidatos
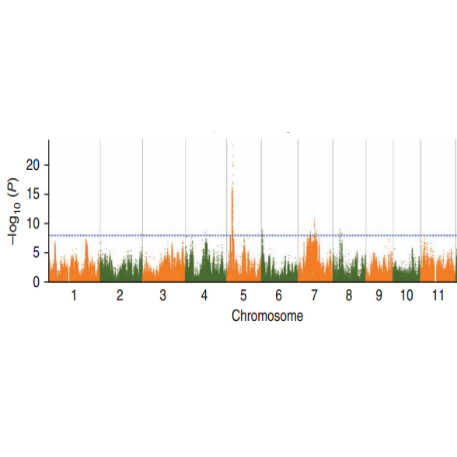
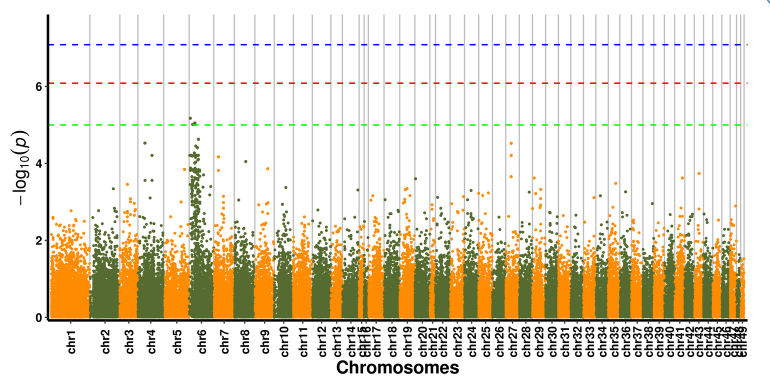
Análise da Associação SNP-Plotagem de Manhattan
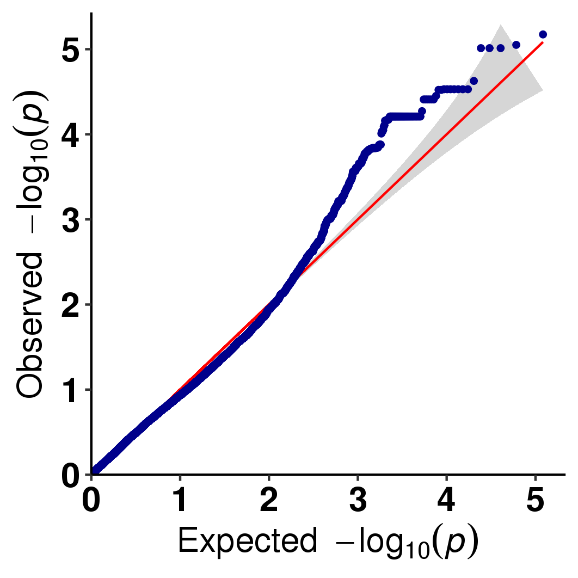
Análise da Associação SNP-Traite-Plot QQ
Explore os avanços facilitados pelos serviços de GWAS da BMKGENE por meio de uma coleção de publicações com curadoria:
LV, L. et al. (2023) 'Insight sobre a base genética da tolerância à amônia em Razor Clam Sinonovacula Constricta pelo estudo da associação em todo o genoma',Aquicultura569, p. 739351. Doi: 10.1016/j.aquacultura.2023.739351.
Li, X. et al. (2022) 'Análises multi-térmicas de 398 acessos de milho de Foxtail revelam regiões genômicas associadas a domesticação, características de metabólitos e efeitos anti-inflamatórios',Planta molecular, 15 (8), pp. 1367-1383. doi: 10.1016/j.molp.2022.07.003.
Li, J. et al. (2022) 'Mapeamento de associação em todo o genoma de fenótipos maltrusos em ambiente de seca',Fronteiras na Ciência Planta, 13, p. 924892. Doi: 10.3389/fpls.2022.924892/bibtex.
Zhao, X. et al. (2021) 'GMST1, que codifica uma sulfotransferase, confere resistência às cepas de vírus do mosaico de soja G2 e G3',Planta, células e meio ambiente, 44 (8), pp. 2777-2792. doi: 10.1111/pce.14066.