Sequenciamento completo de mRNA-Nanopore
Características
● Captura de mRNA poli-A seguida de síntese de cDNA e preparação de biblioteca
● Sequenciamento das transcrições completas
● Análise bioinformática baseada no alinhamento a um genoma de referência
● A análise bioinformática inclui não apenas a expressão em nível de gene e isoforma, mas também a análise de lncRNA, fusões gênicas, poliadenilação e estrutura gênica
Vantagens do serviço
●Quantificação da expressão no nível da isoforma: permitindo análise de expressão detalhada e precisa, revelando alterações que podem ser mascaradas ao analisar toda a expressão genética
●Demandas de dados reduzidas:Comparado ao sequenciamento de próxima geração (NGS), o sequenciamento Nanopore apresenta requisitos de dados mais baixos, permitindo níveis equivalentes de saturação de quantificação da expressão gênica com dados menores.
●Maior precisão na quantificação da expressão: tanto no nível do gene quanto da isoforma
●Identificação de informações transcriptômicas adicionais: poliadenilação alternativa, genes de fusão e lcnRNA e seus genes alvo
●Ampla experiência: Nossa equipe traz uma vasta experiência para cada projeto, tendo concluído mais de 850 projetos completos de transcriptoma Nanopore e processado mais de 8.000 amostras.
●Suporte pós-venda: nosso compromisso vai além da conclusão do projeto com um período de serviço pós-venda de 3 meses. Durante esse período, oferecemos acompanhamento do projeto, assistência na solução de problemas e sessões de perguntas e respostas para esclarecer quaisquer dúvidas relacionadas aos resultados.
Requisitos de amostra e entrega
| Biblioteca | Estratégia de sequenciamento | Dados recomendados | Controle de qualidade |
| Poli A enriquecido | Illumina PE150 | 6/12 GB | Índice de qualidade médio: Q10 |
Requisitos de amostra:
Nucleotídeos:
| Conc.(ng/μl) | Quantidade (μg) | Pureza | Integridade |
| ≥ 100 | ≥ 1,0 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 Contaminação limitada ou inexistente de proteínas ou DNA mostrada no gel. | Para plantas: RIN≥7,0; Para animais: RIN≥7,5; 5,0≥28S/18S≥1,0; elevação limitada ou nenhuma elevação da linha de base |
● Plantas:
Raiz, Caule ou Pétala: 450 mg
Folha ou Semente: 300 mg
Fruta: 1,2g
● Animal:
Coração ou Intestino: 300 mg
Vísceras ou Cérebro: 240 mg
Músculo: 450 mg
Ossos, Cabelo ou Pele: 1g
● Artrópodes:
Insetos: 6g
Crustáceos: 300 mg
● Sangue total: 1 tubo
● Células: 106 células
Entrega de amostra recomendada
Recipiente: tubo de centrífuga de 2 ml (folha de estanho não é recomendada)
Rotulagem da amostra: Grupo+réplica, por exemplo, A1, A2, A3; B1, B2, B3.
Expedição:
1. Gelo seco: As amostras precisam ser embaladas em sacos e enterradas em gelo seco.
2. Tubos RNAstable: As amostras de RNA podem ser secas em tubo de estabilização de RNA (por exemplo, RNAstable®) e enviadas em temperatura ambiente.
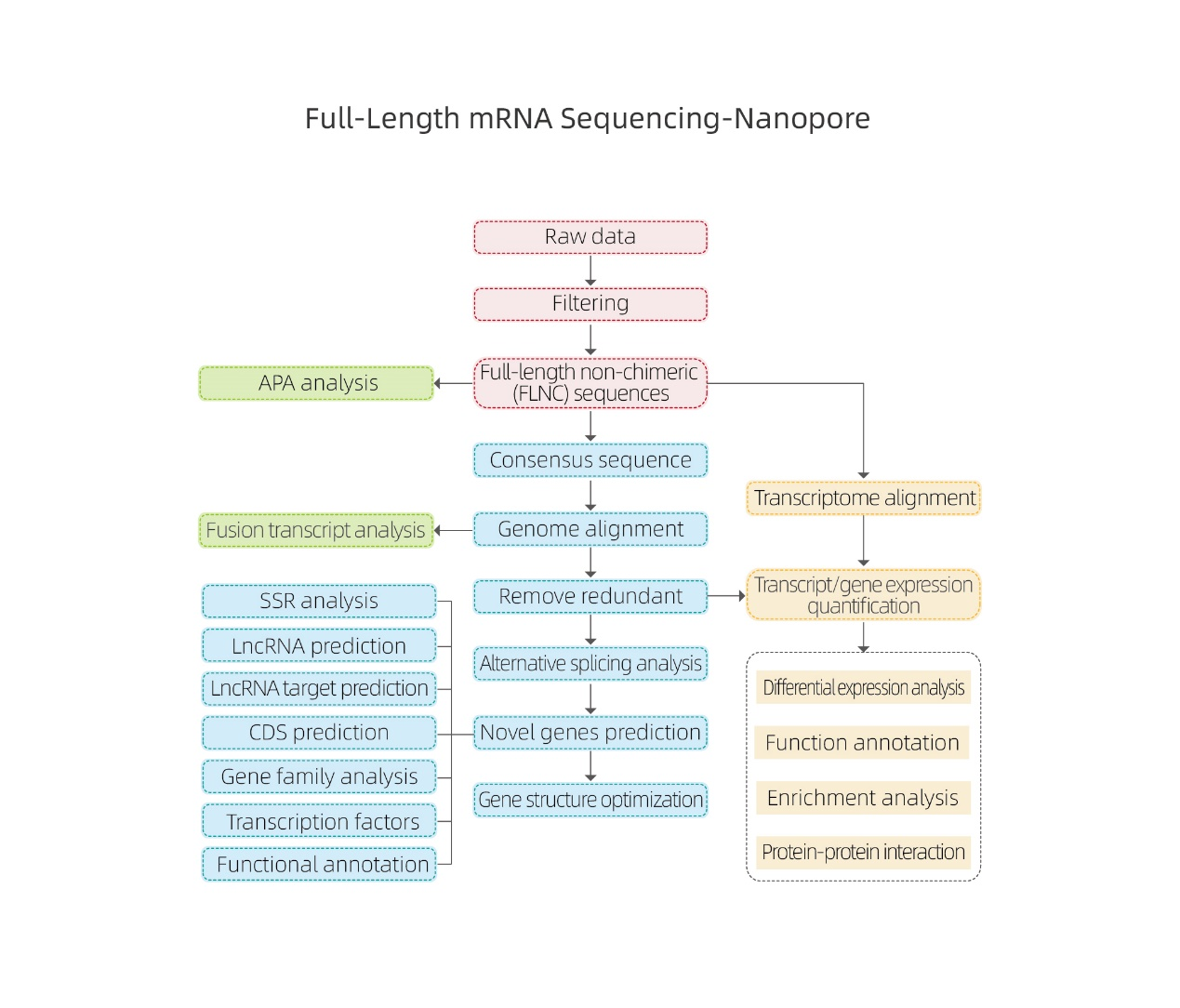
Fluxo de trabalho de serviço
Nucleotídeos:

Entrega de amostra

Construção de biblioteca

Sequenciamento

Análise de dados

Serviços pós-venda
Fluxo de trabalho de serviço
Tecido:

Projeto de experimento

Entrega de amostra

Extração de RNA

Construção de biblioteca

Sequenciamento

Análise de dados

Serviços pós-venda
● Processamento de dados brutos
● Identificação da transcrição
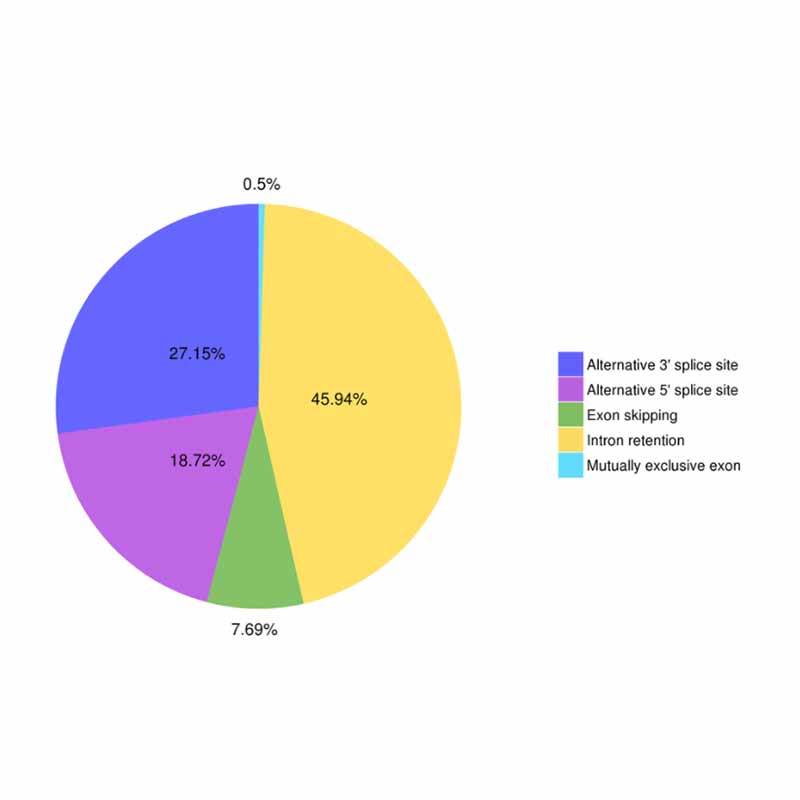
● Emenda alternativa
● Quantificação de expressão em nível de gene e nível de isoforma
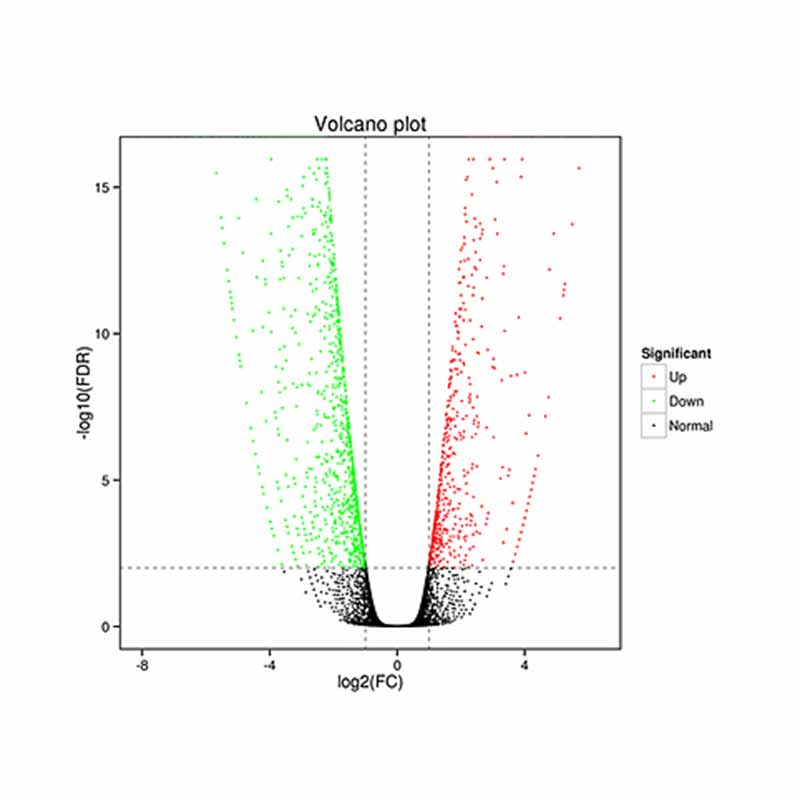
● Análise de expressão diferencial
● Anotação e enriquecimento de função (DEGs e DETs)
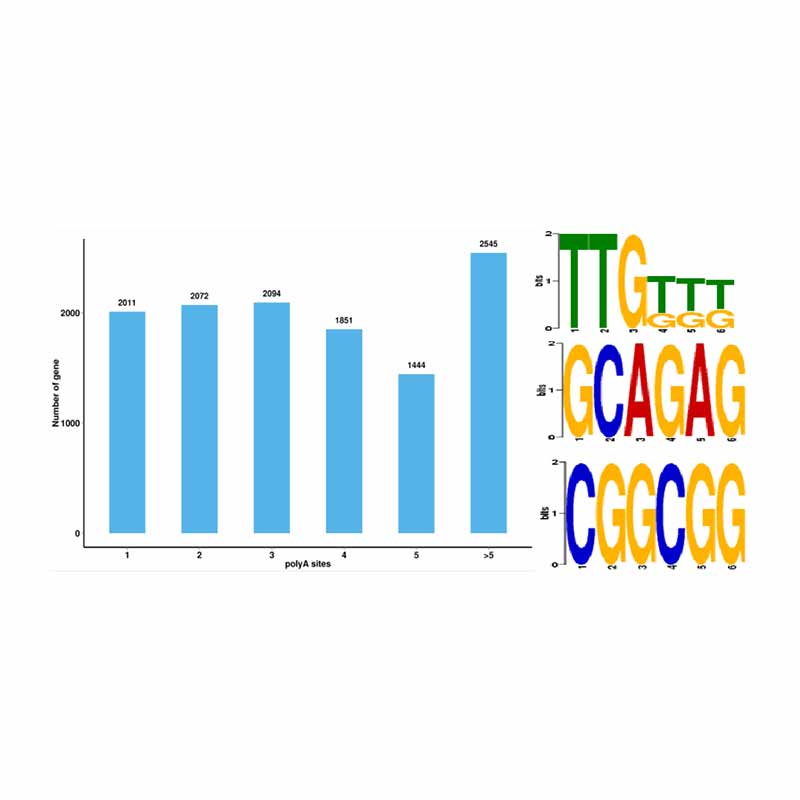
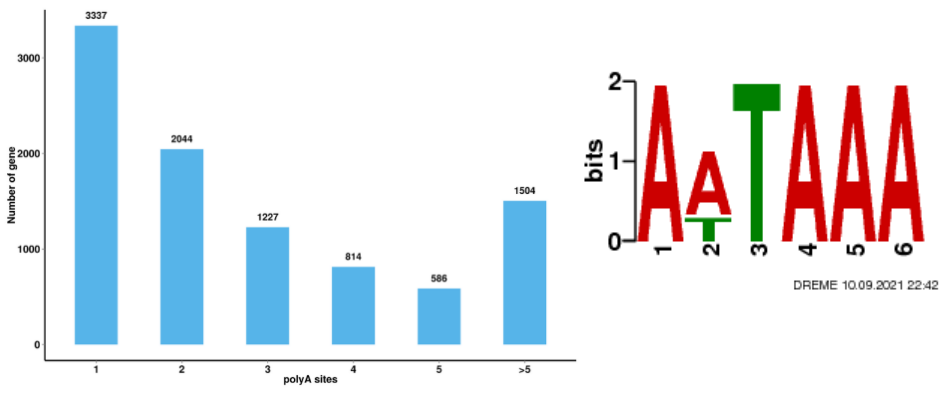
Análise de emenda alternativa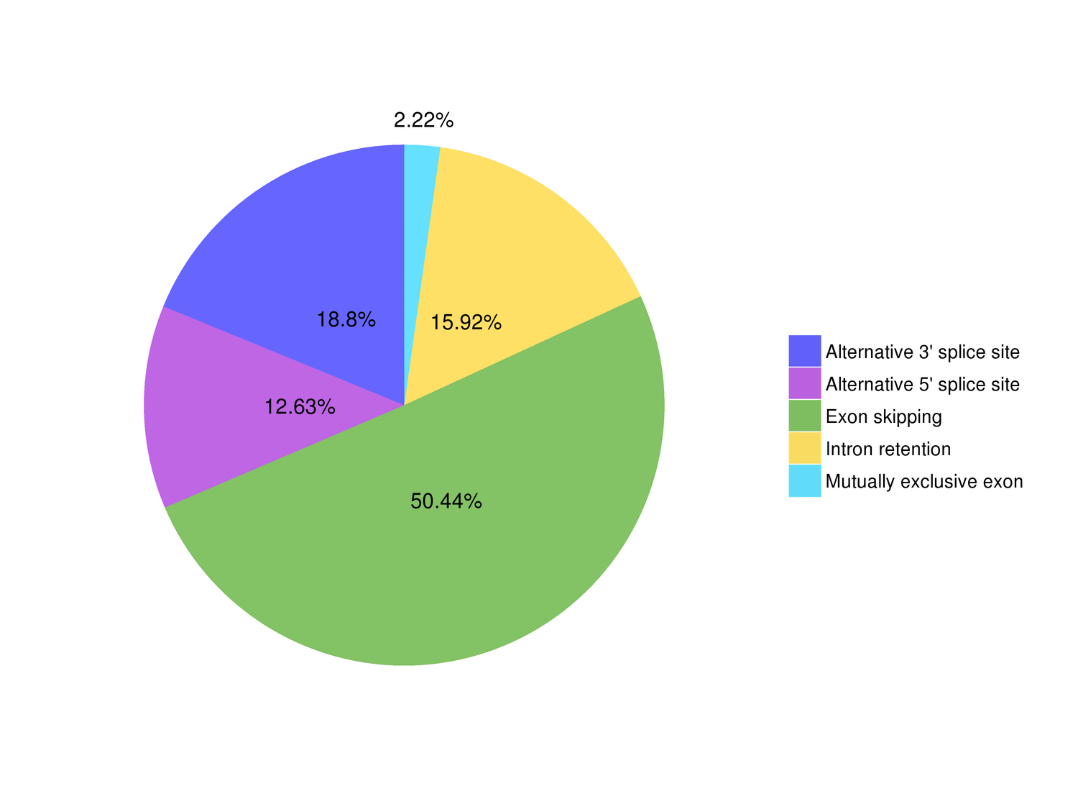 Análise Alternativa de Poliadenilação (APA)
Análise Alternativa de Poliadenilação (APA)
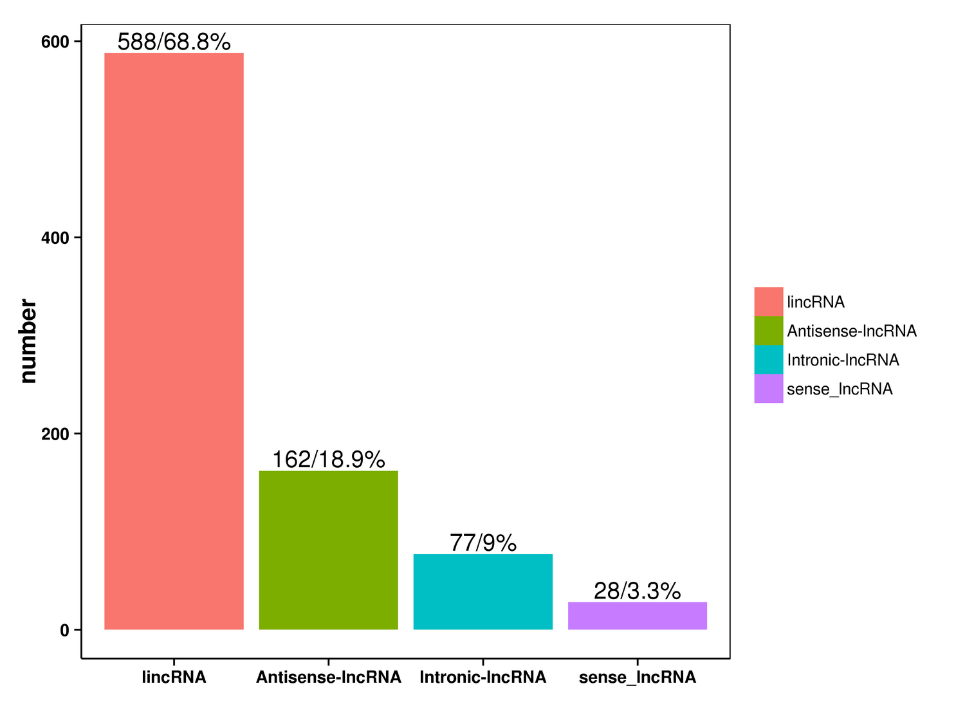
Previsão de lncRNA
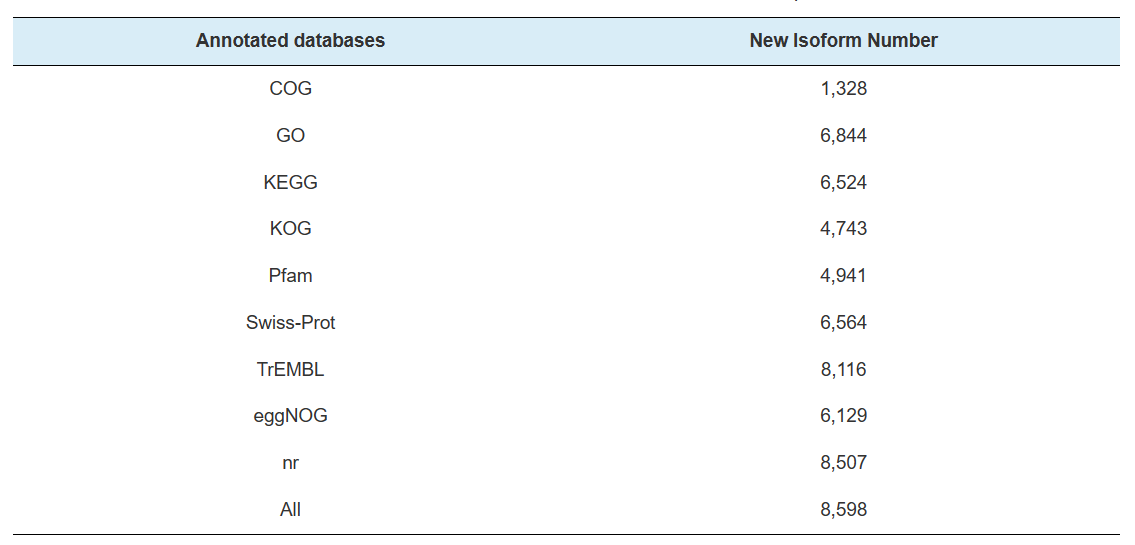
Anotação de novos genes
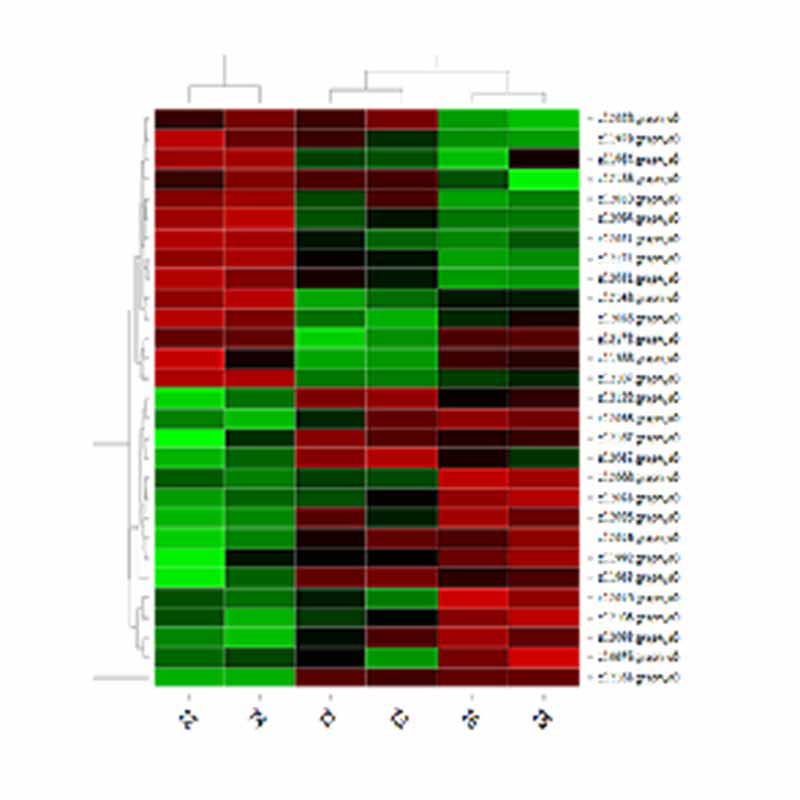
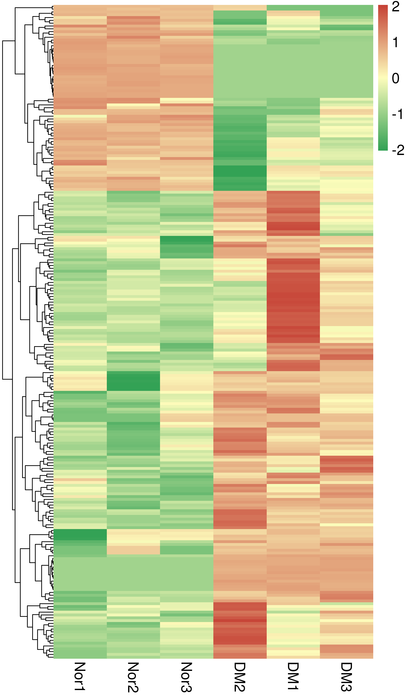
Agrupamento de DERs
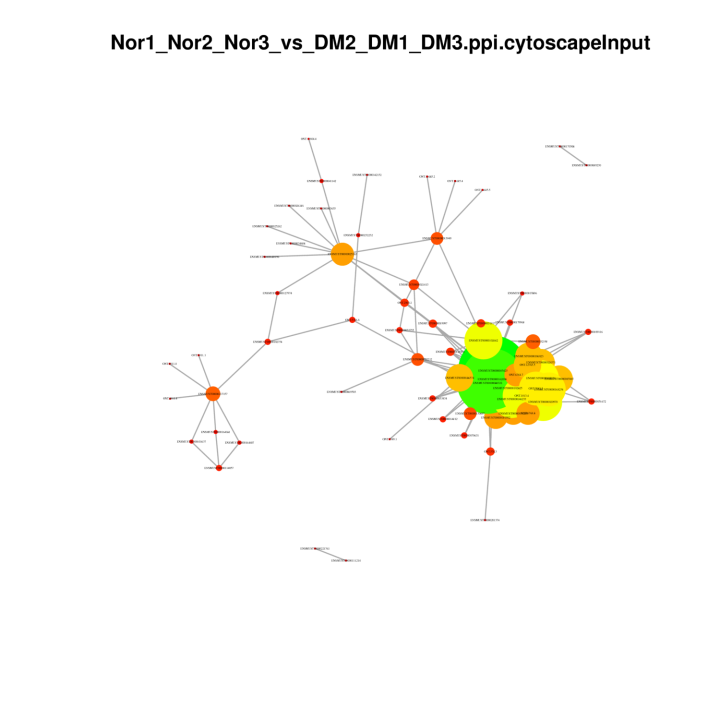
Redes Proteína-Proteína em DEGs
Explore os avanços facilitados pelos serviços completos de sequenciamento de mRNA Nanopore da BMKGene por meio de uma coleção selecionada de publicações.
Gong, B. et al. (2023) 'Ativação epigenética e transcricional da quinase secretora FAM20C como um oncogene no glioma', Journal of Genetics and Genomics, 50(6), pp. doi: 10.1016/J.JGG.2023.01.008.
Ele, Z. et al. (2023) 'O sequenciamento completo do transcriptoma de linfócitos responde ao IFN-γ revela uma resposta imune distorcida por Th1 em linguado (Paralichthys olivaceus)', Fish & Shellfish Immunology, 134, p. 108636. doi: 10.1016/J.FSI.2023.108636.
Mãe, Y. et al. (2023) 'Análise comparativa dos métodos de sequenciamento de RNA PacBio e ONT para identificação do veneno Nemopilema Nomurai', Genomics, 115(6), p. 110709. doi: 10.1016/J.YGENO.2023.110709.
Yu, D. et al. (2023) 'A análise Nano-seq revela diferentes tendências funcionais entre exossomos e microvesículas derivadas de hUMSC', Stem Cell Research and Therapy, 14(1), pp. doi: 10.1186/S13287-023-03491-5/TABELAS/6.