Bibliotecas pré-fabricadas DNBSEQ
Características
●Plataforma:MGI-DNBSEQ-T7
●Modos de sequenciamento:PE150
●Transferência de bibliotecas Illumina paraMGI:permitindo o sequenciamento de grandes volumes de dados a baixo custo.
●Controle de qualidade de bibliotecas antes do sequenciamento.
●Sequenciamento de controle de qualidade e entrega de dados:entrega de relatório de controle de qualidade e dados brutos em formato fastq após demultiplexação e filtragem de leituras Q30.
Vantagens
●Versatilidade dos serviços de sequenciamento:o cliente pode optar por sequenciar por faixa ou quantidade de dados.
●Alta produção de dados:1500 Gb/pista
●Entrega do relatório de CQ de sequenciamento:com métricas de qualidade, precisão dos dados e desempenho geral do projeto de sequenciamento.
●Processo de sequenciamento maduro:com curto tempo de resposta.
●Rigoroso controle de qualidade: implementamos requisitos rigorosos de controle de qualidade para garantir a entrega de resultados consistentemente de alta qualidade.
Requisitos de amostra
| Quantidade de dados (X) | Concentração (qPCR/nM) | Volume | |
| Pista Parcial
| X ≤ 10GB | ≥ 1nM | ≥ 25 μl |
| 10 GB <X ≤ 50 GB | ≥ 2nM | ≥ 25 μl | |
| 50 GB <X ≤ 100 GB | ≥ 3nM | ≥ 25 μl | |
| X > 100 GB | ≥ 4nM | ||
| pista única | Por pista | ≥ 1,5 nM/conjunto de bibliotecas | ≥ 25 μl/conjunto de biblioteca |
Além da concentração e da quantidade total, também é necessário um padrão de pico adequado.
Nota: O sequenciamento de pistas de bibliotecas de baixa diversidade requer pico de PhiX para garantir chamada de base robusta.
Recomendamos enviar bibliotecas pré-agrupadas como amostras. Se você precisar que o BMKGENE execute o pooling de bibliotecas, consulte o
requisitos de biblioteca para sequenciamento parcial de pistas.
Tamanho da biblioteca (mapa de pico)
O pico principal deve estar entre 300-450 pb.
As bibliotecas devem ter um único pico principal, sem contaminação do adaptador e sem dímeros de primer.
Fluxo de trabalho de serviço




Relatório de controle de qualidade da biblioteca
Um relatório sobre a qualidade da biblioteca é fornecido antes do sequenciamento, avaliando a quantidade e a fragmentação da biblioteca.
Relatório de CQ de sequenciamento
Tabela 1. Estatísticas sobre dados de sequenciamento.
| ID da amostra | BMKID | Leituras brutas | Dados brutos (pb) | Leituras limpas (%) | Q20(%) | Q30(%) | GC(%) |
| C_01 | BMK_01 | 22.870.120 | 6.861.036.000 | 96,48 | 99,14 | 94,85 | 36,67 |
| C_02 | BMK_02 | 14.717.867 | 4.415.360.100 | 96,00 | 98,95 | 93,89 | 37.08 |
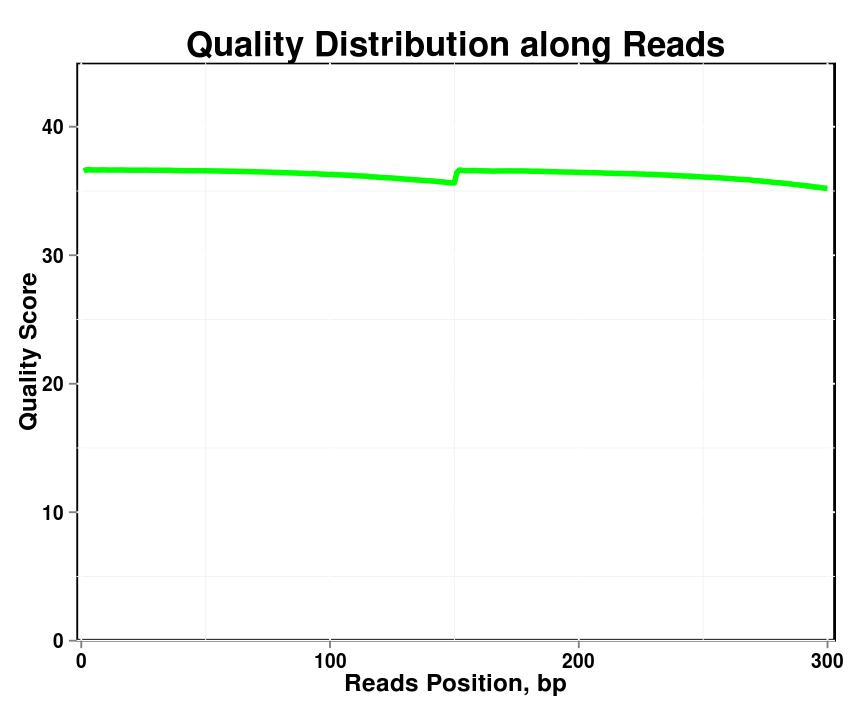
Figura 1. Distribuição de qualidade ao longo das leituras em cada amostra
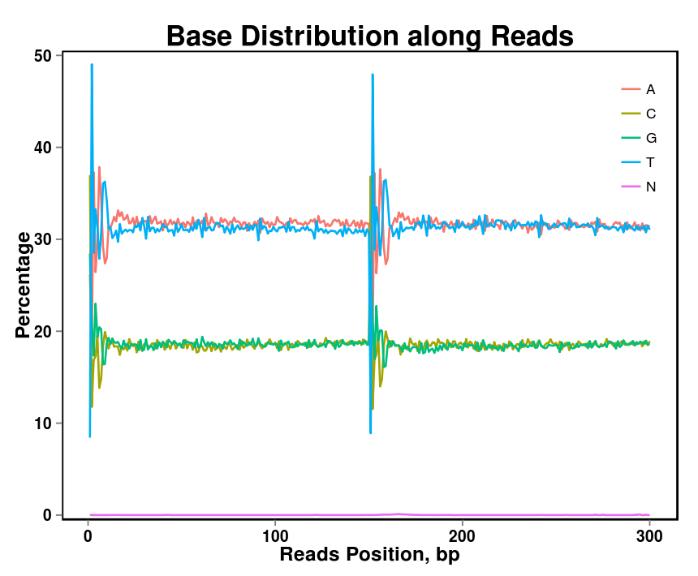
Figura 2. Distribuição de conteúdo base
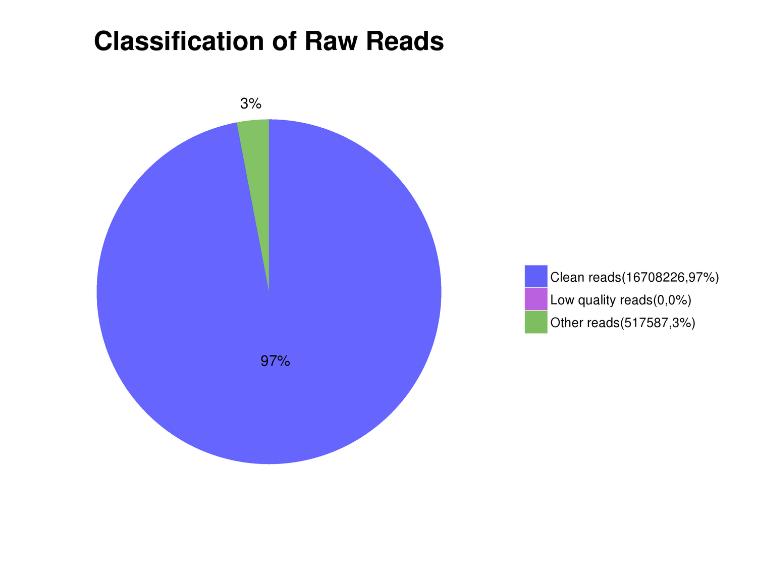
Figura 3. Distribuição do conteúdo lido nos dados de sequenciamento