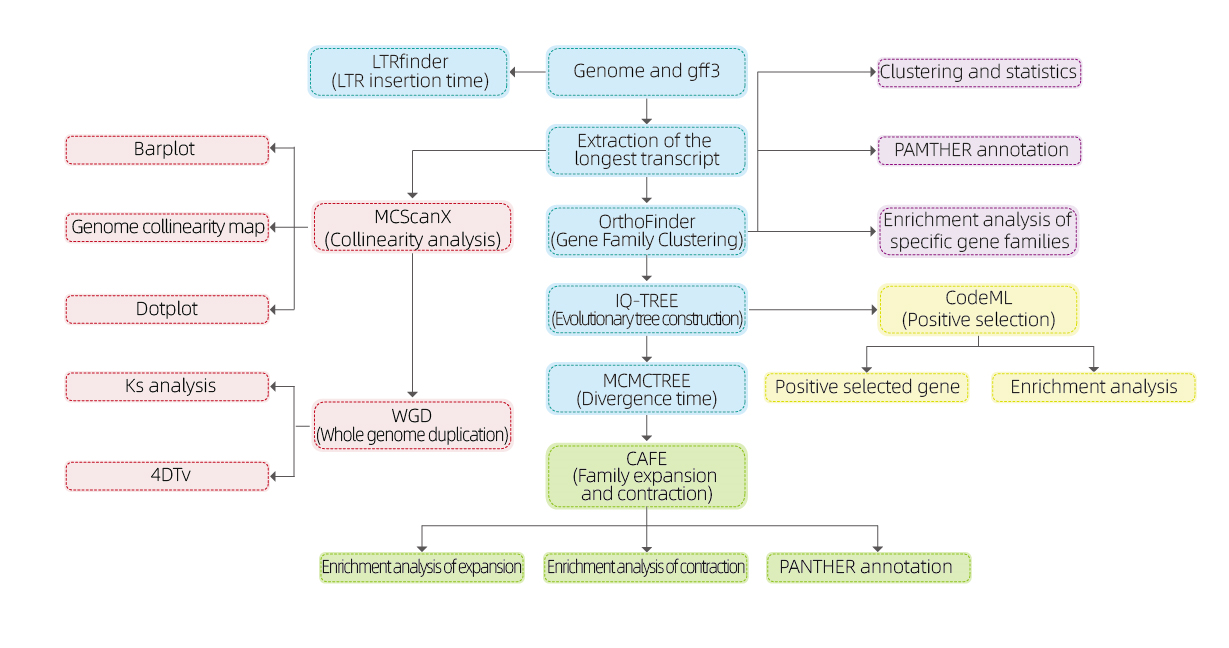
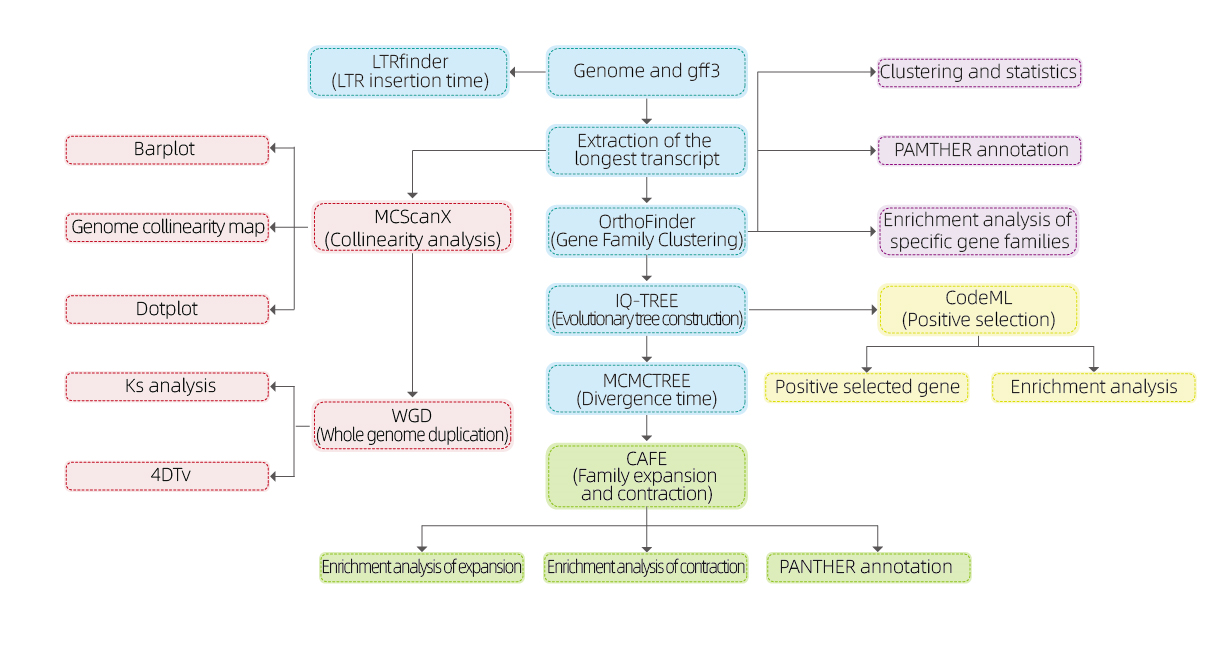
Genômica comparativa
Vantagens de serviço
●Extensos conhecimentos e registros de publicação: Com acumulado, o BMKGENE completou mais de 90 projetos genômicos comparativos, com um fator de impacto cumulativo atingido 900.
●Análise abrangente de bioinformática: O pacote de análises contém as oito análises mais exigidas, fornecendo números prontos para publicação bem projetados e permitindo uma interpretação fácil dos resultados
●Equipe de bioinformática altamente qualificada e ciclo de análise curta: Com ótima experiência em análise de genômica comparativa, a equipe da BMKGENE atende diversas demandas de análise personalizada em um curto tempo de entrega
●Suporte pós-venda:Nosso compromisso se estende além da conclusão do projeto com um período de serviço pós-venda de três meses. Durante esse período, oferecemos acompanhamento do projeto, assistência de solução de problemas e sessões de perguntas e respostas para abordar quaisquer consultas relacionadas aos resultados.
Especificações de serviço
| Tempo estimado de virada | Número de espécies | Análises |
| 30 dias úteis | 6 - 12 | Cluster da família de genes Expansão e contração familiares de genes Construção de árvores filogenéticas Estimativa de tempo de divergência (calibração fóssil necessária) Tempo de inserção LTR (para plantas) Duplicação de genoma inteiro (para plantas) Pressão seletiva Análise de Synteny |
Análises de bioinformática
● Família de genes
● filogenética
● Tempo de divergência
● Pressão seletiva
● Análise de Synteny
Requisitos de amostra e entrega
Requisitos de amostra:
Tecido ou DNA para sequenciamento e montagem do genoma
Para tecido
| Espécies | Tecido | Enquete | Pacbio CCS |
| Animal | Tecido visceral | 0,5 ~ 1 g | ≥ 3,5 g |
| Tecido muscular | |||
| ≥ 5,0 g | |||
| ≥ 5,0 ml | |||
| Sangue de mamíferos | |||
| ≥ 0,5 ml | |||
| Blood de aves/peixes | |||
| Plantar | Folha fresca | 1 ~ 2 g | ≥ 5,0 g |
| PETAL/STEM | 1 ~ 2 g | ≥ 10,0 g | |
| Raiz/semente | 1 ~ 2 g | ≥ 20,0 g | |
| Células | Célula cultivada | - | ≥ 1 x 108 |
Dados
Arquivos de sequência do genoma (.fastA) e arquivos de anotação (.gff3) de espécies intimamente relacionadas
Fluxo de trabalho de serviço

Projeto de experimento

Entrega de amostra

Construção da biblioteca

Sequenciamento

Análise de dados

Serviços pós-venda
*Os resultados da demonstração mostrados aqui são todos de genomas publicados com tecnologias de biomarcadores
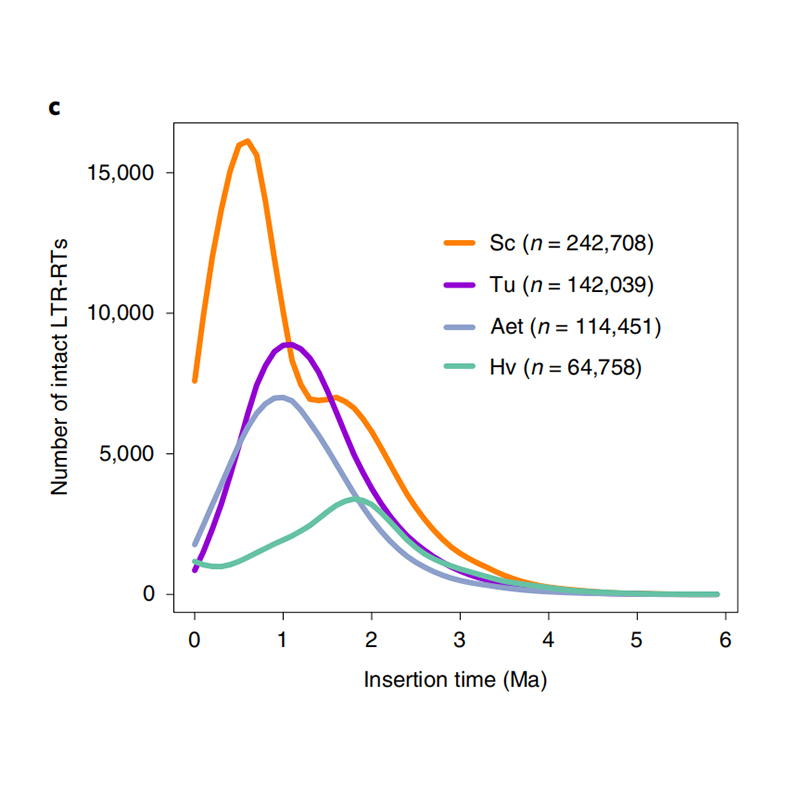
1.LTR INSERIR TEMPO ESTIMAÇÃO: A figura mostrou uma distribuição bimodal única nos tempos de inserção LTR-RTS no genoma do centeio de Weining, em comparação com outras espécies. O pico mais recente apareceu cerca de 0,5 milhão de anos atrás.
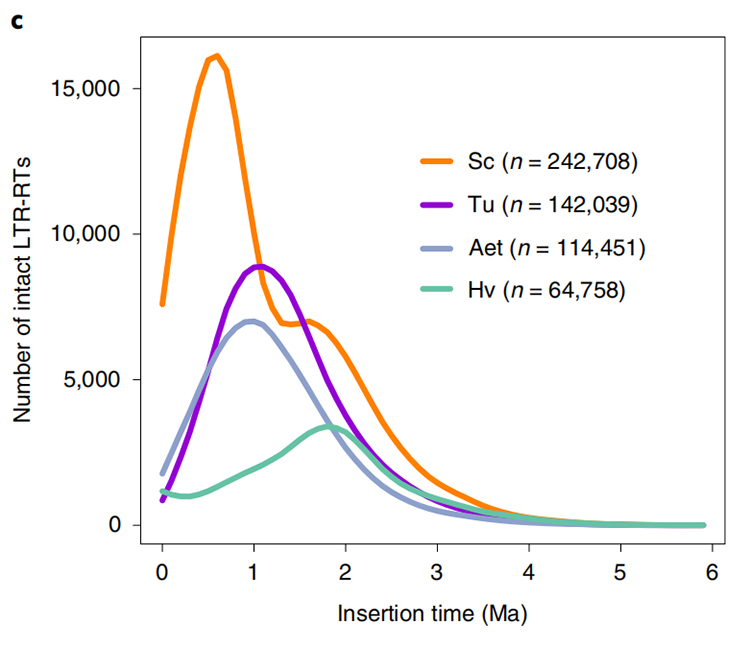
Li Guang et al.,Genética da natureza, 2021
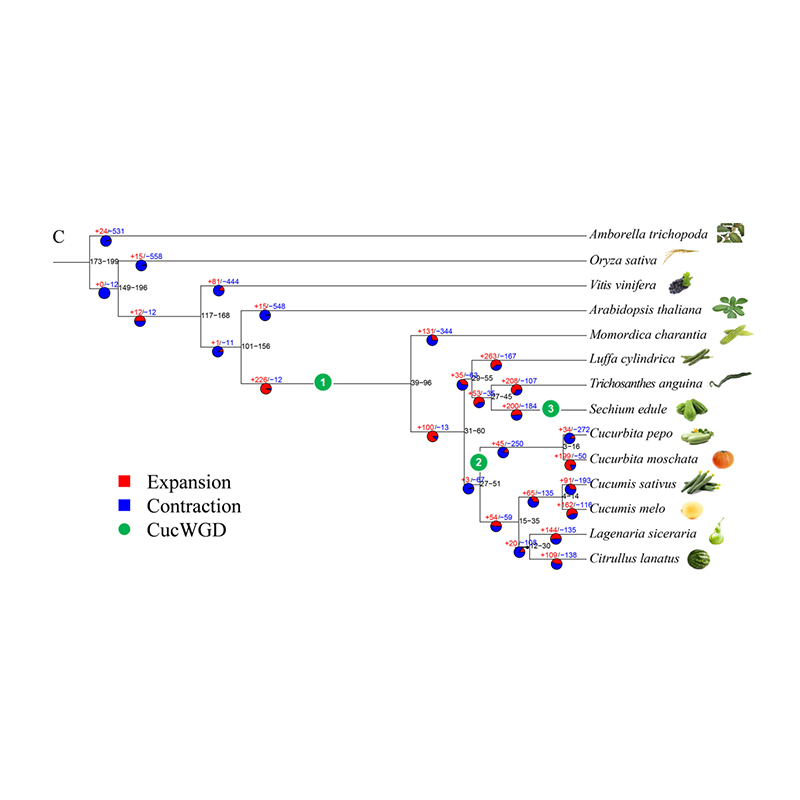
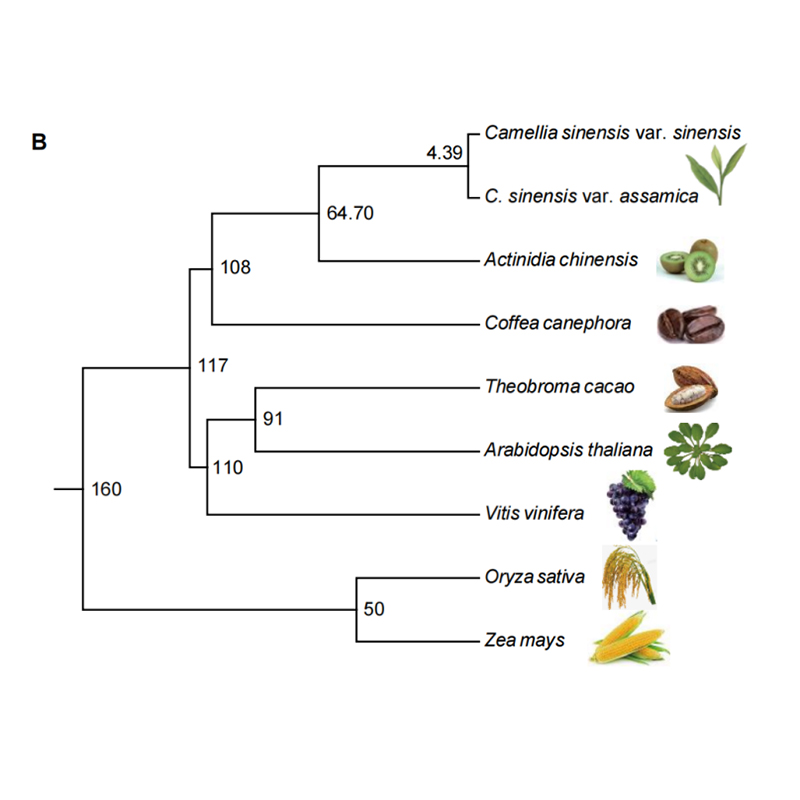
2.filogenia e análise da família de genes em chayote (Sechium EDULE): Ao analisar o chayote e as outras 13 espécies relacionadas na família de genes, verificou -se que o chayote estava mais intimamente relacionado à cabaça de cobra (Trichosanthes Angetina). O chayote derivou da cabaça de cobra em torno de 27-45 mya e duplicação de genoma inteiro (WGD) foi observada em chayote em 25 ± 4 mya, que é o terceiro evento da WGD em Cucuibitaceae.
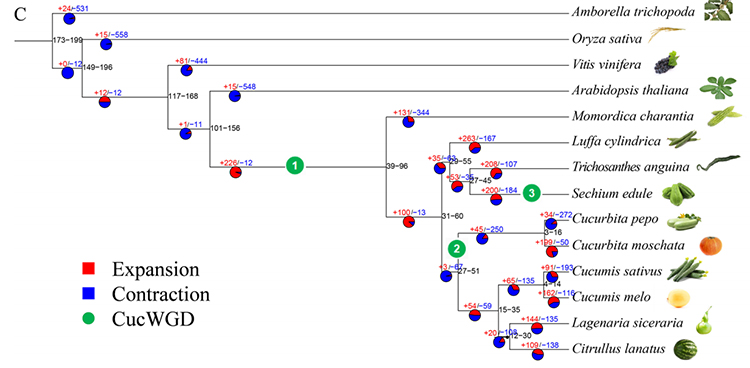
Fu A et al.,Pesquisa de horticultura, 2021
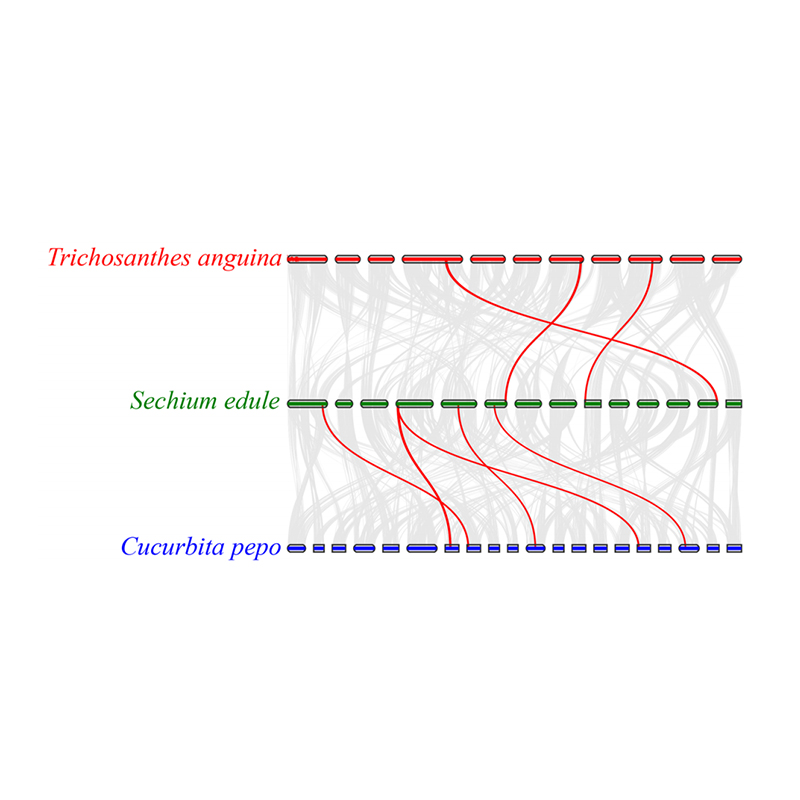
3. Análise Synteny: Alguns genes relacionados aos fitohormônios no desenvolvimento de frutas foram encontrados em chayote, cabaça de cobra e abóbora. A correlação entre o chayote e a abóbora é um pouco mais alta que a cabaça de chayote e cobra.
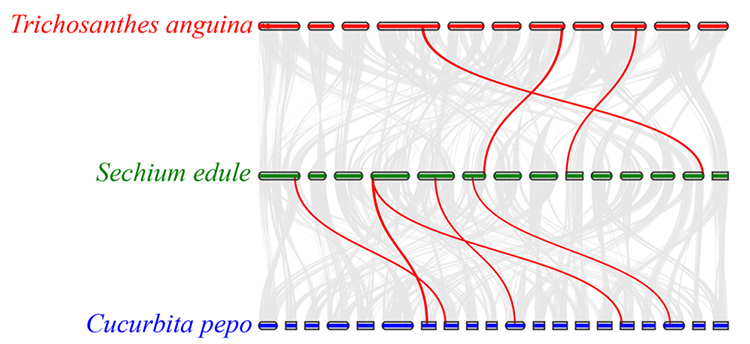
Fu A et al.,Pesquisa de horticultura, 2021
4. Análise da família GENE: Enriquecimento de KEGG na expansão e contração da família de genes nos genomas de G.Thurberi e G.Davidsonii demonstraram que os genes relacionados a biossíntese de esteróides e biossíntese de brassinosteróides foram expandidos.
Yang Z et al.,BMC Biologia, 2021
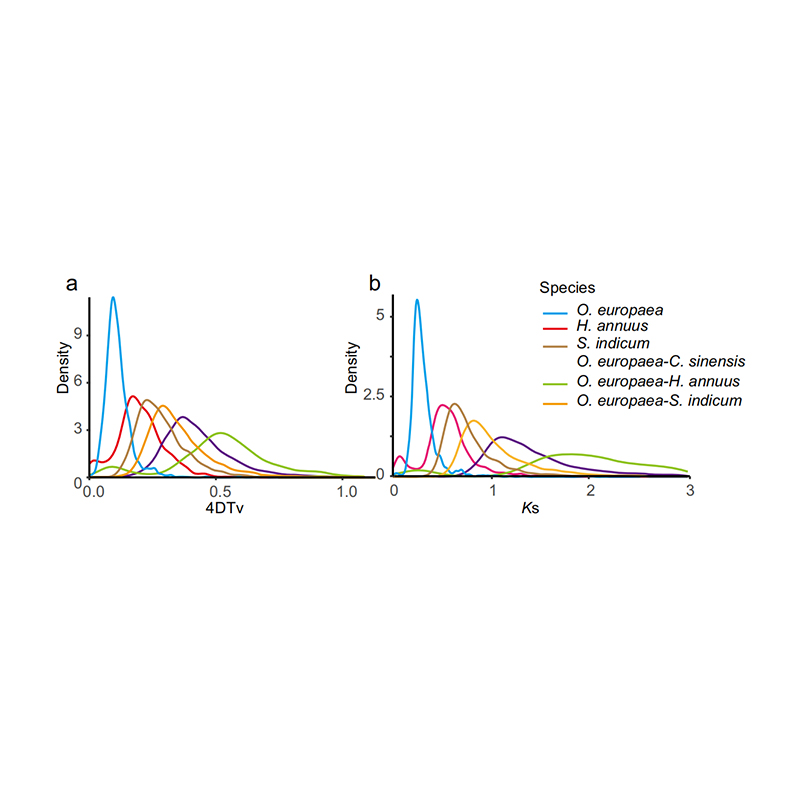
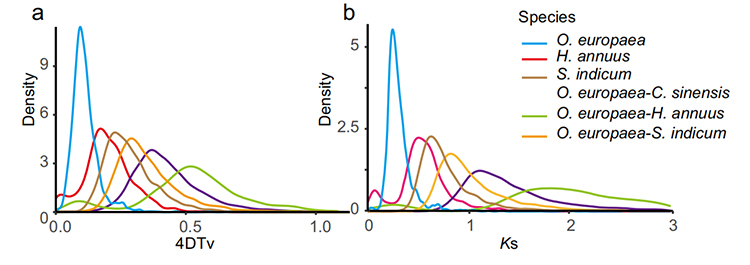
5. Análise de duplicação do genoma do Whole: Análise de distribuição 4DTV e KS mostrou um evento de duplicação de genoma inteiro. Picos de intraespécies mostraram eventos de duplicação. Picos de interespécies mostraram eventos de especiação. A análise indicou que, em comparação com as outras três espécies intimamente relacionadas, O. Europaea passou por uma duplicação de genes em larga escala mais recentemente.

Rao G et al.,Pesquisa de horticultura, 2021
Caso BMK
Rose sem espicada: idéias genômicas ligadas à adaptação de umidade
Publicado: National Science Review, 2021
Estratégia de sequenciamento:
'Basye'sSem espinhos'(R.Wichurainan) Genoma:
Aprox. 93 x Pacbio + aprox. 90 x nanopore + 267 x ilumina
Resultados -chave
1. O genoma da alta qualidade de R.Wichuraiana foi construído usando técnicas de sequenciamento de leitura longa, que produzem uma montagem de 530,07 MB (o tamanho estimado do genoma foi de aproximadamente 525,9 Mb por citometria de fluxo e 525,5 por pesquisa do genoma ; A heterozigidade foi de 1,03%). A pontuação estimada em Busco foi de 93,9%. Comparando com “Old Blush” (haploob), a qualidade e a integridade desse genoma foram confirmadas pela precisão da base única e do índice de montagem LTR (LAI = 20,03). O genoma de R.wichuraiana contém 32.674 genes de codificação de proteínas.
2. Multi-ômicos Análise conjunta, consistindo em genômica comparativa, transcriptômica, análise QTL da população genética, revelou a especiação crucial entre R. Wichuraiana e Rosa Chinensis. Além disso, é provável que a variação de expressão de genes relacionados no QTL esteja associada ao padrão de bico -tronco.

A genômica comparativa de Anaysis entre Basye, sem espinhos e Rosa chinensis, incluindo análise de sinteny, agrupamento de família de genes, análise de expansão e contração, revelou um grande número de variações, relacionadas a características cruciais em rosas. A expansão única na família de genes NAC e FAR1/FRS provavelmente estava associada à resistência à mancha preta.
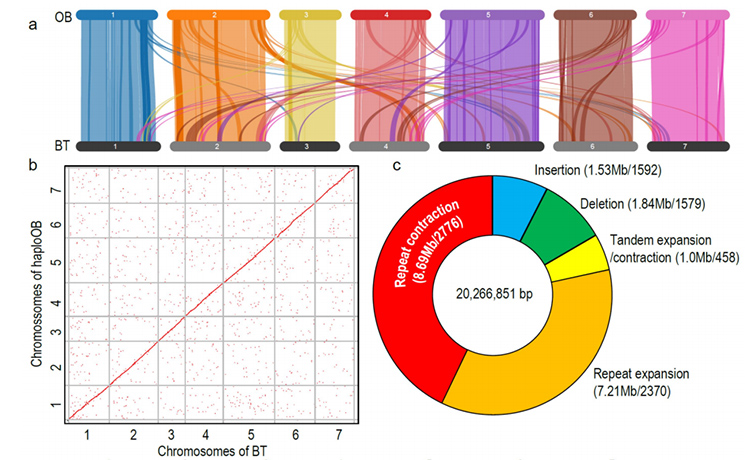

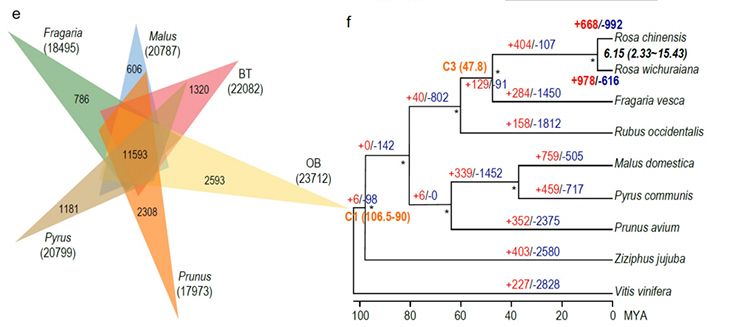
Análise genômica comparativa entre os genomas de BT e haploob.
Zhong, M., et al. “Rose sem espicada: idéias genômicas ligadas à adaptação para umidade”National Science Review, 2021;, NWAB092.