Sequenciamento de Amplicon 16S/18S/ITS-PacBio
Recursos de serviço
● Plataforma de sequenciamento: PacBio Revio
● Modo de sequenciamento: CCS (leituras HiFi)
● Amplificação da região alvo seguida de ligação em tandem de amplicons antes da preparação da biblioteca de sinos HiFi SMRT
Vantagens do serviço
●Maior resolução taxonômica: Tsequenciamento de amplicon curto han,permitindo taxas de classificação OTU mais altas em nível de espécie.
●Chamada de base altamente precisa: Sequenciamento do modo PacBio CCS (leituras HiFi).
●Livre de isolamento: Identificação rápida da composição microbiana em amostras ambientais.
●Amplamente aplicável: Diversos estudos de comunidades microbianas.
●Análise bioinformática abrangente: O mais recente pacote QIIME2 (visão quantitativa da ecologia microbiana) com diversas análises em termos de banco de dados, anotação, OTU/ASV.
●Ampla experiência: Com milhares de projetos de sequenciamento de amplicons realizados anualmente, a BMKGENE traz mais de uma década de experiência, uma equipe de análise altamente qualificada, conteúdo abrangente e excelente suporte pós-venda.
Especificações de serviço
| Biblioteca | Estratégia de sequenciamento | Dados recomendados |
| Amplicão | PacBio Revisão | 30/10/50 mil tags (CCS) |
Requisitos de amostra
| Concentração (ng/µL) | Quantidade total (µg) | Volume (µL) |
| ≥5 | ≥0,3 | ≥20 |
Entrega de amostra recomendada
Congele as amostras em nitrogênio líquido por 3-4 horas e armazene em nitrogênio líquido ou -80 graus para reserva de longo prazo. É necessário enviar amostras com gelo seco.
Fluxo de trabalho de serviço

Entrega de amostra

Construção de biblioteca

Sequenciamento

Análise de dados

Serviços pós-venda
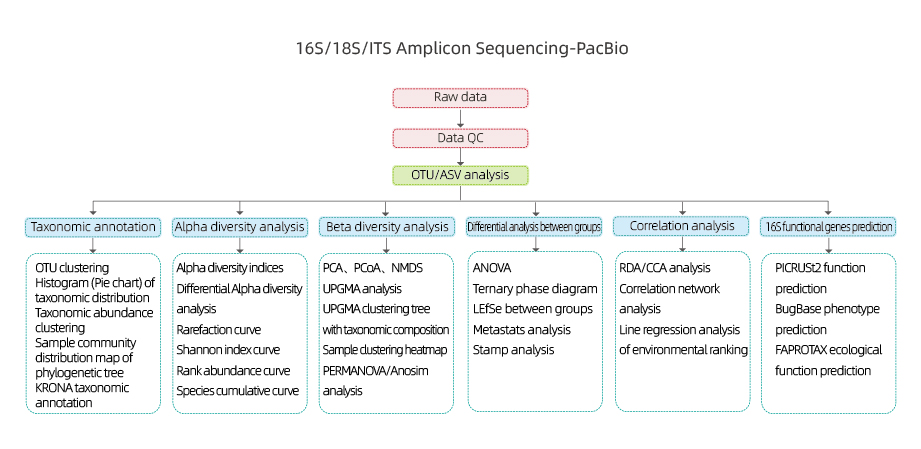
Inclui a seguinte análise:
●Controle de qualidade de dados brutos
● Clustering/eliminação de ruído de OTU (ASV)
● Anotação OTU
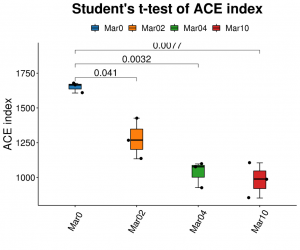
●Análise de diversidade alfa: múltiplos índices, incluindo Shannon, Simpson e ACE.
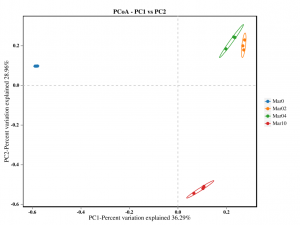
●Análise de diversidade beta
●Análise intergrupo
●Análise de correlação: entre fatores ambientais e composição e diversidade OUT
●Predição do gene funcional 16S
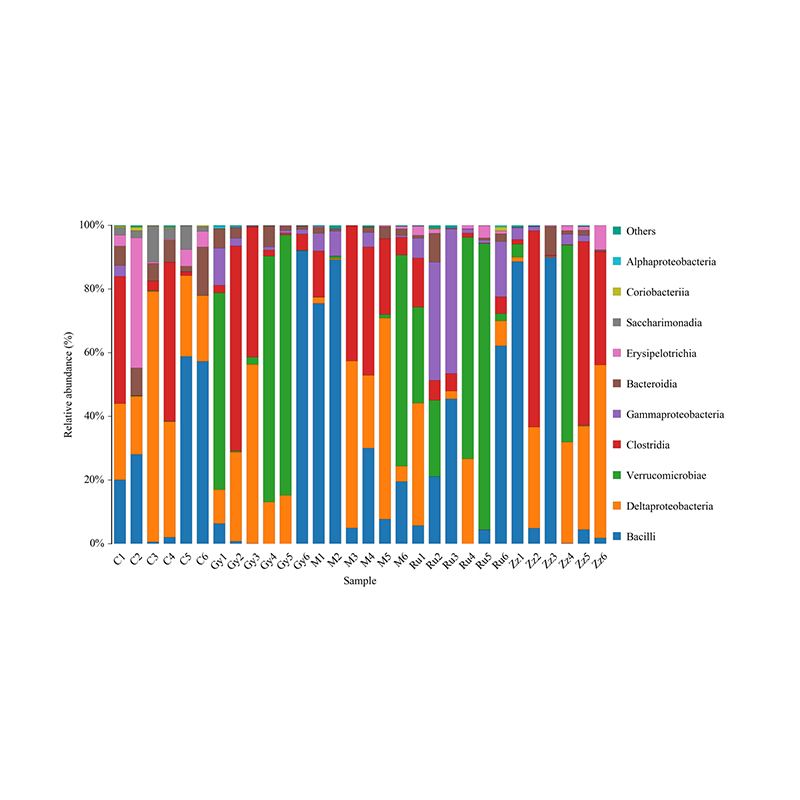
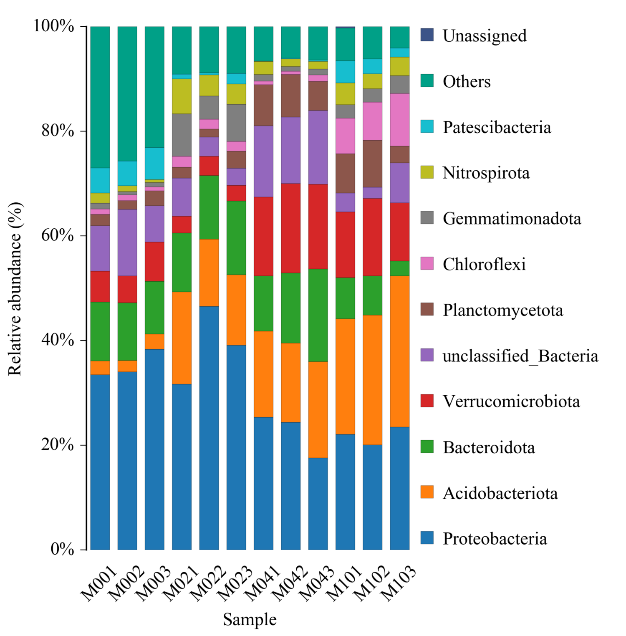
Histograma de distribuição taxonômica
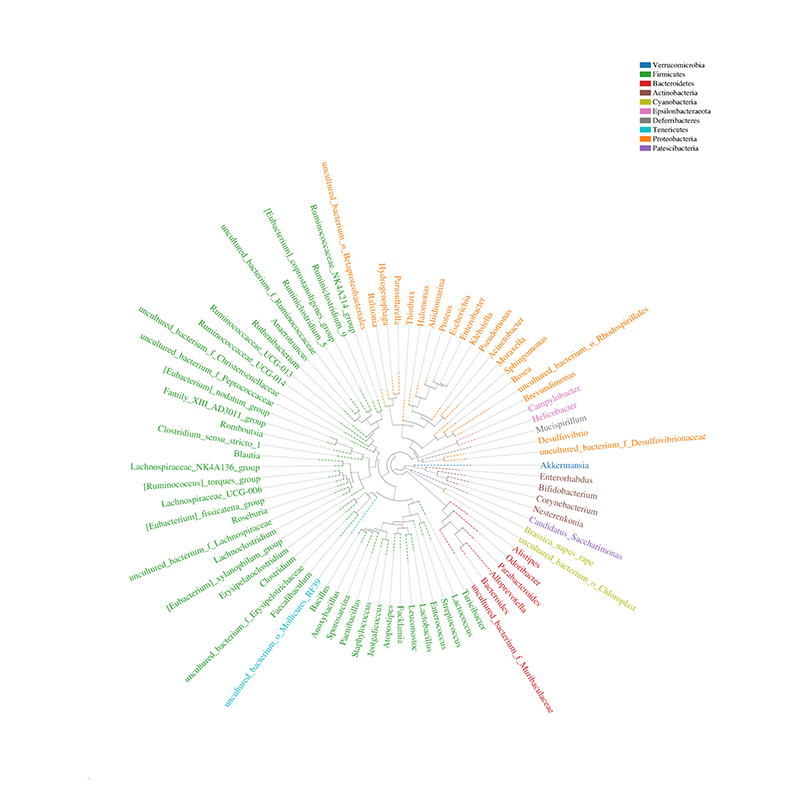
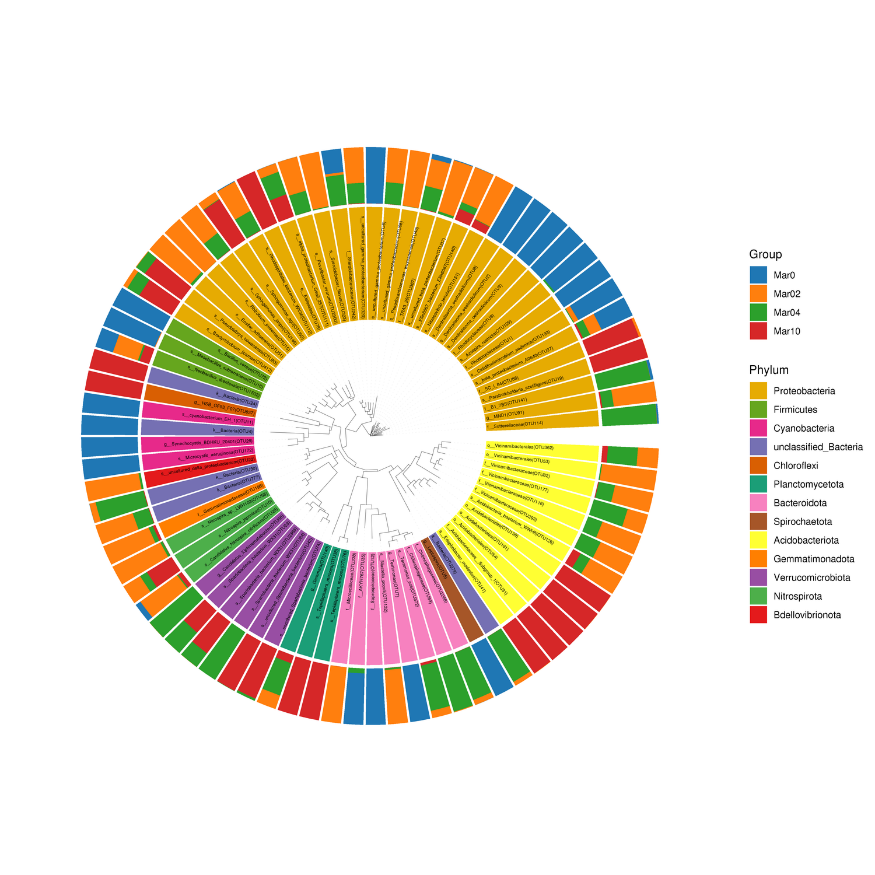
Árvore filogenética de distribuição comunitária
Análise de diversidade alfa: ACE
Análise de diversidade beta: PCoA
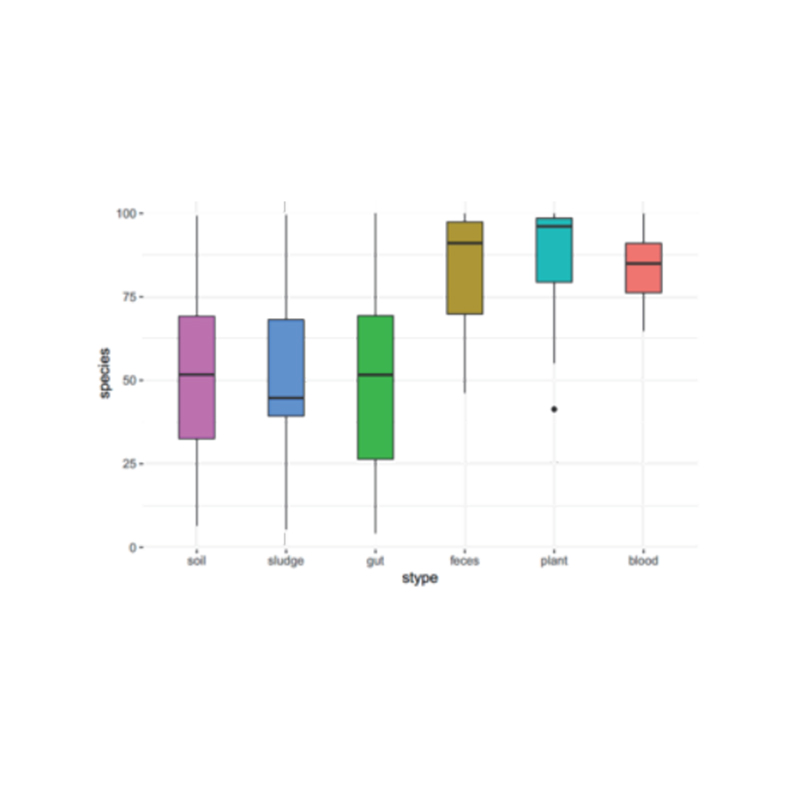
Análise intergrupo: ANOVA
Explore os avanços facilitados pelos serviços de sequenciamento de amplicons da BMKGene com PacBio por meio de uma coleção selecionada de publicações.
Gao, X. e Wang, H. (2023) 'Análise comparativa de perfis e funções bacterianas do rúmen durante a adaptação a diferentes fenologia (Regreen vs. Grassy) em ovelhas Merino alpinas com dois estágios de crescimento em um prado alpino', Fermentação, 9 ( 1), pág. 16. doi: 10.3390/FERMENTAÇÃO9010016/S1.
Li, S. et al. (2023) 'Capturando a matéria escura microbiana em solos desérticos usando metagenômica baseada em culturômica e análise de alta resolução', npj Biofilms and Microbiomes 2023 9:1, 9(1), pp. doi: 10.1038/s41522-023-00439-8.
Mu, L. et al. (2022) 'Efeitos dos sais de ácidos graxos nas características de fermentação, diversidade bacteriana e estabilidade aeróbica de silagem mista preparada com alfafa, palha de arroz e farelo de trigo', Journal of the Science of Food and Agriculture, 102(4), pp. 1487. doi: 10.1002/JSFA.11482.
Yang, J. et al. (2023) 'A interação entre biomarcadores de estresse oxidativo e microbiota intestinal nos efeitos antioxidantes de extratos de Sonchus brachyotus DC. em Estresse Oxidativo Intestinal Induzido por Oxazolona em Zebrafish Adulto', Antioxidantes 2023, Vol. 12, página 192, 12(1), p. 192.doi:10.3390/ANTIOX12010192.