Całe sekwencjonowanie transkryptomu - Illumina
Cechy
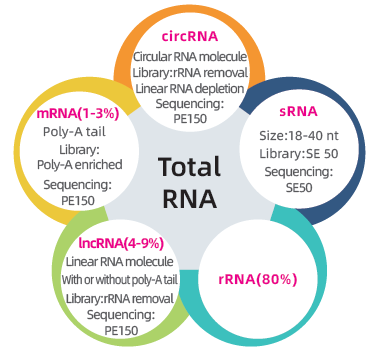
● Podwójna biblioteka w celu sekwencjonowania kompletnego transkryptomu: RRNA zubożenie, a następnie przygotowanie biblioteki PE150 i wybór rozmiaru, a następnie przygotowanie biblioteki SE50
● Pełna analiza bioinformatyczna mRNA, LNCRNA, CIRCRNA i miRNA w oddzielnych raportach bioinformatycznych
● Wspólna analiza wszystkich ekspresji RNA w połączonym raporcie, w tym analiza sieci CERNA.
Zalety serwisowe
●Dogłębna analiza sieci regulacyjnych: Analiza sieci CERNA jest włączona przez wspólne sekwencjonowanie mRNA, LNCRNA, CIRCRNA i miRNA oraz przez wyczerpujący bioinformatyczny przepływ pracy.
●Kompleksowa adnotacja: Używamy wielu baz danych do funkcjonalnego adnotowania genów o różnej ekspresji (DEG) i przeprowadzania odpowiedniej analizy wzbogacania, zapewniając wgląd w procesy komórkowe i molekularne leżące u podstaw odpowiedzi transkryptomu.
●Rozległa wiedza specjalistyczna: Dzięki osiągnięciu pomyślnego zamykania ponad 2100 projektów całego transkryptomu w różnych dziedzinach badań, nasz zespół wnosi bogate doświadczenie w każdym projekcie.
●Rygorystyczna kontrola jakości: Wdrażamy podstawowe punkty sterowania na wszystkich etapach, od przygotowania próbek i biblioteki po sekwencjonowanie i bioinformatykę. To skrupulatne monitorowanie zapewnia dostarczanie konsekwentnie wysokiej jakości wyników.
●Wsparcie po sprzedaży: Nasze zobowiązanie wykracza poza zakończenie projektu z 3-miesięcznym okresem usług po sprzedaży. W tym czasie oferujemy obserwację projektu, pomoc w rozwiązywaniu problemów oraz sesje pytań i odpowiedzi
Przykładowe wymagania i dostawa
| Biblioteka | Strategia sekwencjonowania | Zalecane dane | Kontrola jakości |
| RRNA wyczerpano | Illumina PE150 | 16 GB | Q30 ≥85% |
| Wybrany rozmiar | Illumina SE50 | Odczyty 10-20 m |
Przykładowe wymagania:
Nukleotydy:
| Conc. (Ng/μl) | Ilość (μg) | Czystość | Uczciwość |
| ≥ 80 | ≥ 1,6 | OD260/280 = 1,7-2,5 OD260/230 = 0,5-2,5 Ograniczone lub brak zanieczyszczenia białka lub DNA pokazanego na żelu. | Rin ≥6,0 5.0 ≥28S/18S ≥1,0; Ograniczona lub brak wyjściowej wzniesienia |
Zalecana dostawa próbki
Pojemnik: 2 ml rurki wirówki (folia cyny nie jest zalecana)
Etykietowanie przykładowe: grupa+replikuj EG A1, A2, A3; B1, B2, B3.
Wysyłka:
1. Suche ice: Próbki należy pakować w torby i zakopane w suchym lodzie.
2. RnAstable Rurki: Próbki RNA można wysuszyć w rurce stabilizacyjnej RNA (np. Rnastable®) i wysyłania w temperaturze pokojowej.
Przepływ pracy usług

Projekt eksperymentu

Dostawa próbki

Ekstrakcja RNA

Konstrukcja biblioteki

Sekwencjonowanie

Analiza danych

Usługi po sprzedaży
Bioinformatyka
Przegląd ekspresji RNA
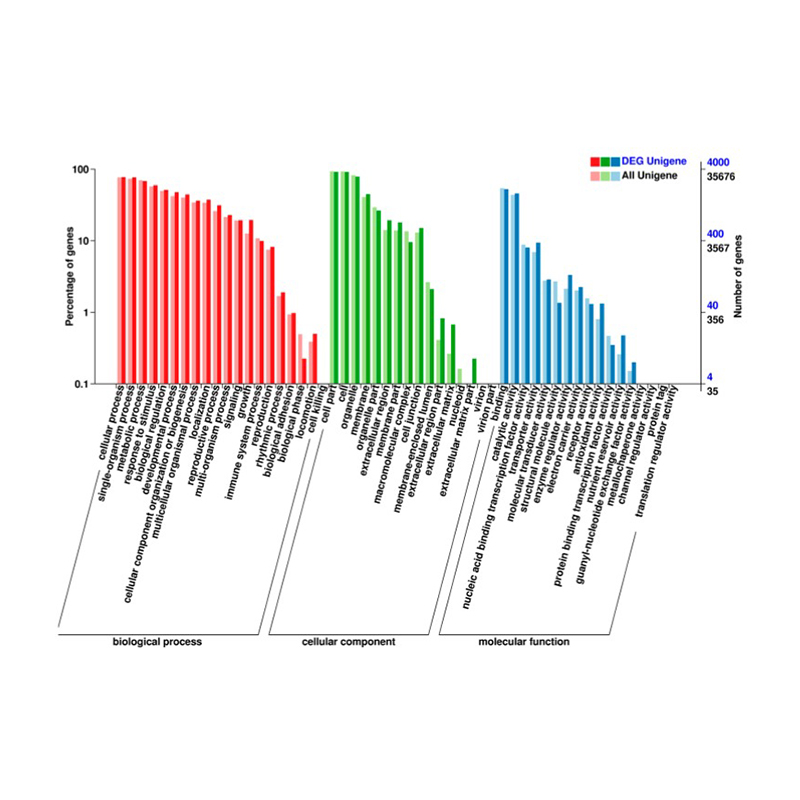
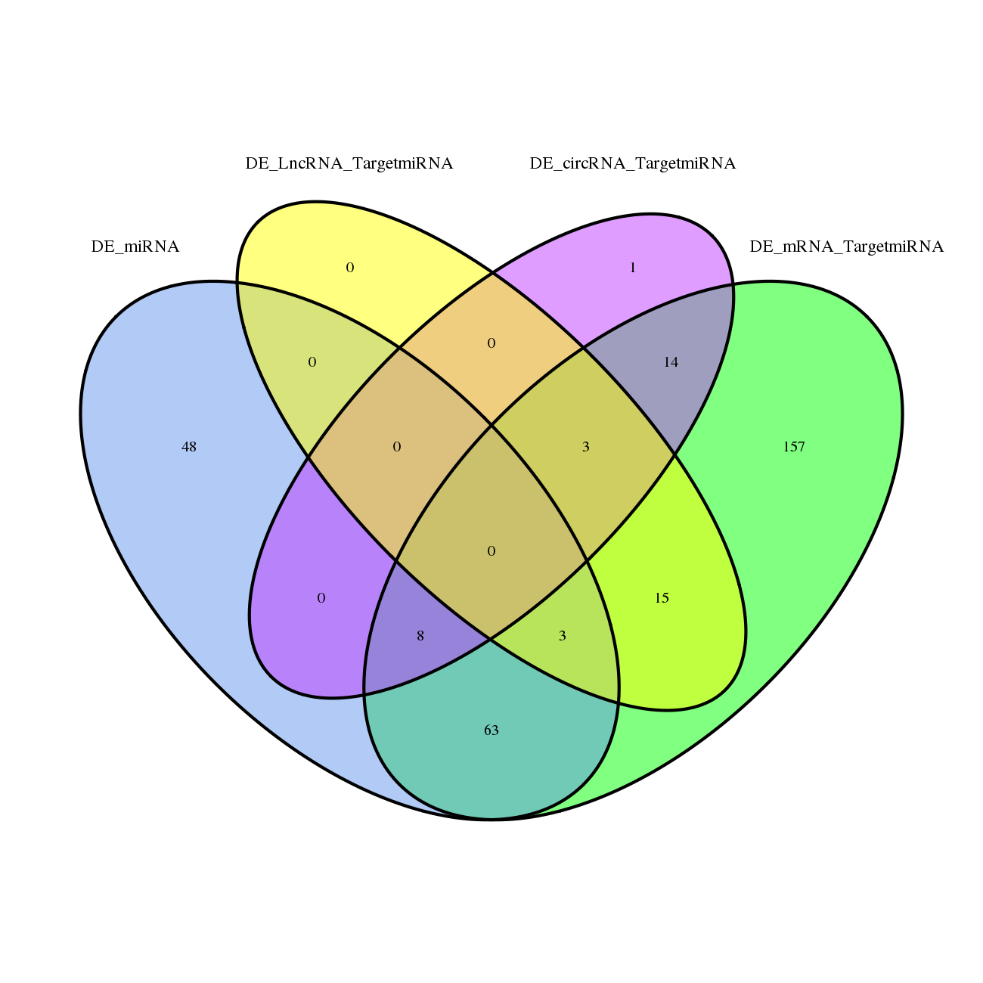
Geny wyrażane w różny sposób
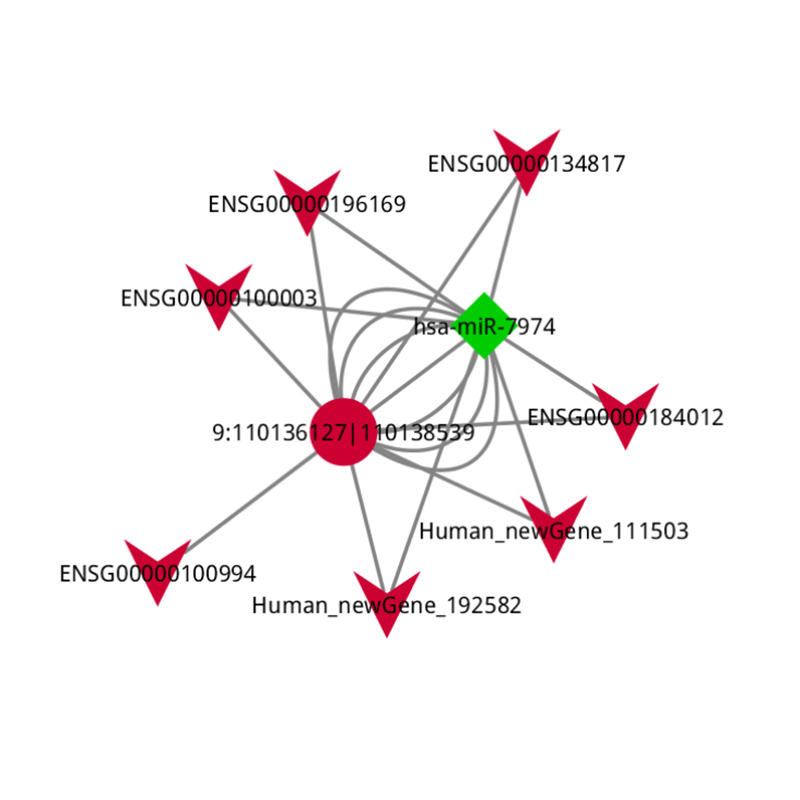
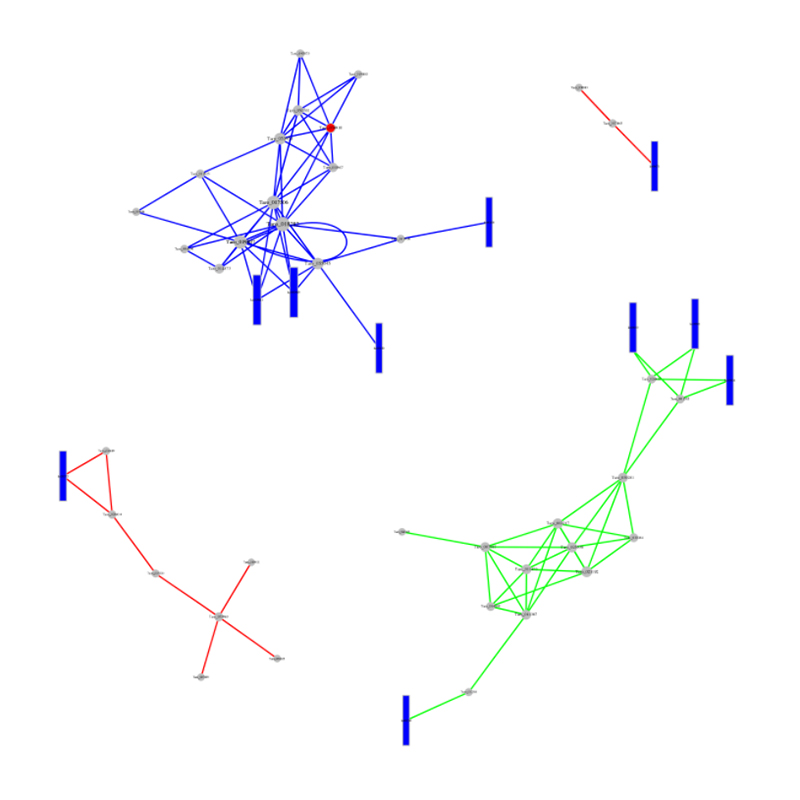
Analiza CERNA
Zbadaj postępy badawcze ułatwione przez usługi sekwencjonowania całego transkryptomu BMKGENE za pośrednictwem wyselekcjonowanego zbioru publikacji.
Dai, Y. i in. (2022) „Kompleksowe profile ekspresji mRNA, lncRNA i miRNA w chorobie Kashin-Beck zidentyfikowanej przez sekwencjonowanie RNA”, Molecular Omics, 18 (2), s. 154–166. DOI: 10.1039/D1MO00370D.
Liu, N. Nan i in. (2022) „Analiza transkryptomów pełnej długości oporności na zimno API Cerana w górach Changbai w okresie zimowania.”, Gene, 830, s. 146503–146503. doi: 10.1016/j.gene.2022.146503.
Wang, XJ i in. (2022) „Oparta na integracji wielorakowej priorytetyzowanie konkurencyjnych endogennych sieci regulacji RNA w małych komórkach raka płuc: cechy molekularne i kandydatów na leki”, granice onkologii, 12, str. 904865. Doi: 10.3389/fonc.2022.904865/bibtex.
Xu, P. i in. (2022) „Zintegrowana analiza profili ekspresji lncRNA/circRNA-miRNA-MRNA ujawnia nowe spostrzeżenia w potencjalnych mechanizmach w odpowiedzi na nicienie z korzeni w orzechach ziemnych”, BMC Genomics, 23 (1), s. 1–12. doi: 10.1186/S12864-022-08470-3/Figures/7.
Yan, Z. i in. (2022) „Sekwencjonowanie RNA całego transskrypcji podkreśla mechanizmy molekularne związane z utrzymaniem jakości postharvest na brokułach przez promieniowanie czerwonych LED”, Postharvest Biology and Technology, 188, s. 1. 111878. Doi: 10.1016/j.postharvbio.2022.111878.