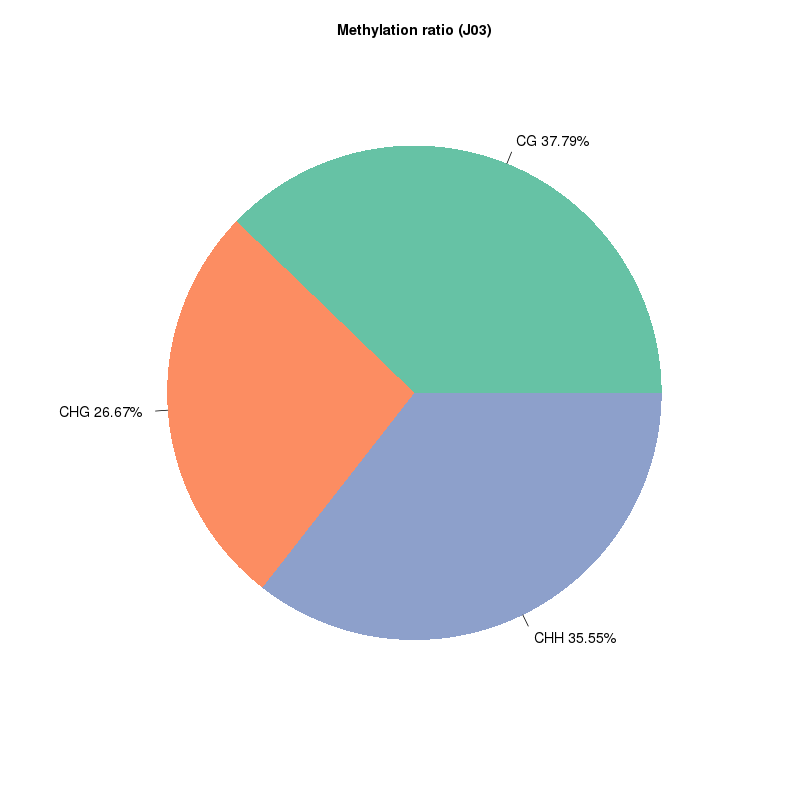Sekwencjonowanie wodorosiarczynem całego genomu (WGBS)
Funkcje usługi
● Wymaga genomu referencyjnego.
● Dodano DNA lambda w celu monitorowania wydajności konwersji wodorosiarczynem.
● Sekwencjonowanie na Illumina NovaSeq.
Zalety serwisu
●Złoty standard badań nad metylacją DNA: Ta dojrzała technologia przetwarzania konwersji metylacji charakteryzuje się wysoką dokładnością i dobrą powtarzalnością.
●Szeroki zasięg i rozdzielczość jednobazowa:wykrywanie miejsc metylacji na poziomie całego genomu.
●Kompletna platforma:zapewniają doskonałą obsługę w jednym miejscu, od przetwarzania próbek, budowy bibliotek, sekwencjonowania po analizę bioinformatyczną.
●Rozległa wiedza specjalistyczna: dzięki pomyślnie zakończonym projektom sekwencjonowania WGBS dla różnorodnych gatunków, BMKGENE wnosi ponad dziesięcioletnie doświadczenie, wysoko wykwalifikowany zespół analityczny, obszerną treść i doskonałe wsparcie posprzedażowe.
●Możliwość dołączenia do analizy transkryptomicznej: umożliwiając zintegrowaną analizę WGBS z innymi danymi omicznym, takimi jak RNA-seq.
Przykładowe specyfikacje
| Biblioteka | Strategia sekwencjonowania | Zalecane wyjście danych | Kontrola jakości |
| Obróbka wodorosiarczynem | Illumina PE150 | Głębokość 30x | Q30 ≥ 85% Konwersja wodorosiarczynu > 99% |
Przykładowe wymagania
| Stężenie (ng/µL) | Całkowita ilość (µg) | Dodatkowe wymagania | |
| Genomowe DNA | ≥ 5 | ≥ 400 ng | Ograniczona degradacja lub zanieczyszczenie |
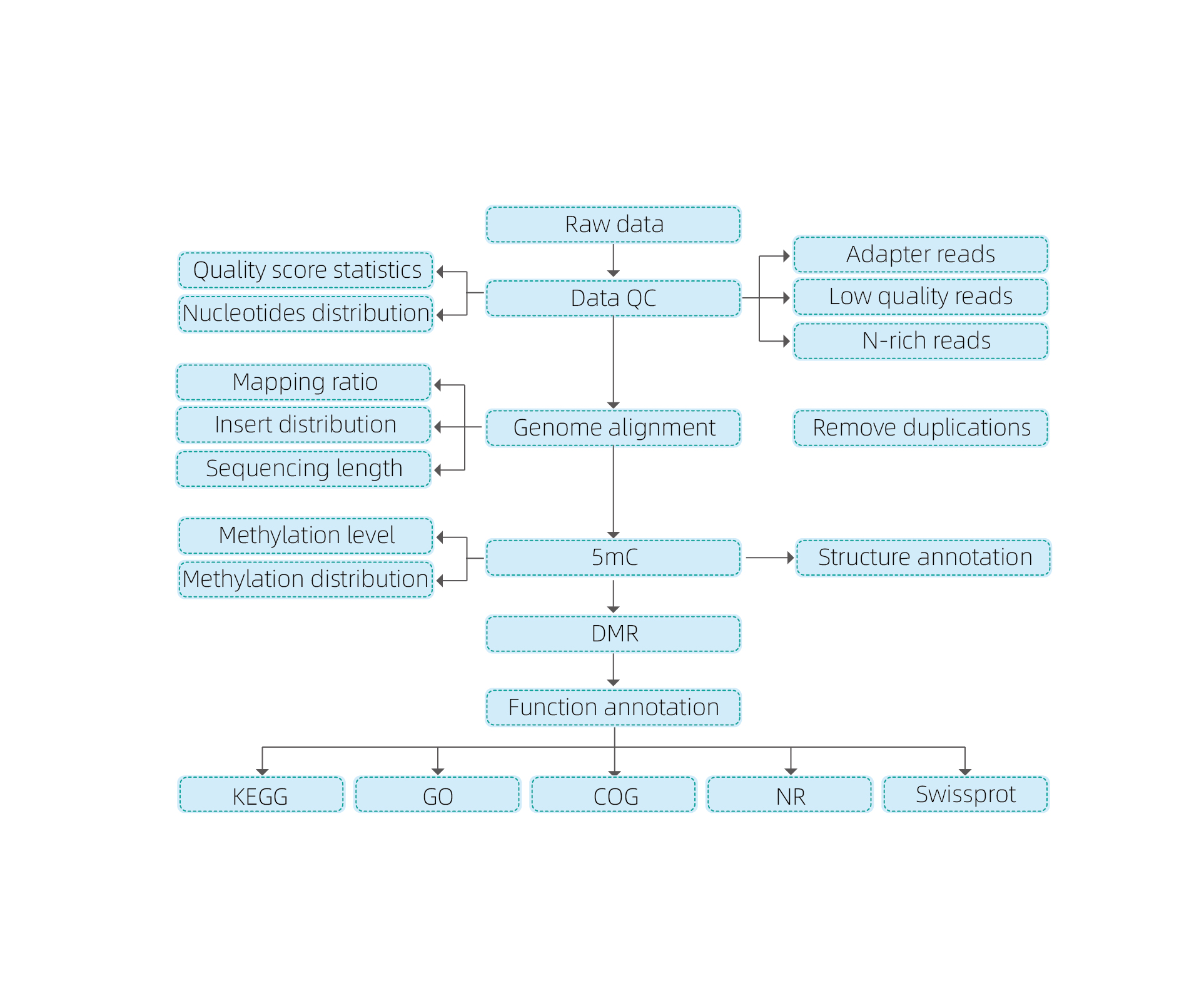
Przebieg prac serwisowych

Dostawa próbek

Ekstrakcja DNA

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Dostarczanie danych
Zawiera następującą analizę:
● Kontrola jakości surowego sekwencjonowania;
● Mapowanie genomu referencyjnego;
● Wykrywanie zasad metylowanych 5mC;
● Analiza rozkładu metylacji i adnotacja;
● Analiza regionów różnicowo metylowanych (DMR);
● Adnotacja funkcjonalna genów związanych z DMR.
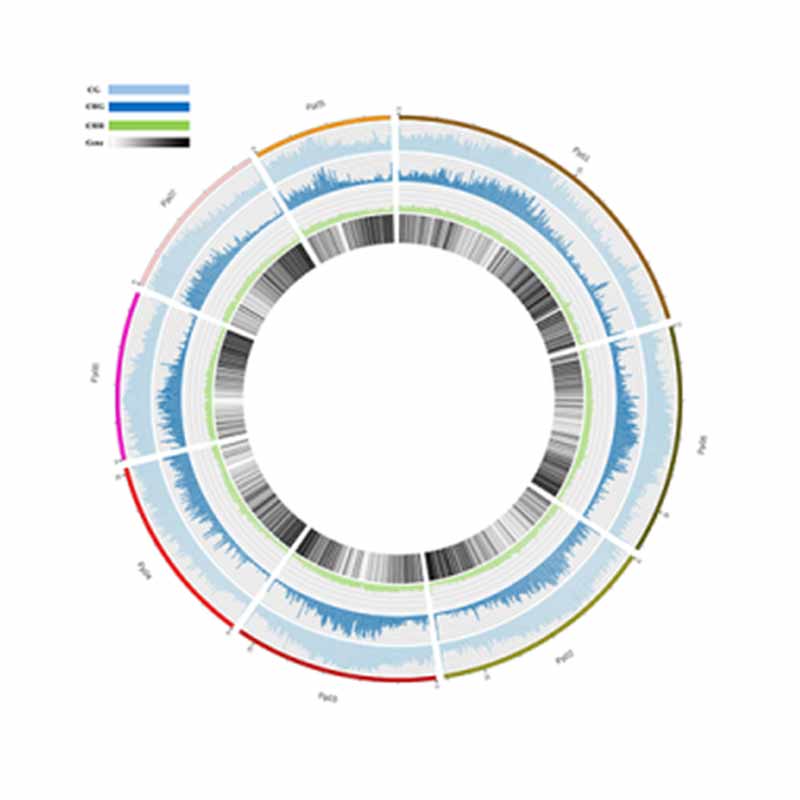
Detekcja metylacji 5mC: rodzaje miejsc metylowanych
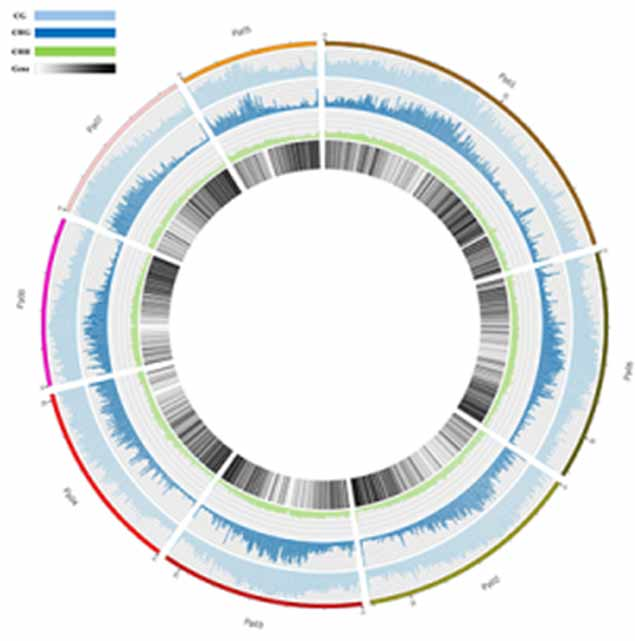
Mapa metylacji. Rozkład metylacji 5mC w całym genomie
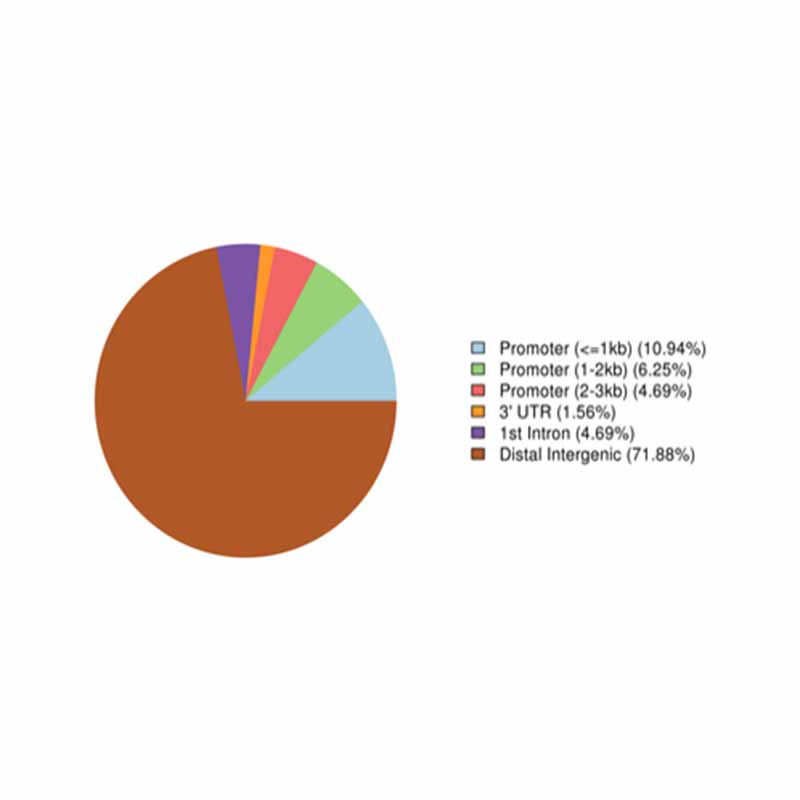
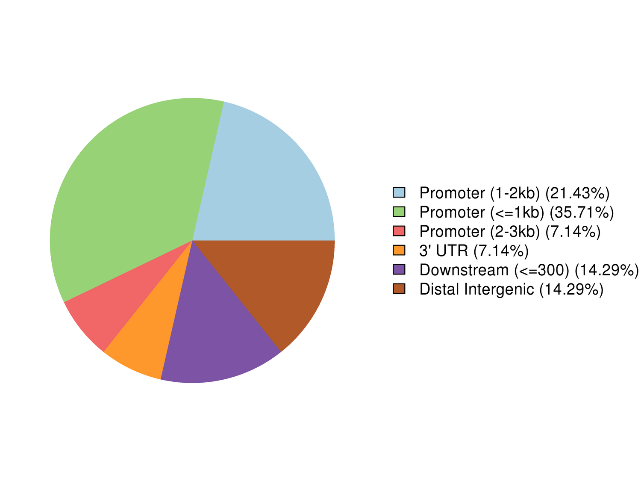
Adnotacja regionów silnie metylowanych
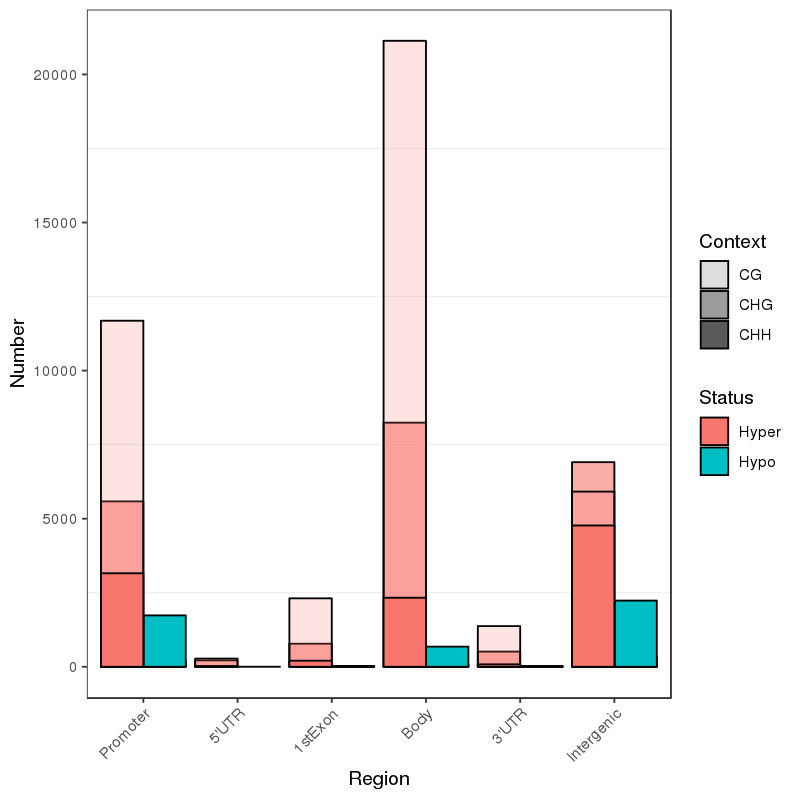
Regiony różnicowo metylowane: powiązane geny
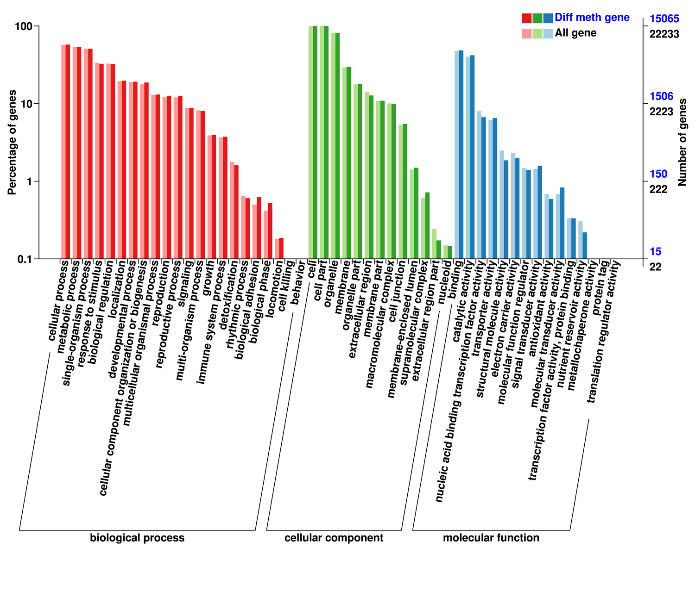
Regiony różnicowo metylowane: adnotacja powiązanych genów (ontologia genów)
Zapoznaj się z postępami badawczymi, jakie umożliwiły usługi BMKGene w zakresie sekwencjonowania wodorosiarczynem całego genomu, poprzez wyselekcjonowany zbiór publikacji.
Fan, Y. i in. (2020) „Analiza profili metylacji DNA podczas rozwoju mięśni szkieletowych owiec przy użyciu sekwencjonowania wodorosiarczynem całego genomu”,BMC Genomika, 21(1), s. 1–15. doi: 10.1186/S12864-020-6751-5.
Zhao, X. i in. (2022) „Nowe zaburzenia metylacji kwasu dezoksyrybonukleinowego u pracowników narażonych na działanie chlorku winylu”,Toksykologia i zdrowie przemysłowe, 38(7), s. 377–388. doi: 10.1177/07482337221098600
Zuo, J. i in. (2020) „Związki między metylacją genomu, poziomami niekodujących RNA, mRNA i metabolitów w dojrzewających owocach pomidora”,Dziennik roślin, 103(3), s. 980–994. doi: 10.1111/TPJ.14778.