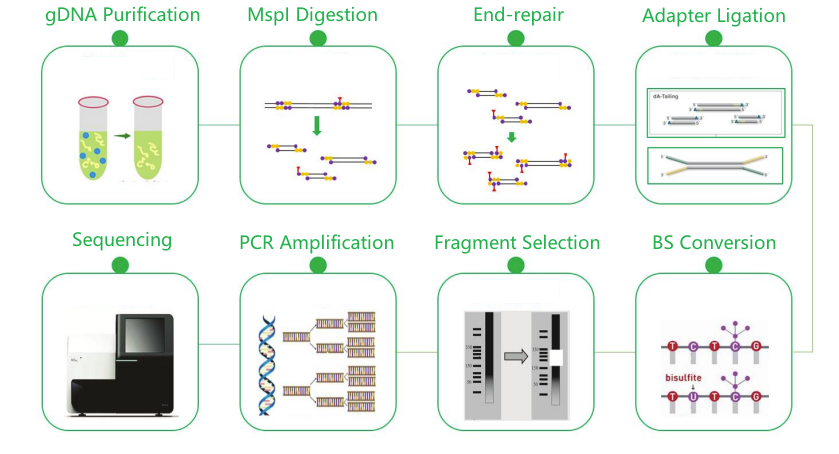
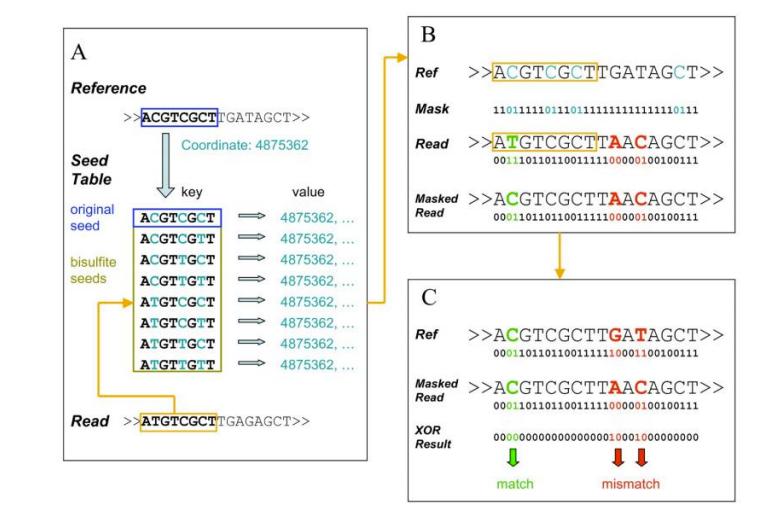
Zmniejszona reprezentacja sekwencjonowanie bisulfitów (RRB)
Funkcje serwisowe
● Wymaga genomu referencyjnego.
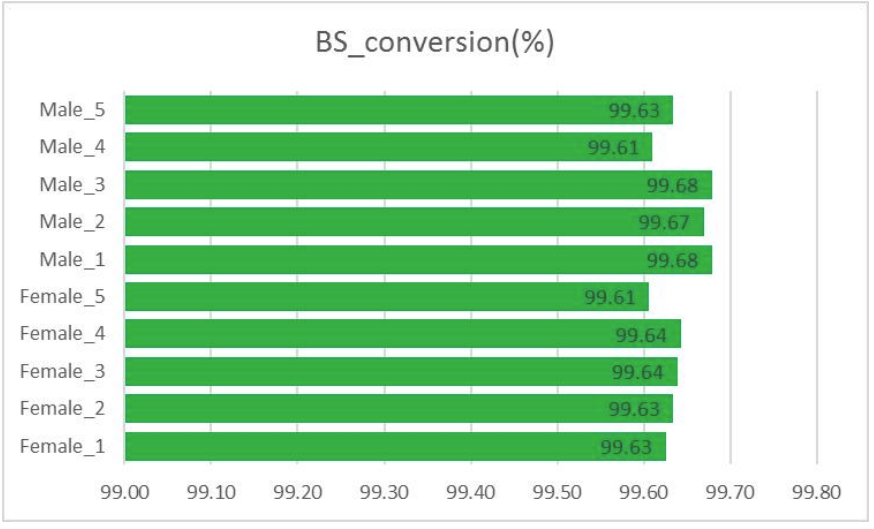
● DNA Lambda służy do monitorowania wydajności konwersji bisulfitu.
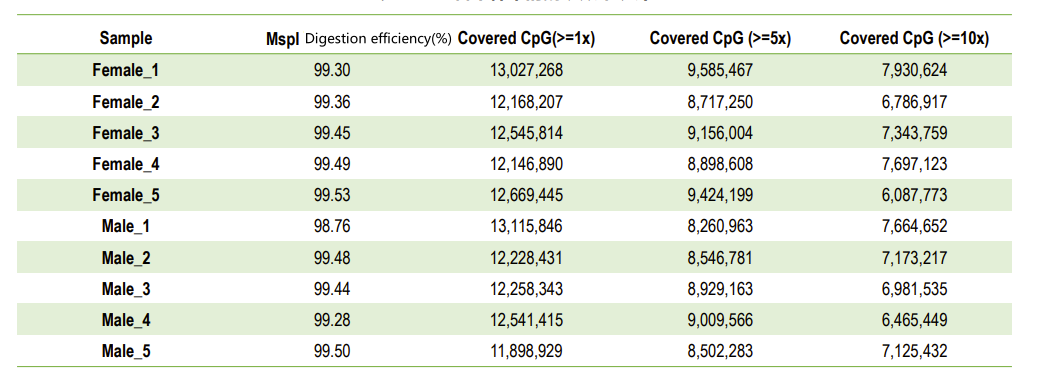
● MSPI Skuteczność trawienia MSPI jest również monitorowana.
● Podwójne trawienie enzymu dla próbek roślin.
● Sekwencjonowanie na Illumina Novaseq.
Zalety serwisowe
●Opłacalna i wydajna alternatywa dla WGBS: Umożliwiając analizę przeprowadzoną po niższych kosztach i z niższymi wymaganiami próbki.
●Kompletna platforma:Zapewnij kompleksową doskonałą obsługę przetwarzania próbek, budowy biblioteki i sekwencjonowania po analizę bioinformatyki.
●Rozległa wiedza specjalistyczna: Dzięki projektom sekwencjonowania RRBS z powodzeniem ukończonym w różnych gatunkach, Bmkgene przynosi ponad dekadę doświadczenia, wysoce wykwalifikowany zespół analizy, kompleksową treść i doskonałe wsparcie po sprzedaży.
Specyfikacje usług
| Biblioteka | Strategia sekwencjonowania | Zalecane dane wyjściowe danych | Kontrola jakości |
| MSPI Trawiono i biblioteka traktowana Bisulfit | Illumina PE150 | 8 GB | Q30 ≥ 85% Konwersja bisulfitu> 99% Wydajność cięcia MSPI> 95% |
Wymagania przykładowe
| Stężenie (ng/µl) | Całkowita ilość (µg) |
| |
| Genomowy DNA | ≥ 30 | ≥ 1 | Ograniczona degradacja lub zanieczyszczenie |
Przepływ pracy usług

Dostawa próbki

Konstrukcja biblioteki

Sekwencjonowanie

Analiza danych
Usługi po sprzedaży
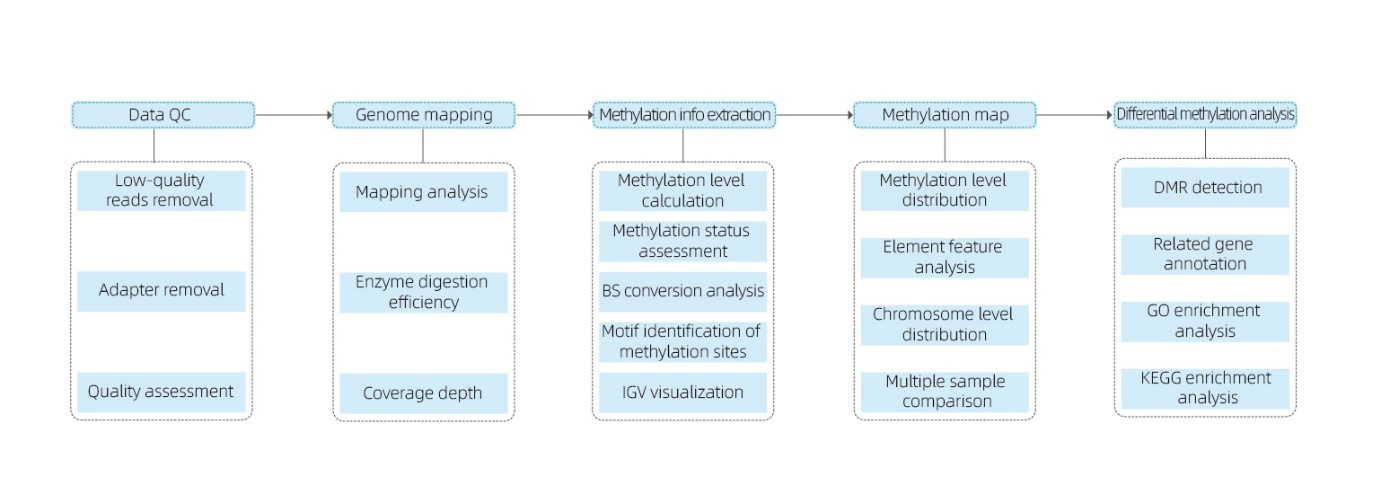
Obejmuje następującą analizę:
● Surowe kontrola jakości sekwencjonowania;
● Mapowanie na genom referencyjny;
● Wykrywanie 5MC metylowanych zasad i identyfikacji motywów;
● Analiza rozkładu metylacji i porównania próbek;
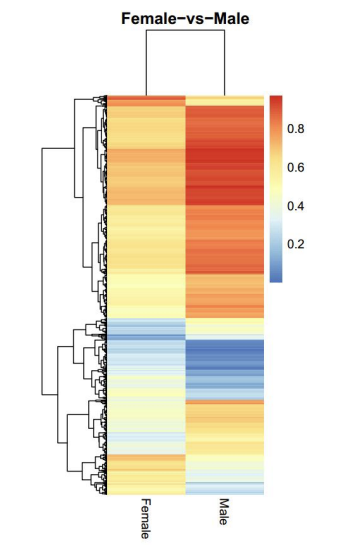
● Analiza regionów metylowanych różnie metylowanych (DMR);
● Funkcjonalna adnotacja genów związanych z DMR.
Kontrola jakości: wydajność trawienia (w mapowaniu genomu)
Kontrola jakości: Konwersja bisulfitów (w ekstrakcji informacji o metylacji)
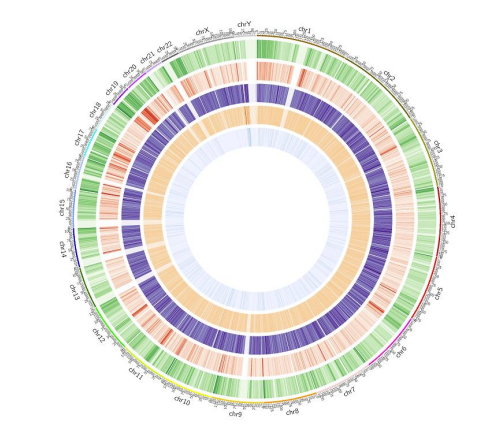
Mapa metylacji: rozkład genomu w całym genomie 5MC
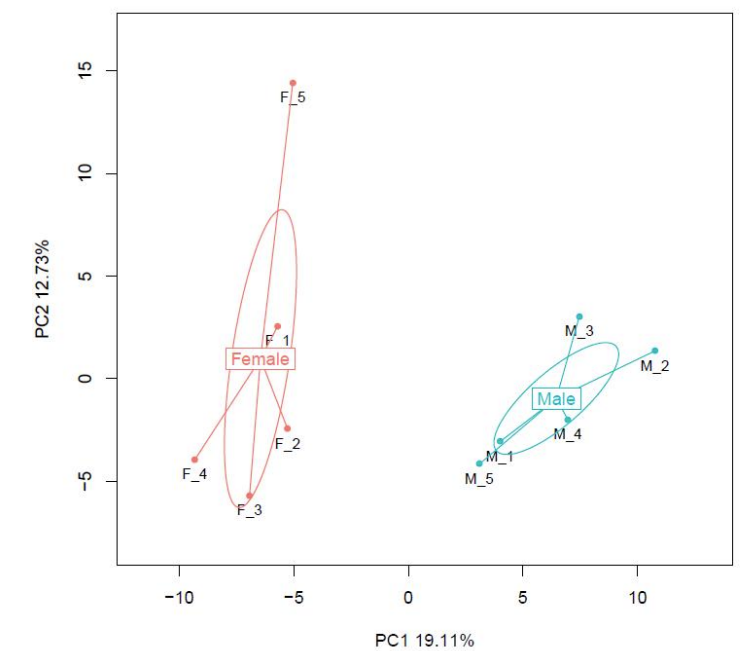
Porównanie próbki: analiza głównego składnika
Analiza różnicowo metylowanych regionów (DMRS): mapa cieplna
Przeglądaj postępy badawcze ułatwione przez usługi sekwencjonowania całego genomu BMKGENE w zakresie sekwencjonowania bisulfitów genomowych poprzez wyselekcjonowany zbiór publikacji.
Li, Z. i in. (2022) „przeprogramowanie o wysokiej wierności do komórek podobnych do Leydiga przez aktywację CRISPR i czynniki parakrynowe”,PNAS Nexus, 1 (4). doi: 10.1093/pnasnexus/PGAC179.
Tian, H. i in. (2023) „Analiza metylacji DNA w całym genomie składu ciała w chińskich bliźniakach monozygotycznych”,Europejski Journal of Clinical Investigation, 53 (11), s. 1 E14055. doi: 10.1111/eci.14055.
Wu, Y. i in. (2022) „Metylacja DNA i stosunek talii do bioder: badanie asocjacyjne całego epigenomu u chińskich monozygotycznych bliźniaków”,Journal of Endocrinological Investigation, 45 (12), s. 2365–2376. doi: 10.1007/s40618-022-01878-4.