Sekwencjonowanie prokariotycznego RNA
Cechy
● Przetwarzanie próbki RNA obejmowało wyczerpanie rRNA, a następnie kierunkowe przygotowanie biblioteki RNA.
● Analiza bioinformatyczna oparta na dopasowaniu do genomu referencyjnego
● Analiza obejmuje ekspresję genów i DEG, ale także analizę struktury transkryptu i sRNA
Zalety serwisu
●Rygorystyczna kontrola jakości: wdrażamy podstawowe punkty kontroli na wszystkich etapach, od przygotowania próbek i bibliotek po sekwencjonowanie i bioinformatykę. To skrupulatne monitorowanie zapewnia niezmiennie wysoką jakość wyników.
●Dane sekwencjonowania specyficzne dla nici: ze względu na kierunkowy charakter przygotowania biblioteki RNA, umożliwiający identyfikację transkryptów antysensownych.
●Kompletna analiza dostosowana do transkryptomów prokariotycznych: rurociąg bioinformatyczny obejmuje nie tylko analizę ekspresji genów, ale także analizę struktury transkryptu, w tym identyfikację operonów, UTR i promotorów. Obejmuje również analizę sRNA, a mianowicie adnotację i przewidywanie struktury drugorzędowej i celów.
●Wsparcie posprzedażowe: nasze zaangażowanie wykracza poza ukończenie projektu z 3-miesięcznym okresem obsługi posprzedażnej. W tym czasie oferujemy monitorowanie projektu, pomoc w rozwiązywaniu problemów oraz sesje pytań i odpowiedzi, aby odpowiedzieć na wszelkie pytania związane z wynikami.
Przykładowe wymagania i dostawa
| Biblioteka | Strategia sekwencjonowania | Zalecane dane | Kontrola jakości |
| Biblioteka kierunkowa zubożona w rRNA | Illumina PE150 | 1-2 Gb | Q30≥85% |
Przykładowe wymagania:
| Stężenie (ng/μl) | Ilość (μg) | Czystość | Uczciwość |
| ≥ 50 | ≥ 1 | OD260/280=1,8-2,0 OD260/230=1,0-2,5 Na żelu widoczne jest ograniczone lub żadne zanieczyszczenie białkiem lub DNA. | RIN≥6,5 |
Zalecana dostawa próbek
Pojemnik: probówka wirówkowa o pojemności 2 ml (nie zaleca się stosowania folii aluminiowej)
Przykładowe oznakowanie: Grupa+replika, np. A1, A2, A3; B1, B2, B3.
Wysyłka:
1. Suchy lód: Próbki należy zapakować do worków i zakopać w suchym lodzie.
2. Probówki RNAstable: Próbki RNA można suszyć w probówkach do stabilizacji RNA (np. RNAstable®) i przesyłać w temperaturze pokojowej.
Przebieg prac serwisowych

Dostawa próbek

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Usługi posprzedażowe
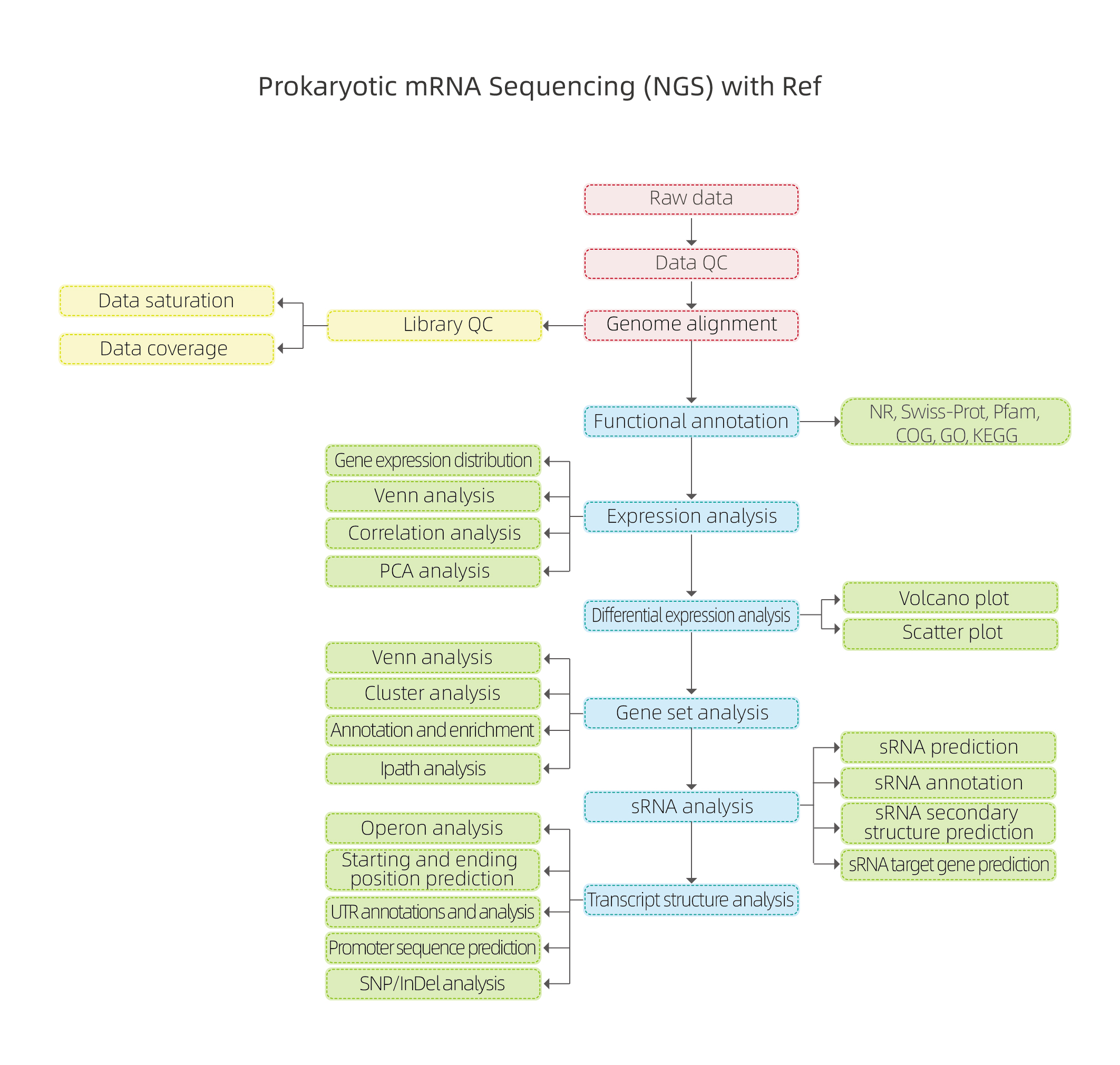
Przebieg analizy bioinformatycznej
Zawiera następującą analizę:
● Kontrola jakości surowych danych
● Dopasowanie do genomu referencyjnego
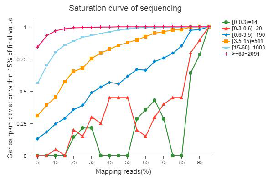
● Ocena jakości biblioteki: losowość fragmentacji RNA, wielkość wstawki i nasycenie sekwencjonowania
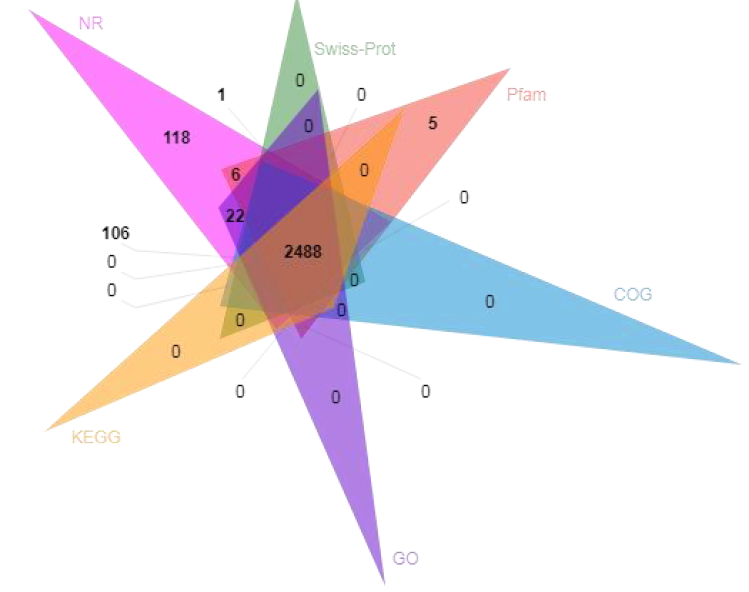
● Adnotacja funkcjonalna przewidywanych genów kodujących
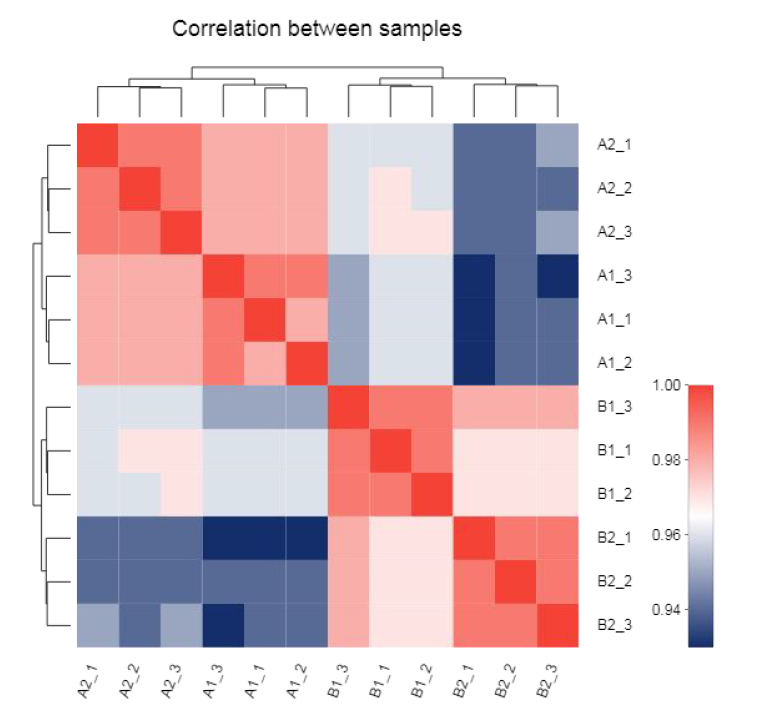
● Analiza wyrażeń: korelacja i analiza głównych składowych (PCA)
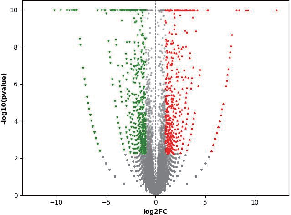
● Różnicowa ekspresja genów (DEG)
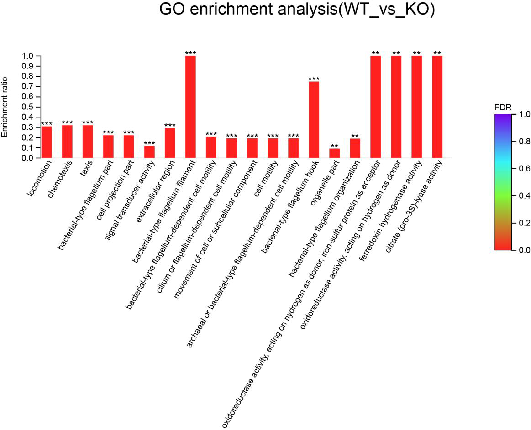
● Adnotacje funkcjonalne i wzbogacanie DEG
● Analiza sRNA: przewidywanie, adnotacja, przewidywanie struktury docelowej i drugorzędowej
● Analiza struktury transkryptu: operony, pozycje początkowe i końcowe, region nieulegający translacji (UTS), promotor i analiza SNP/InDel
Sekwencjonowanie nasycenia
Adnotacja funkcjonalna genów kodujących
Korelacja między próbkami
Analiza genów ulegających ekspresji różnicowej (DEG).
Analiza wzbogacenia funkcjonalnego
Adnotacja sRNA
W tej wyróżnionej publikacji zapoznaj się z postępami, jakie umożliwiły usługi pełnometrażowego sekwencjonowania mRNA Nanopore firmy BMKGene.
Guan, CP i in. (2018) „Globalne zmiany transkryptomu Staphylococcus epidermidis tworzący biofilm w odpowiedzi na całkowite alkaloidy Sophorea alopecuroides”,Polskie czasopismo Mikrobiologii, 67 ust. 2, s. 1. 223. doi: 10.21307/PJM-2018-024.